WIPO Sequence Benutzerhandbuch
Zweck dieses Dokuments ist es, Benutzern eine Anleitung zur Durchführung grundlegender Vorgänge mit der Desktop-Anwendung WIPO Sequence zur Verfügung zu stellen. Normalerweise handelt es sich bei den Benutzern um einen Patentanmelder oder dessen Vertreter, der eine Patentanmeldung samt einem Sequenzprotokoll einreichen möchte.
Dieses Benutzerhandbuch gilt für Version 3.0.0.
1. Zusammenfassung der Funktionen
In dieser Auflistung werden alle vom Tool implementierten Funktionen in der aktuellen Version des Tools, mit Links zu den entsprechenden Abschnitten, beschrieben.
2. Tool-Funktionen
2.1 Startansicht Projekte
In diesem Abschnitt werden die verschiedenen Optionen der Projekte-Startansicht genauer beschrieben.
Ein Projekt ist die von dem Tool verwendete Objektstruktur, um die zur Erstellung eines Sequenzprotokolls erforderlichen Daten zu speichern. Nachdem diese Daten als konform mit dem WIPO-Standard ST.26 validiert wurden, verwendet das Tool die im Projekt gespeicherten Daten als Werte innerhalb des generierten Sequenzprotokolls.
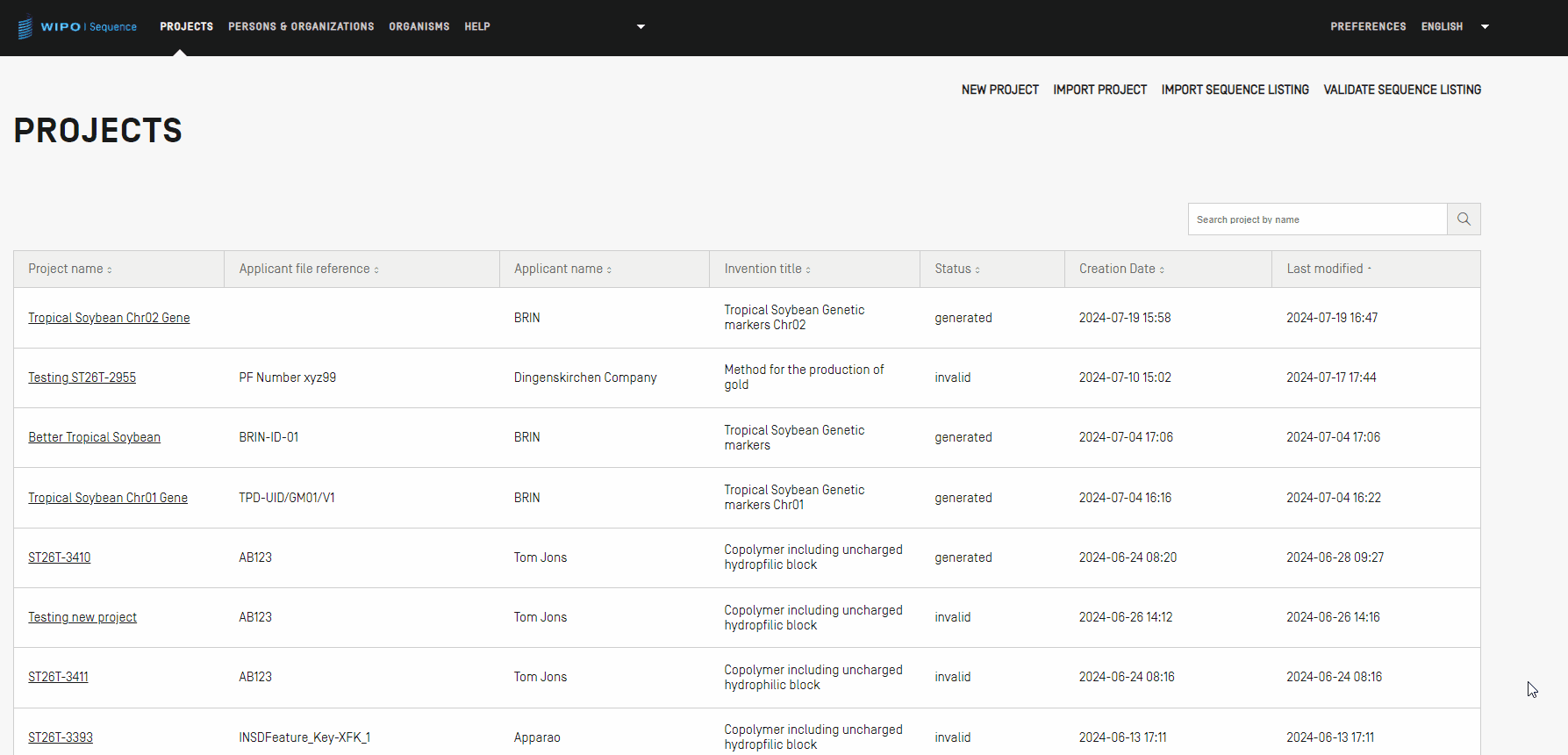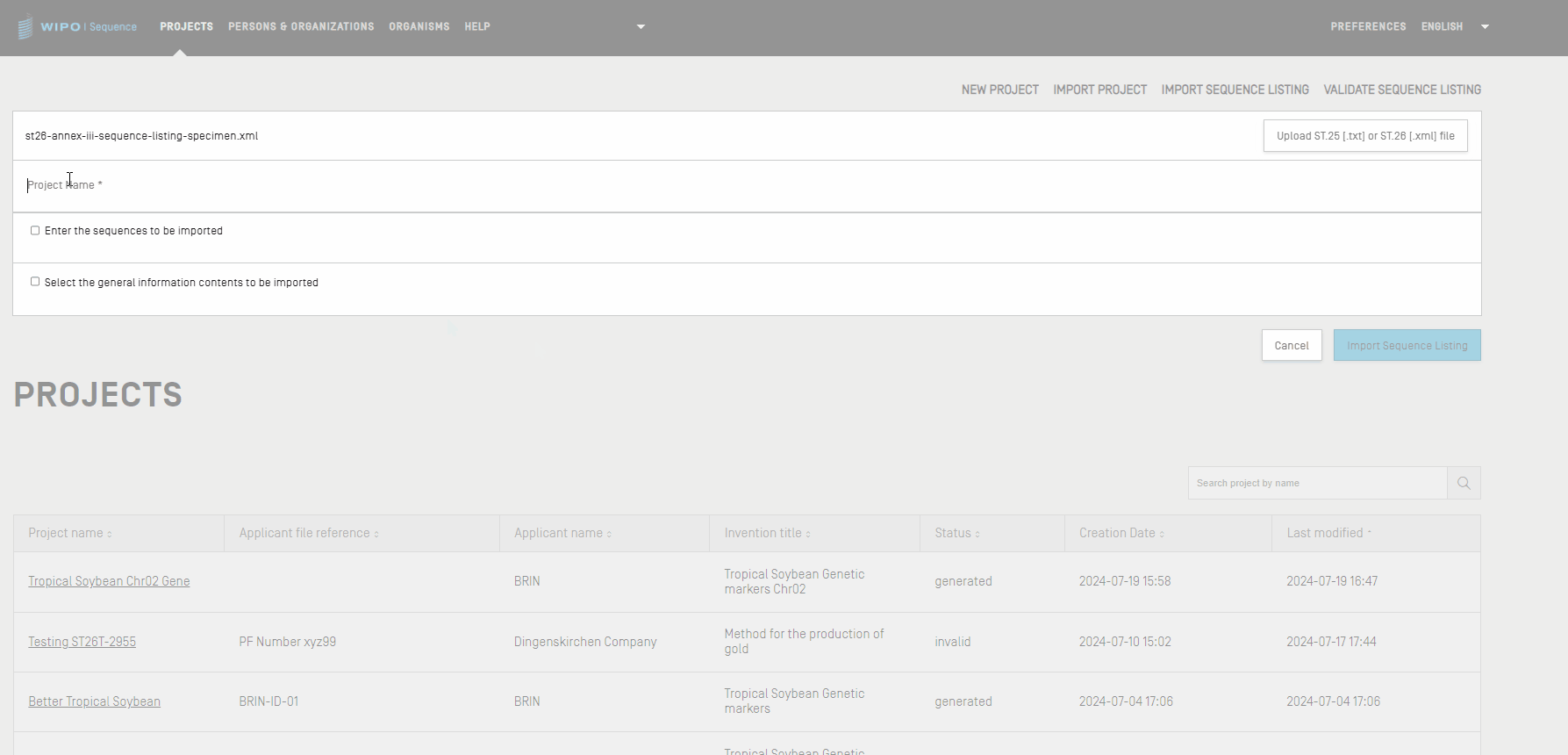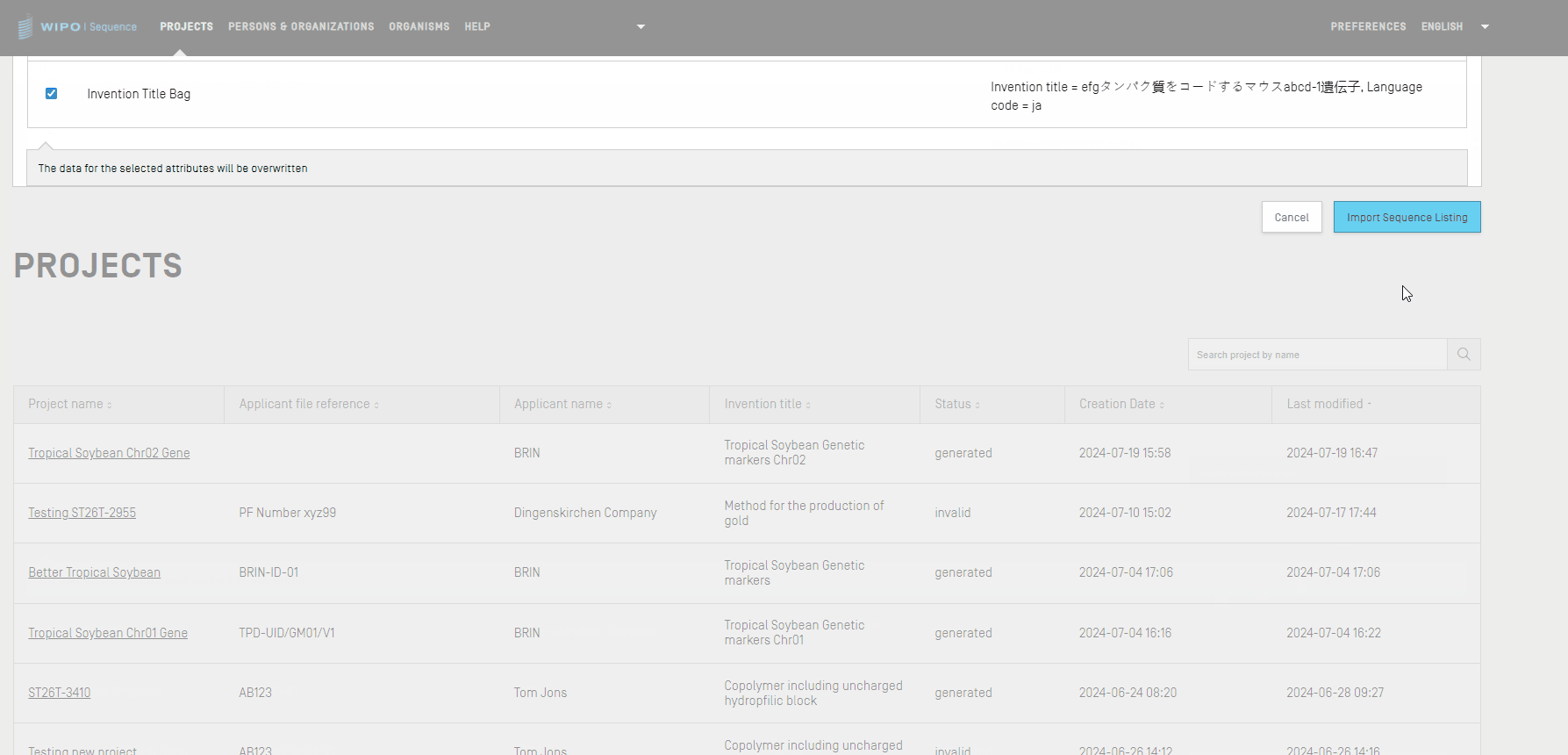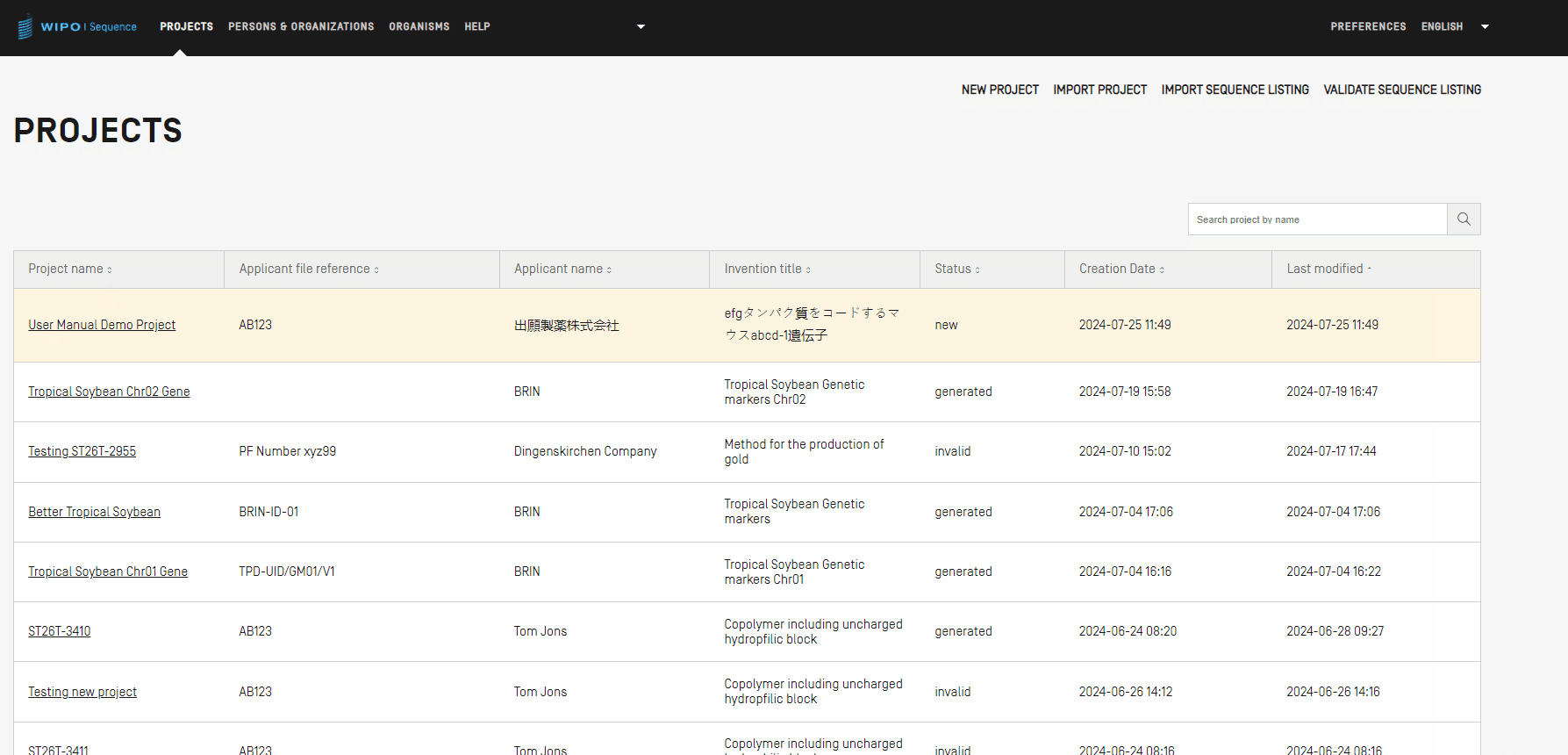
In dieser Ansicht wird die Liste der erstellten Projekte angezeigt, die Sie sortieren oder mithilfe der Suchfunktion nach Projektname, Aktenzeichen des Anmelders, Name des Anmelders, Bezeichnung der Erfindung, Status oder Erstellungsdatum filtern können.
Hinweis: Das Tool zeigt maximal 1000 Projekte an. Zeigt das Tool ein Projekt in der Projekte-Startansicht nicht an, verwenden Sie die Suchfunktion, um das Projekt anhand seines Namens zu identifizieren, denn es ist weiterhin lokal gespeichert und nur in dieser Ansicht nicht sichtbar.
Projekt erstellen
Gehen Sie zur Startansicht Projekte, um ein neues Projekt zu erstellen.

1) Klicken Sie oben in der Ansicht auf den Link "NEW PROJECT" (NEUES PROJEKT). Wie angezeigt verlangt das Tool einen Namen (obligatorisch) und eine Beschreibung (optional).

2) Nachdem Sie einen Wert in das Namensfeld eingegeben haben, wird die Schaltfläche "Save" freigeschaltet und Sie können das neue Projekt speichern. Die Projektliste enthält nun dieses neue Projekt in der Startansicht Projekte.
Projekt importieren
Verwenden Sie diese Funktion, um ein zuvor exportiertes Projekt in das Tool zu importieren. Um eine Projektdatei zu importieren, gehen Sie zur Startansicht Projekte.
Klicken Sie oben in der Ansicht auf den Link "IMPORT PROJECT" (PROJEKT IMPORTIEREN) und befolgen Sie dann die Schritte im folgenden Video:
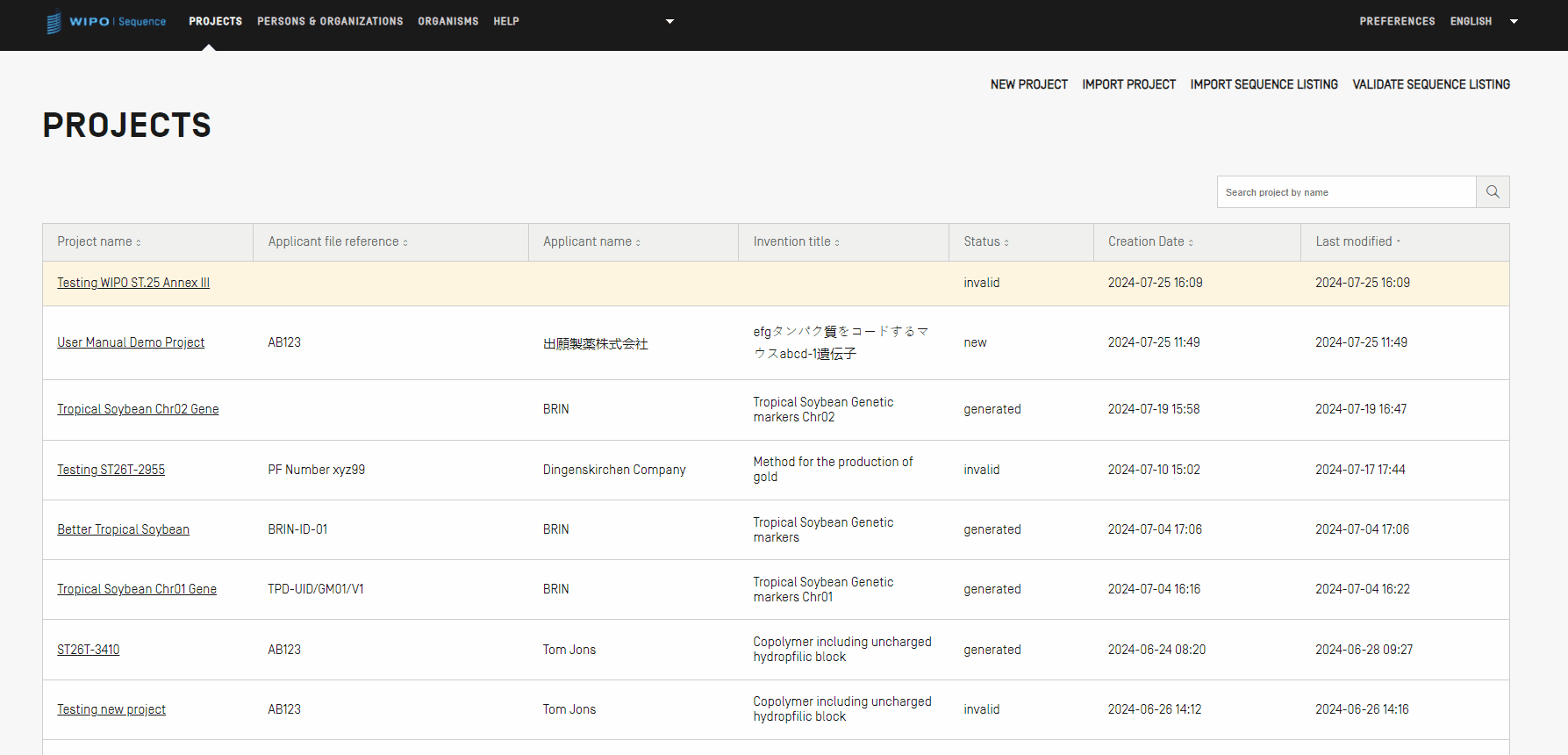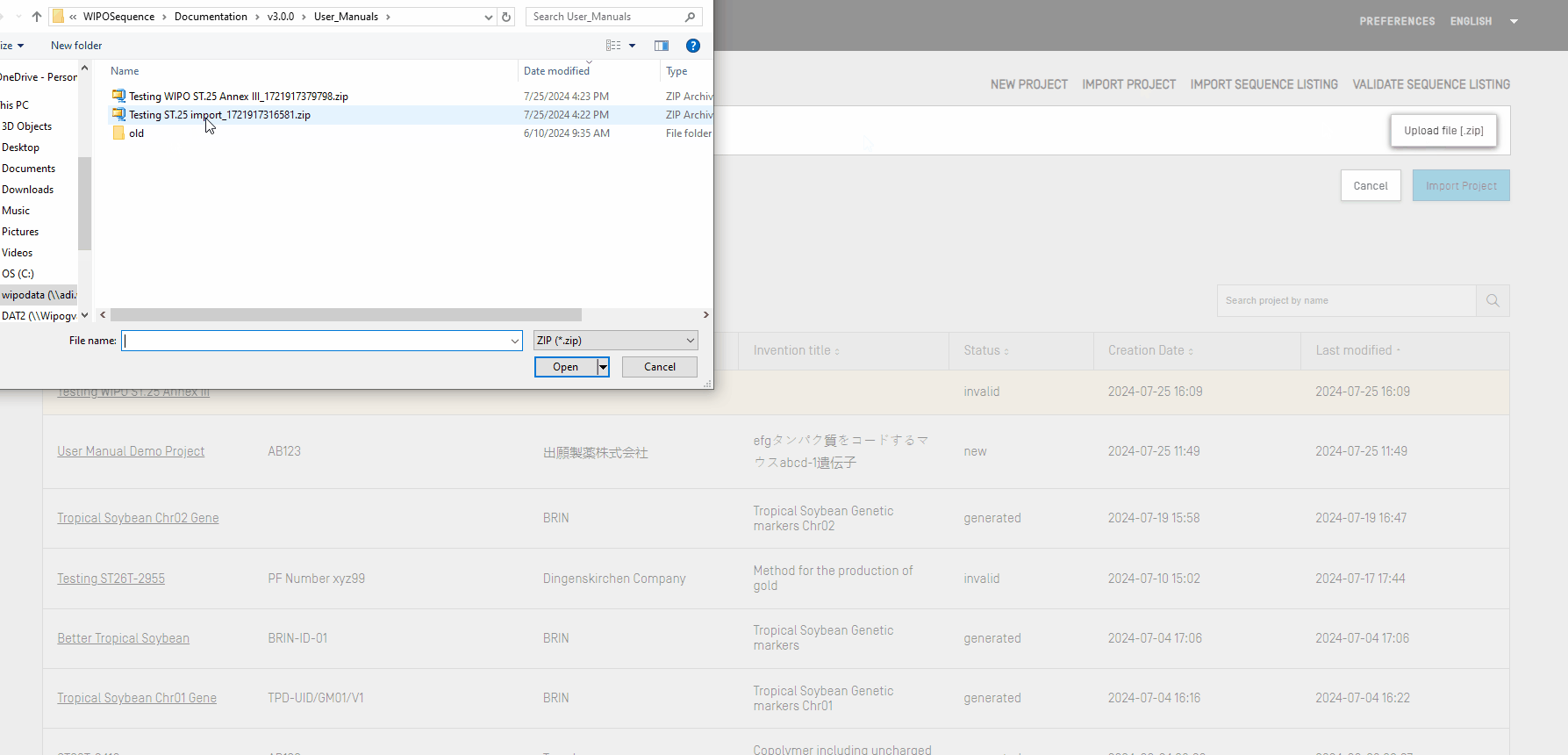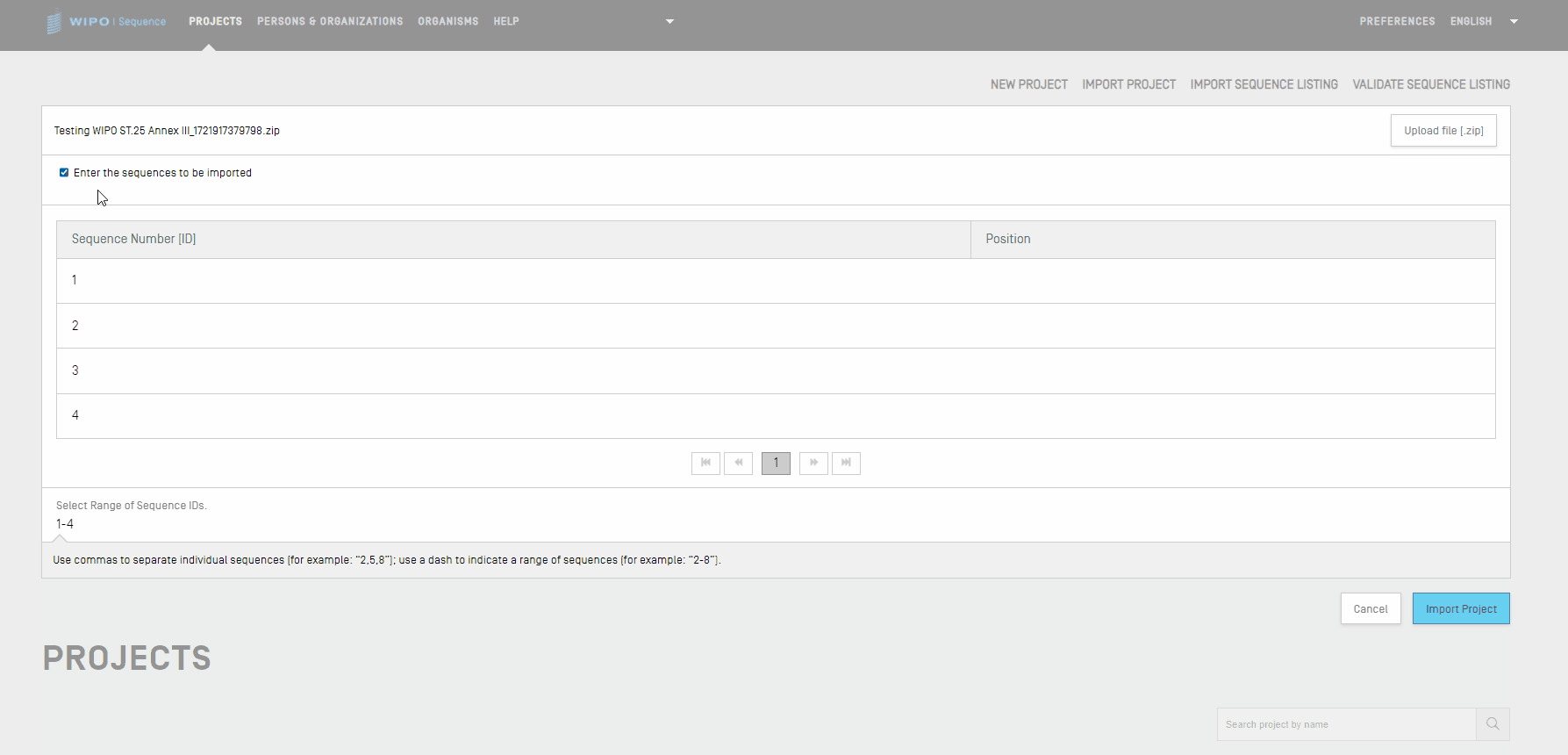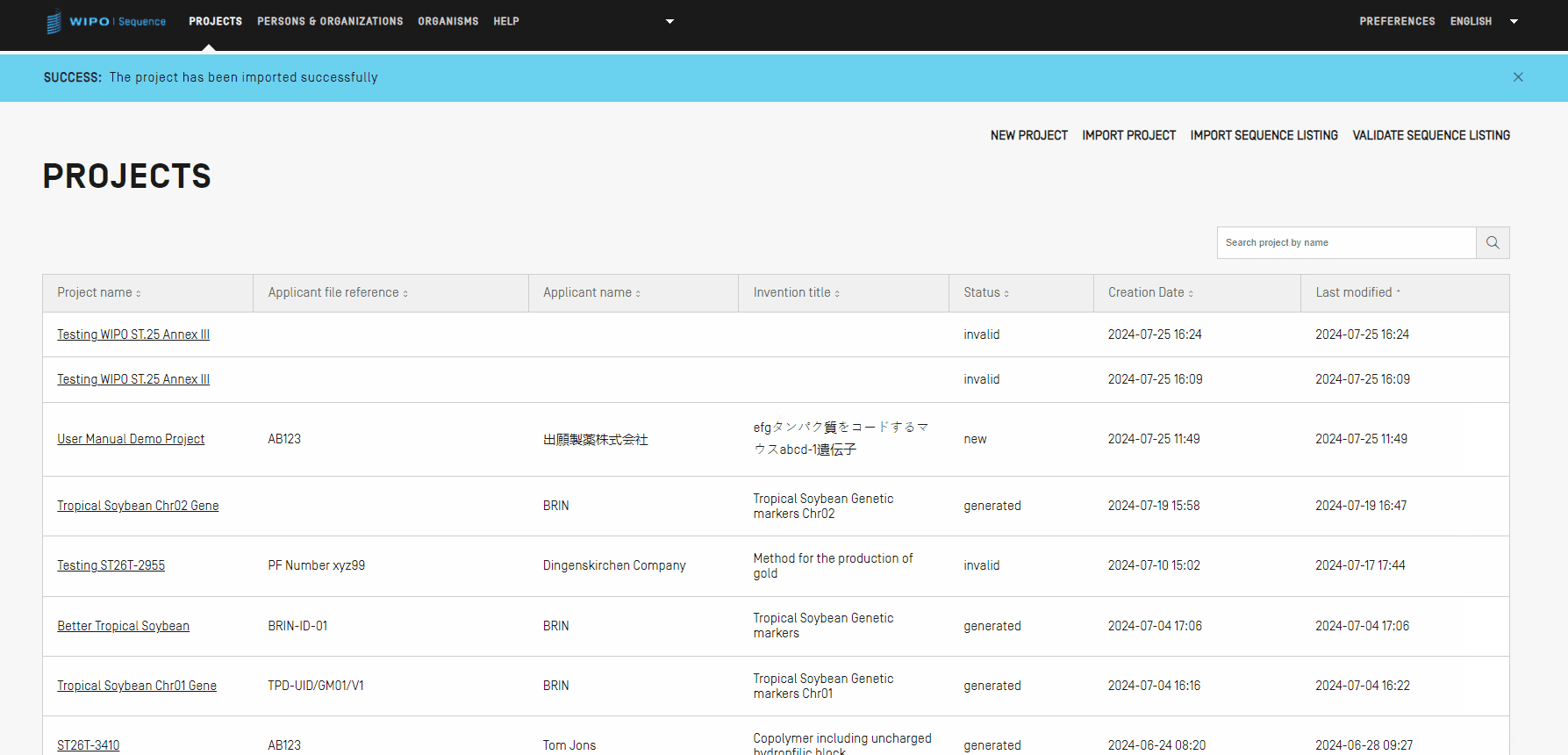
Ist "Select Range Sequences" (Sequenzbereich auswählen) deaktiviert, werden alle Sequenzen importiert. Wenn Sie auswählen möchten, welche Sequenzen in das Projekt importiert werden sollen, aktivieren Sie das Kontrollkästchen "Select Range Sequences" (Sequenzbereich auswählen) und geben Sie die ID-Nummern der gewünschten Sequenzen in das entsprechende Feld ein. Sie können sowohl eine einzelne Sequenz eingeben als auch eine Liste von durch Kommas getrennten Sequenzen oder einen Sequenzbereich in der Schreibweise x-y. Standardmäßig wird die Gesamtzahl der Sequenzen des importierten Projekts als Bereich angezeigt, d.h.: 1– Gesamtzahl von Sequenzen.
Beispiel: “1, 3, 7, 13-20, 30-50”
Wurde das Projekt erfolgreich importiert, wird das folgende blaue Banner mit einer Meldung oben in der Ansicht angezeigt.
Hinweis: Stellen Sie sicher, dass das importierte Zip-Projekt mit der aktuellen Datenbank übereinstimmt. Wenn das Projekt in einer Version vor 3.0.0 exportiert wurde, kann es nicht richtig funktionieren; das importierte Projekt muss dieselbe Datenbank verwenden. Dies liegt an der neuen Datenbankimplementierung ab Version 3.0.0 .
Sequenzprotokoll importieren
In der Startansicht Projekte können Sie ausschließlich die Sequenzinformationen aus einem ST.26- oder ST.25-konformen Sequenzprotokoll importieren. Die Dateiformate hierfür sind jeweils *.xml für das ST.26-Format und *.txt für ST.25-Dateien. Weitere Informationen zu den einzelnen Schritten finden Sie im folgenden Video.
Hinweis: Beim Importieren eines Sequenzprotokolls wird bei den Merkmalen und Qualifiern zwischen Groß- und Kleinschreibung unterschieden. Achten Sie deshalb darauf, dass sie den in Anhang I von WIPO ST.26 angegebenen Werten entsprechen.
Beachten Sie außerdem, dass importierte ST.25-konforme Sequenzprotokolle gültig sein müssen, da sonst die Funktionalität von WIPO Sequence während des Imports nicht garantiert werden kann.
Die Importbericht-Tabelle wird nur angezeigt, wenn beim Import einer Datei Fehler auftreten. Die Tabelle enthält die folgenden Spalten:
- Art des Hinweises: "INDIVIDUAL" für eine Meldung zu einer bestimmten Sequenz oder "GLOBAL" für eine oder mehrere Sequenzen allgemein
- Datenelement: Aus der Quell-Datei für ST.25-Sequenzprotokolle
- Meldungstext: Ausführliche Meldung mit Angaben zu dem festgestellten Problem und den zur Behebung vorgenommenen Änderungen (falls zutreffend)
- Ermittelte Sequenz: Sequenznummer der importierten Sequenz in Bezug auf die Meldung (nur wenn der Hinweis "INDIVIDUAL" ist, andernfalls bleibt dieses Feld leer).

Im Falle des Dateiformats ST.25 enthält die "Import Report" Ansicht (Importbericht) zunächst einen Importbericht sowie einen Bericht der geänderten Daten ("Changed Data"). In dem Bericht der geänderter Daten ("Changed Data") werden alle Daten angezeigt, die während des Importvorgangs eine Transformation oder Änderung erfahren haben. Die folgenden Daten werden in einer Übersichtstabelle dargestellt:
- Origin Tag (Ursprungstag): Das Datenelementcode für die Elementart, für ST.25-Sequenzprotokolle
- Ursprungselementname: Der entsprechende Name für die Elementart
- Ursprungselementwert: Der entsprechende Wert des Originalelements in der Quell-Datei
- (Zielelementname: Ein äquivalenter ST.26-Elementname, in dem die Informationen im Projekt gespeichert werden
- Zielelementwert: Der Wert, der für den Zielelementnamen im Projekt eingestellt wird
- Transformation: Eine Beschreibung der an dem Element vorgenommenen Änderungen/Transformation
- Sequenz-ID-Nummer: ID-Nummer der relevanten Sequenz des transformierten Elements im Projekt

An dieser Stelle können Sie zur Ansicht Projekt-Start zurückkehren oder einen Bericht über diese Änderungen im PDF-Format drucken. Anweisungen zum Herunterladen/Drucken der PDF-Datei finden Sie im Abschnitt Display sequence listing (Sequenzprotokoll anzeigen).
Umgekehrt kann der Importvorgang fehlschlagen, wenn die Sequenzprotokolldatei Fehler enthält. In diesem Fall werden Sie nach dem Importversuch mit einem roten Banner darauf hingewiesen, dass beim Import ein Fehler aufgetreten ist.

Hinwies: Das Tool arbeitet am besten am Schwellenwert von 100k Sequenzen. Bei der Bearbeitung umfangreicher Sequenzprotokolle kann der Benutzer das folgende Workaround durchführen: Teilen Sie den Importvorgang in eine Reihe von Schritten auf, indem Sie einen bestimmten Bereich der zu importierenden Sequenzen auswählen und diese Sequenzen dann innerhalb eines Projekts Bereich nach Bereich importieren. Zum Beispiel kann ein Sequenzprotokoll von ~100k Sequenzen in eine Reihe von 10 x 10k Sequenzen aufgeteilt werden, und diese können einzeln importiert werden. Die ersten 10k würden bei der Erstellung des Projekts verwendet.
Sequenzprotokoll validieren
Sie können eine ST.26-Sequenzprotokolldatei validieren, indem Sie auf die Schaltfläche "VALIDATE SEQUENCE LISTING" (SEQUENZPROTOKOLL VALIDIEREN) oben rechts in der Ansicht Projekte klicken

Wird das Sequenzprotokoll validiert, erscheint ein Banner wie dargestellt.

Wenn die Validierung des Sequenzprotokolls fehlschlägt, wird in Ihrem Browser ein Prüfbericht geöffnet, in dem die Validierungsfehler wie im Beispiel aufgelistet sind. Der Speicherort der HTML-Datei wird zusammen mit dem XML-Prüfbericht angezeigt, falls Sie die Dateien an einen anderen Ort kopieren möchten.
Wenn Sie einen Internet Explorer-Browser verwenden, müssen Sie die Ausführung eines internen Skripts auf Ihrem Computer zulassen, damit das Format korrekt geladen werden kann. Andernfalls werden die Sequenzen nicht im Standardformat angezeigt und sind weniger lesbar.
Bitte beachten Sie, dass für die Validierung eines Sequenzprotokolls die ST.26-Datei folgende Voraussetzungen erfüllen sollte:
-
Das Sequenzprotokoll muss in UTF-8 kodiert sein und gültige Zeichen gemäß der XML 1.0-Spezifikation enthalten.
-
Sie muss eine wie folgt lautende DOCTYPE-Zeile enthalten:
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing 1.3//EN" "ST26SequenceListing_V1_3.dtd">
Die Datei muss der DTD-Datei ST26SequenceListing_V1_3.dtd entsprechen.
Projekt löschen
Gehen Sie zur Startansicht Projekte, um ein Projekt zu löschen.
Klicken Sie auf die Schaltfläche mit dem Papierkorbsymbol in der Zeile der Tabelle der Ansicht Projekte, um das gewünschte Projekt zu löschen.
Klicken Sie in der sich öffnenden Pop-up-Box auf "Delete" (Löschen), um zu bestätigen, dass Sie das ausgewählte Projekt löschen möchten.

2.2 Ansicht Projektdetails
Die Ansicht Projektdetails enthält alle spezifischen Informationen zu einer bestimmten Patentanmeldung oder einem bestimmten Sequenzprotokoll. Sie ist in zwei Abschnitte unterteilt: Allgemeine Angaben und Sequenzen. In der Ansicht oben finden Sie eine Tabelle mit den allgemeinen Angaben zum Projekt, einschließlich:
- Name des Projekts
- Datum und Uhrzeit der Erstellung des Projekts
- Datum und die Uhrzeit der letzten Aktualisierung des Projekts
- Projektstatus (mögliche Werte: "new"/"modified"/"generated"/"invalid"/"valid"/"warnings" für: neu/geändert/erstellt/ungültig/gültig/Warnungen) - Dieses Feld kann nicht bearbeitet werden!
- Beschreibung des Projekts – optional
- Name der importierten Datei (falls das Projekt importiert wurde)
- Original Freitext-Sprachcode
- Anzahl der Sequenzen (bezeichnet durch "Sequenzen")
- Ein Kontrollkästchen zum automatischen Hinzufügen eines Translations-Qualifiers wenn ein CDS-Merkmal erstellt wird (Funktion auf Projektebene)
- Nicht-englischer Freitext-Sprachcode

Es gibt zwei Menüebenen: Die erste bezieht sich auf die Daten in der Ansicht Projektdetails (gelb dargestellt) und die zweite ist ein Navigationsmenü zu anderen Ansichten, die mit dem Projekt verbunden sind (blau dargestellt). Um das Projekt zu verlassen, klicken Sie auf "Return to project home" (Zurück zum Projekt-Start).
Für das Navigationsmenü gibt es sechs verschiedene Ansichten, die Sie über die Seite Projektdetails-Seite erreichen:
- Ansicht "Project Details" (Projektdetails, aktuelle Ansicht), angegeben durch den Namen des Projekts
- Ansicht "Verification Report" (Prüfbericht), um auf den Prüfbericht zuzugreifen
- Ansicht "Language-Dependent Qualifiers" (Sprachabhängige Qualifier), um auf sprachabhängige Freitext-Qualifier zuzugreifen und diese zu exportieren/importieren
- Ansicht "Import Report" (Importbericht), um auf den Importbericht zuzugreifen
- Ansicht "Display Sequence Listing" (Sequenzprotokoll anzeigen), um auf visuell lesbare Formate des generierten ST.26-Sequenzprotokolls zuzugreifen
- "Help Menu" (Hilfe) mit Verweisen auf das Benutzerhandbuch, WIPO Sequence und die ST.26-Wissensdatenbank
- Ansicht "Preferences" (Einstellungen), die für alle Projekte in dieser Instanz der WIPO-Sequenz relevant ist.
Projekt drucken
Zum Drucken eines Projekts rufen Sie die Ansicht Projektdetails des gewünschten Projekts auf und klicken Sie in der Ansicht oben auf "Print" (Drucken).
Als Nächstes werden Ihnen zwei Kontrollkästchen angezeigt, aus denen Sie wählen können, welche Informationen aus dem Projekt gedruckt werden sollen: Allgemeine Angaben drucken und/oder Sequenzen drucken.
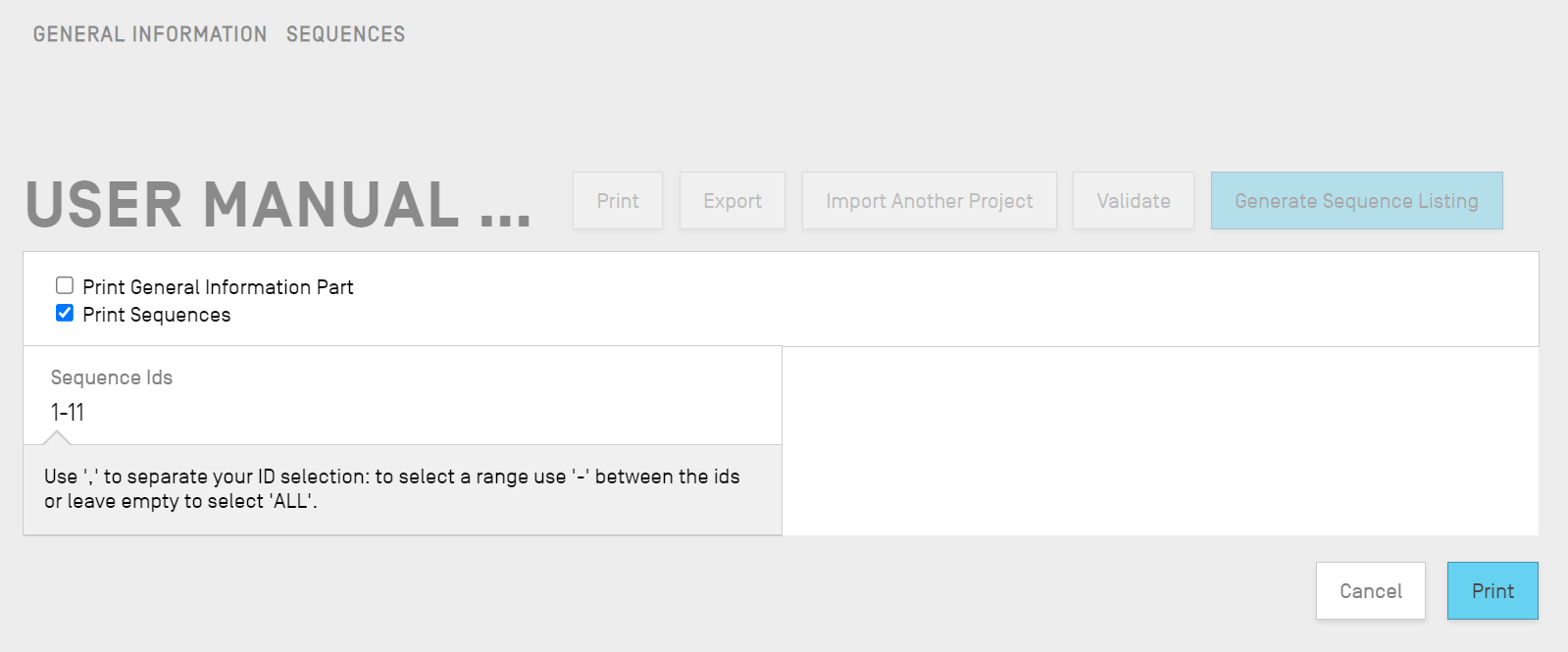
Wenn Sie "Print Sequences" (Sequenzen drucken) auswählen, können Sie einschränken, welche Sequenzen gedruckt werden sollen, indem Sie im Feld "Sequence-IDs" den Bereich der entsprechenden ID-Nummern angeben, oder aber alle Sequenzen drucken, indem Sie dieses Feld leer lassen.
Standardmäßig wird die Gesamtzahl der Sequenzen des Projekts als Bereich angezeigt.
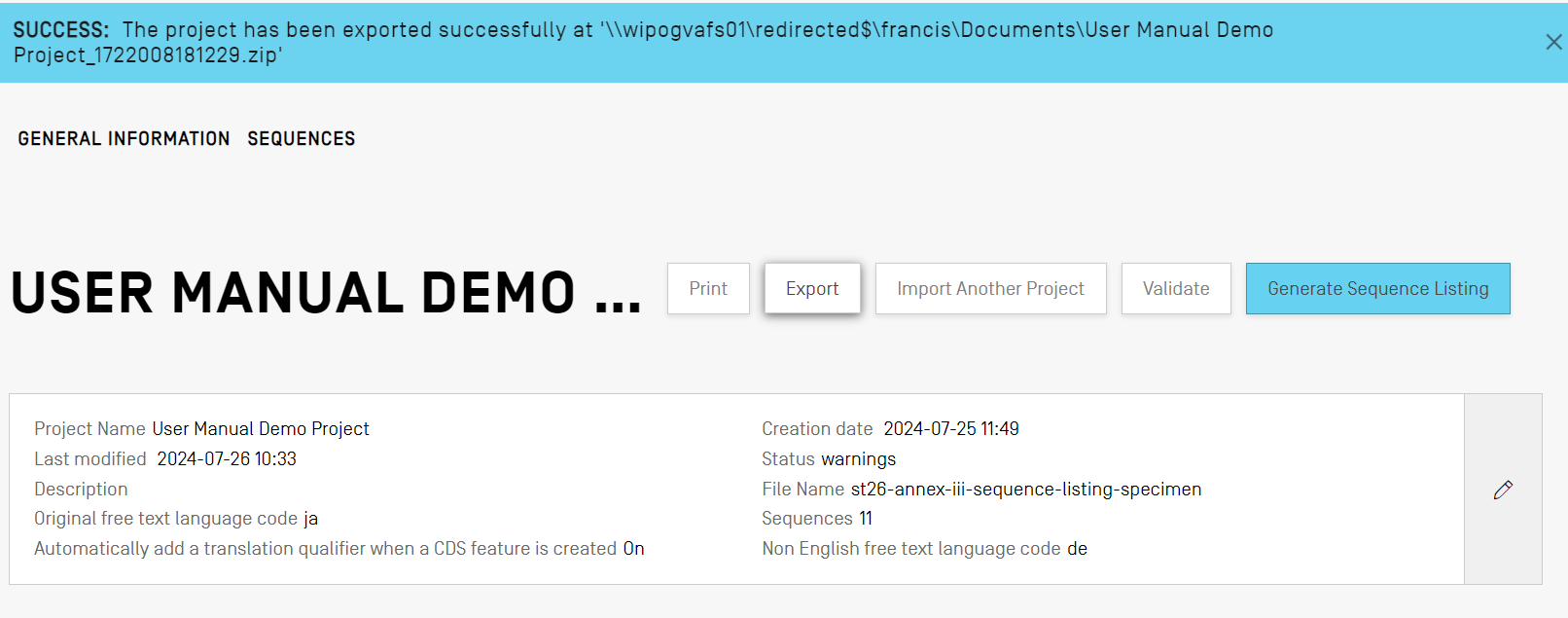
Projekt exportieren
Sie können ein Projekt in eine .zip-Datei exportieren, um Projektdaten zu sichern oder alternativ mit einem anderen Desktop-Computer, auf dem WIPO Sequence installiert ist, zu importieren. Klicken Sie einfach auf die Schaltfläche "Export" (Exportieren) und wählen Sie einen Speicherort für die .zip- Datei aus. War der Export erfolgreich, erscheint das folgende blaue Banner:
Importieren Sie ein anderes Projekt in das aktuelle Projekt
Sie können Informationen aus anderen Projekten, die im Tool gespeichert sind, in das aktuell geöffnete Projekt kopieren. Die importierten Informationen können entweder in den Abschnitt "General Information" (Allgemeine Angaben), in den Abschnitt "Sequences" (Sequenzen) oder in beide der genannten Abschnitte eingefügt sein. Importierte Allgemeine Angaben ersetzen die aktuell vorhandenen Allgemeinen Angaben im Projekt, während importierte Sequenzen an die aktuelle Liste der bereits im Projekt vorhandenen Sequenzen angehängt werden.

Wählen Sie zunächst das Projekt, aus dem Sie Informationen importieren möchten, aus dem angezeigten Dropdown-Menü aus. Als Nächstes bestimmen Sie, ob Sie Teile der Angaben im Abschnitt Allgemeine Angaben des Projekts einbeziehen möchten. Wollen Sie auch Sequenzen importieren, müssen Sie einen Bereich von Sequenz-ID-Nummern angeben, um festzulegen, welche der Sequenzen in das Projekt importiert werden sollen. Standardmäßig wird die Gesamtzahl der Sequenzen des Projekts als Bereich angezeigt.
Ist das Kontrollkästchen bezüglich der Allgemeinen Angaben aktiviert, wird eine Tabelle angezeigt, die alle Elemente des Abschnitts Allgemeine Angaben beider Projekte anzeigt: des aktuell ausgewählten (Ziel-)Projekts und des importierten Projekts (Alternative). Anschließend müssen Sie auswählen, welche Elemente der Allgemeinen Angaben durch die Allgemeinen Angaben des entsprechenden importierten Projekts ersetzt werden sollen.
Nachdem Sie entschieden haben, welche Elemente der Allgemeinen Angaben und Sequenzen in das Projekt importiert werden sollen, müssen Sie auf die blaue Schaltfläche "Import Project" (Projekt importieren) klicken. Das Tool zeigt ein blaues Banner an, wenn die Elemente korrekt importiert wurden.
Projekt validieren
Bevor das Sequenzprotokoll als ST.26-konforme XML-Datei erstellt wird, durchläuft ein Projekt eine Validierungsprüfung. Dieser Schritt wird immer vor der Generierung des Sequenzprotokolls durchgeführt, kann aber auch unabhängig davon erfolgen.
Zur Validierung eines Projekts klicken Sie in der Ansicht "Project Details" (Projektdetails) oben auf die Schaltfläche "Validate" (Validieren).
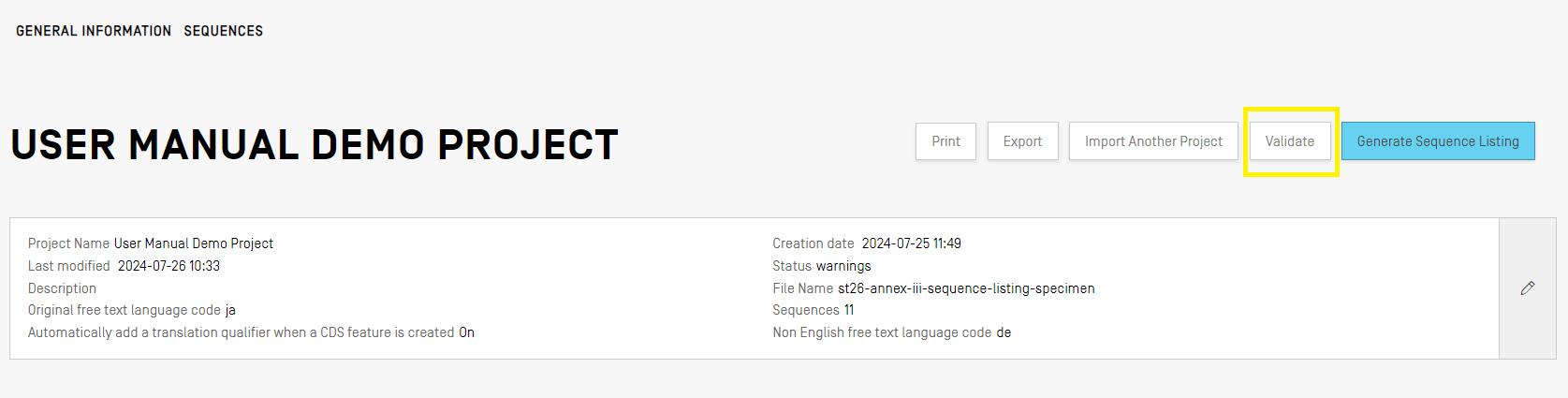
Nach Abschluss der Validierung wird die Ansicht "Verification Report" (Prüfbericht) geöffnet, in der alle eventuell generierten Prüffehler/Warnungen angezeigt werden. Wurde die Validierung erfolgreich abgeschlossen, zeigt das Tool zeigt ein blaues Banner an.
Werden während des Validierungsprozesses Fehler oder Warnungen festgestellt, so wird ein Prüfbericht mit einer Tabelle erstellt, in der die erkannten Prüfregeln und Richtlinien, gegen die verstoßen wurde, aufgeführt sind. In jeder Zeile wird angegeben, ob es sich um einen Fehler handelt, der korrigiert werden muss, oder um eine Warnung, die ignoriert werden kann.
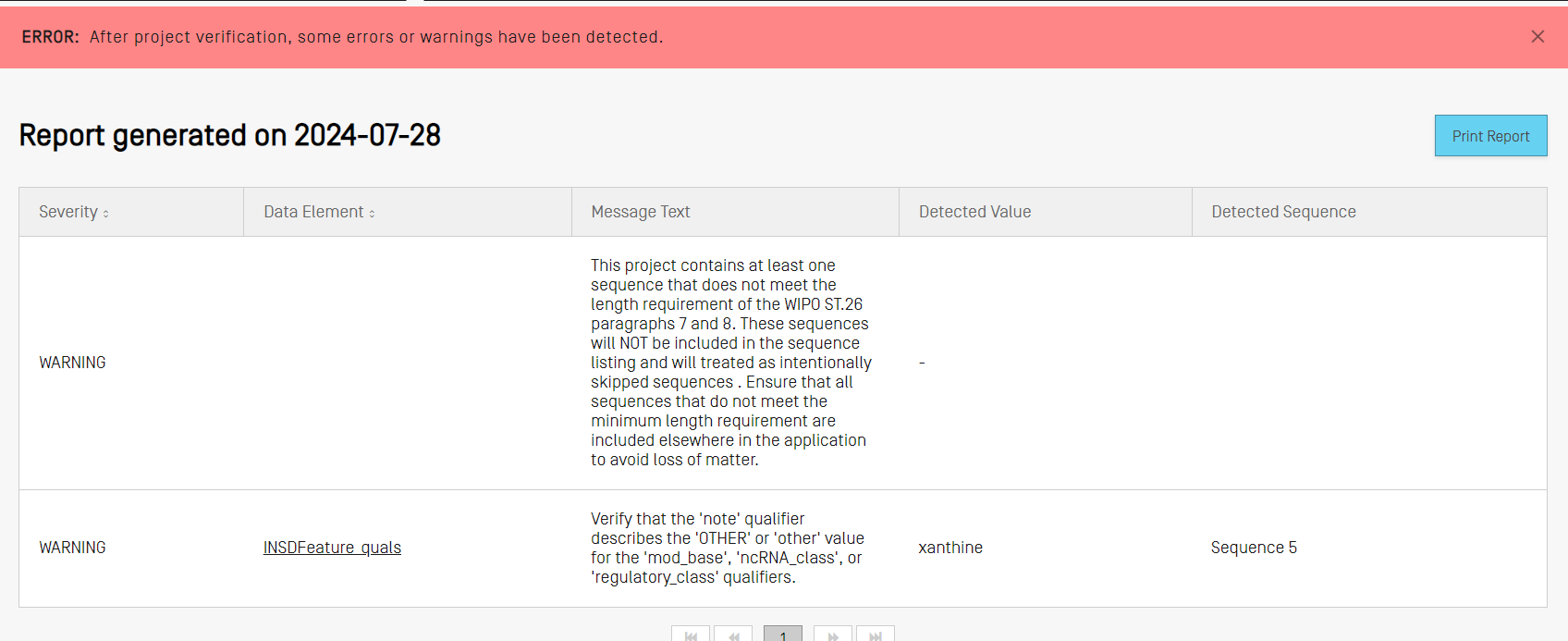
Generieren eines Sequenzprotokolls
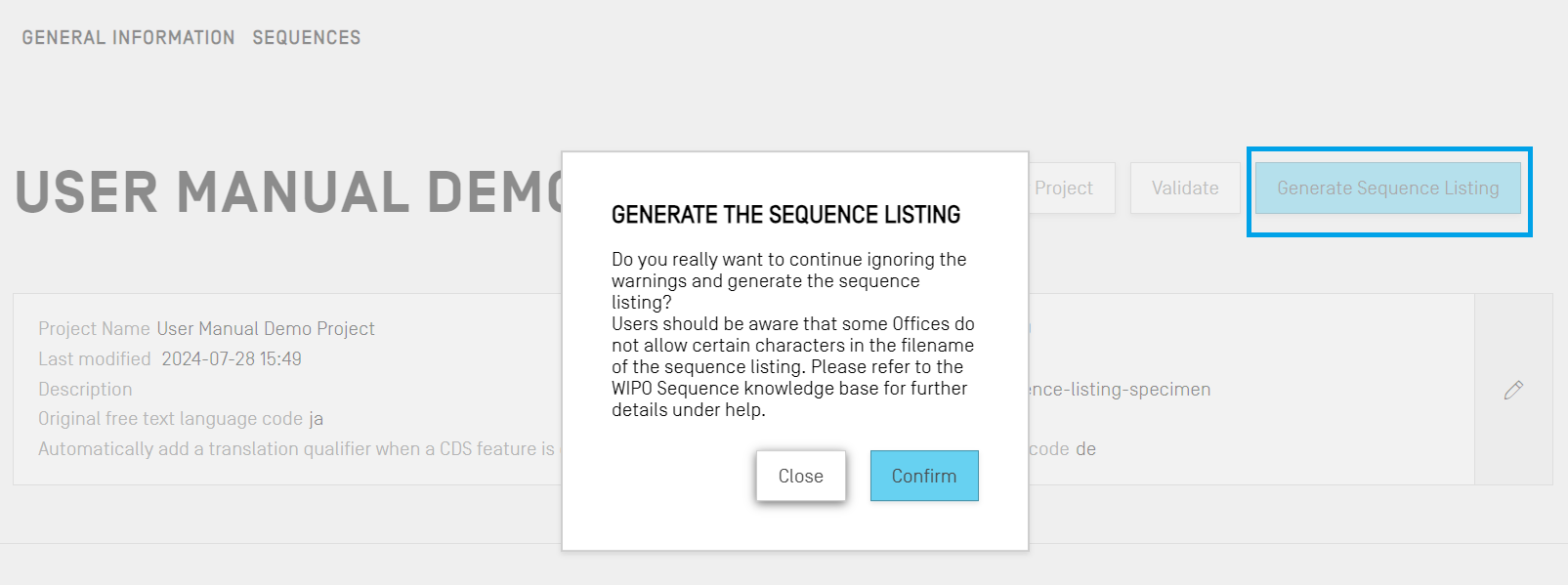
Die letzte und vielleicht wichtigste Aktion, die an einem Projekt durchgeführt werden kann, ist die Generierung des Sequenzprotokolls. Um das Sequenzprotokoll zu generieren, klicken Sie oben in der Ansicht Projektdetails auf die blaue Schaltfläche "Generate Sequence Listing" (Sequenzprotokoll generieren). Dadurch startet automatisch der Validierungsprozess, der zunächst für das Projekt ausgeführt wird.
Wenn das Projekt den Validierungsprozess besteht, öffnet sich eine Dialogbox, in der Sie wählen können, wo Sie das generierte ST.26-konforme Sequenzprotokoll (.xml) speichern möchten.
Wenn die Validierung des Projekts jedoch fehlschlägt, wird stattdessen die Ansicht Prüfbericht zusammen mit einem roten Banner angezeigt. Ist die Validierung erfolgreich, zeigt das Tool zeigt ein blaues Banner an.
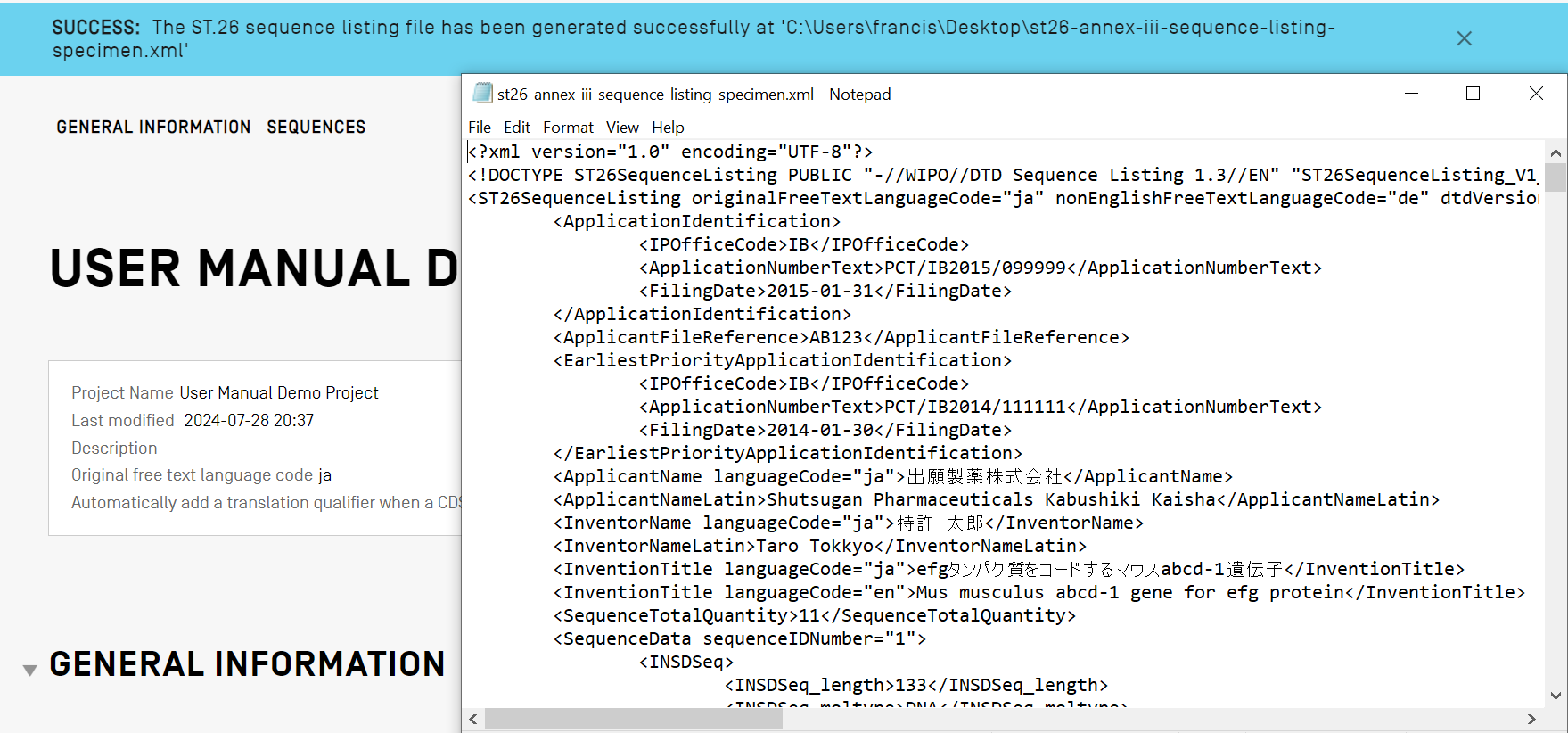
Sequenzprotokoll anzeigen
Mit WIPO Sequence können Sie ein Sequenzprotokoll in einem Format generieren, das visuell besser lesbar ist als XML. Nach der Erstellung eines Sequenzprotokolls können Sie die XML-Datei im HTML-Format anzeigen lassen oder als Textdatei exportieren. Mit einem beliebigen Internetbrowser können Sie das angezeigte Sequenzprotokoll im HTML-Format auch als PDF-Datei speichern. Die Exportfunktion erreichen Sie über die Ansicht "Display the Sequence Listing" (Sequenzprotokoll anzeigen).
Wenn ein Sequenzprotokoll für ein bestimmtes Projekt nicht erfolgreich generiert wurde, werden in der Ansicht "Display Sequence Listing" (Sequenzprotokoll anzeigen) die Schaltflächen "Display Sequence Listing" und "Export Sequence Listing as .txt file" deaktiviert und der Fehler wird angezeigt.
Sprachabhängige Freitext-Qualifier
Die Qualifier, die einen Freitextwert in einem Projekt zulassen, werden in der Ansicht "LANGUAGE DEPENDENT QUALIFIERS" (Sprachabhängige Qualifier) auf der Projektseite weiter referenziert. Immer wenn ein sprachabhängiger Qualifier dem aktuellen Projekt hinzugefügt wird, wird der Qualifier ebenfalls dieser Ansicht hinzugefügt.
Sie können den zugehörigen translatierten Freitextwert eines Qualifiers modifizieren, indem Sie auf den Wert "Qualifier Name" klicken, woraufhin sich ein Overlay mit einem Bearbeitungsbedienfeld unterhalb der Tabelle öffnet.
Zum Exportieren der Freitext-Qualifier im XLIFF-Dateiformat müssen Sie den Quellsprachen-Code und den Zielsprachen-Code angeben. Die translatierte Werte für die Zielwerte müssen von Translatoren bereitgestellt werden, bevor die XLIFF-Datei reimportiert wird.
Bitte achten Sie darauf, dass der Translations-Qualifier-Wert, der in der Spalte "Non English Qualifier Value" (Nicht-englischer Qualifier-Wert) erscheint, mit der ausgewählten Sprache übereinstimmt, die unter "Non English free text language code" (Nicht-englischer Freitext-Sprachcode) festgelegt wurde.
Durch Klicken auf "IMPORT FREE TEXT QUALIFIERS" (Freitext-Qualifier importieren), öffnet das Tool den Dateimanager. Dort können Sie die Ordner durchsuchen, um die (.XLIFF)-Datei auszuwählen, die Sie importieren möchten. Das System stellt mehrere Validierungsschritte bereit, die sicherstellen, dass die korrekten Zuordnungen zwischen den Ausgangs- und Zielsprachenwerten vorgenommen werden.
Freitext-Qualifier aus einer XLIFF-Datei importieren
Die ausgewählte Datei muss die folgenden Datenelemente enthalten:
- Den Namen des Projekts
- Den Zielsprachencode
- Den Ausgangssprachencode
- Für jedes XLIFF-Element:
- die eindeutige ID des Qualifiers (Format: eine Zahl mit dem vorangestelltem Buchstaben "q");
- den Qualifier-Wert im Ausgangssprachen-Tag;
- den Qualifier-Wert im Zielsprachen-Tag.
Sobald Sie die zu importierende Datei bestätigt haben, verlangt das Tool, dass Sie prüfen, ob Sie mit der Bestätigung einer Reihe von Verifizierungsschritten fortfahren möchten:
- Das System vergleicht den Projektnamen aus der Eingabedatei mit dem Namen des ausgewählten Projekts.
- Das System informiert Sie, wenn ein Qualifier nicht zugeordnet werden konnte.
- Das System informiert Sie über die Änderungen in Bezug auf die Ausgangssprache und die Qualifier-Werte.
- Das System informiert Sie über die Änderungen in Bezug auf die Zielsprache und die translatierten Werte der Qualifier.
Nach Abschluss dieser Schritte erscheint oben ein blaues Banner mit der Nachricht "SUCCESS: THE FREE TEXT QUALIFIER HAS BEEN IMPORTED SUCCESSFULLY" (Der Freitext-Qualifier wurde erfolgreich importiert) zusammen mit einem Importbericht, der im Detail die vorherigen und aktuellen importierten Werte für die sprachabhängigen Freitext-Qualifier anzeigt. Durch Klicken auf "RETURN TO FREE TEXT QUALIFIERS" (Zurück zu den Freitext-Qualifiern) kehren Sie zurück zur Ansicht Freitext-Qualifier.
Freitext-Qualifier im XLIFF-Format exportieren
Wenn Sie in der Ansicht oben auf die Schaltfläche "EXPORT FREE TEXT QUALIFIERS" (Freitext-Qualifier exportieren) klicken und dann im Dialogfeld den Dateinamen und Speicherort auswählen, um die Qualifier-Textdatei zu speichern, werden alle Freitext-Qualifier des Projekts exportiert und in einem XLIFF-Dateiformat gespeichert.
Die Datei enthält:
- Die Ausgangssprache des Projekts
- Die Zielsprache des Projekts
- Die Freitext-Qualifier
- Die translatierten Freitext-Qualifier-Werte
- Die zugehörigen Qualifier- und Merkmal-Informationen, die in der Tabelle bereitgestellt werden.
Diese XLIFF-Datei kann angezeigt, bearbeitet und wieder in das Tool importiert werden, nachdem die entsprechenden translatierten Werte bereitgestellt wurden.
Projektdaten eingeben
Ein Projekt ist, genauso wie ein generiertes Sequenzprotokoll, in zwei Abschnitte unterteilt, und zwar in Allgemeine Angeben und Sequenzen. Sie können Daten ausschneiden und in ein Projekt einfügen oder Daten aus einem anderen Projekt oder einem anderen Sequenzprotokoll importieren. Im Allgemeinen können Sie Projektfelder durch Klicken auf das Bleistiftsymbol bearbeiten. Pflichtfelder sind mit einem Sternchen (“*”) gekennzeichnet.
Allgemeine Angaben
Im Abschnitt Allgemeine Angaben können Sie Angaben zur Patentanmeldung eingeben, die verwendet werden, um das generierte Sequenzprotokoll mit dieser Anmeldung zu verknüpfen. Der erste Unterabschnitt “Application Identification” (Identifikation der Anmeldung) bezieht sich auf den Status der Patentanmeldung und Informationen zum ausgewählten Projekt. Die folgenden Anweisungen führen Sie Schritt für Schritt durch die Informationen, die in diesem Abschnitt angegeben müssen.
Schritt 1: Identifikation der Anmeldung
Um Angaben innerhalb des Unterabschnitts "Application Identification" zu bearbeiten, klicken Sie auf das markierte Stiftsymbol rechts neben dem Unterabschnitt. Geben Sie anschließend Angaben basierend auf den folgenden Schritten ein:
- Wenn der Anmeldung bereits eine Anmeldenummer erteilt wurde, müssen Sie den Code des Amts für geistiges Eigentum ("IP-Office") auswählen, bei dem die Anmeldung eingereicht wurde. Dies ist der WIPO ST.3-Code.
- Durch Auswahl des entsprechenden Optionsfeldes müssen Sie angeben, ob Ihnen die Anmeldenummer bereits erteilt wurde oder ob Sie das Aktenzeichen des Anmelders angeben haben.
- Falls die Anmeldenummer nicht vorliegt, MÜSSEN Sie in diesem Feld das Aktenzeichen des Anmelders angeben.
- Wenn bereits eine Anmeldenummer erteilt wurde, sollten Sie die vorgesehene Anmeldenummer eingeben.
- Wählen Sie dann das Anmeldedatum der Anmeldung in der Datumsauswahl aus, falls ein Datum erteilt wurde.
- Klicken Sie auf die blaue Schaltfläche "Speichern".
Im gezeigten Beispiel wurden auch alle optionalen Werte eingegeben:

Hinweis: Auch wenn alle obligatorischen Pflichtwerte eingegeben wurden, erscheint im Prüfbericht immer eine Warnung, die darauf hinweist, dass die Anmeldenummer fehlt: "The application identification number is absent. The application number is mandatory if the application number has been assigned." (Die Anmeldenummer fehlt. Die Anmeldenummer ist obligatorisch, falls eine solche erteilt wurde.).
Schritt 2: Prioritätsanmeldung(en)
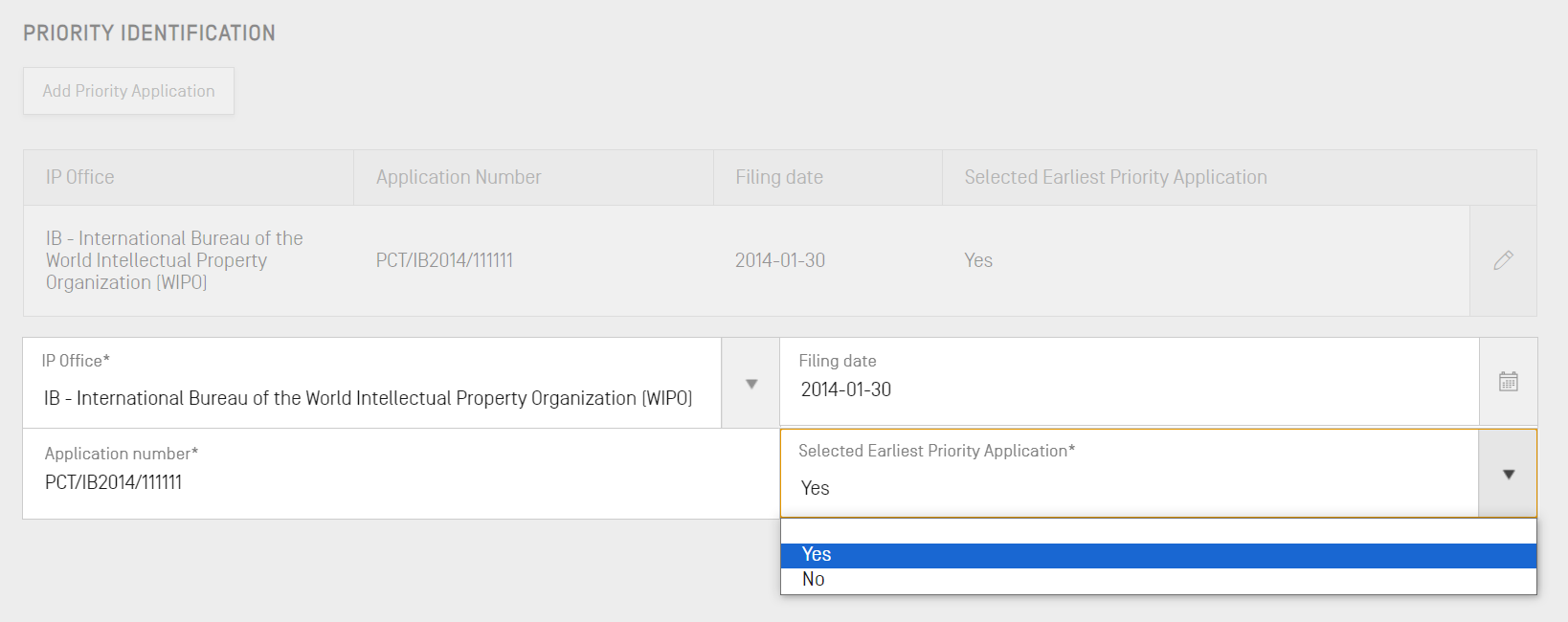
Als Nächstes werden, falls es eine Prioritätsanmeldung im Zusammenhang mit der vorliegenden Anmeldung gibt, diese Details im Unterabschnitt "Priority Identification" (Prioritäts-Identifikation) eingegeben. Zum Hinzufügen einer Prioritätsanmeldung zu dem Projekt klicken Sie im Abschnitt "Allgemeine Angaben" auf die Schaltfläche "Add Priority Application" (Prioritätsanmeldung hinzufügen) klicken.
Um die aktuell ausgewählte Prioritätsanmeldung als die früheste festzulegen, wählen Sie im Dropdown-Menü "Selected Earliest Priority Application" (Ausgewählte früheste Prioritätsanmeldung) die Option "Yes" (Ja). Dadurch wird diese Prioritätsanmeldung als die früheste Prioritätsanmeldung festgelegt oder der Status wird zu dieser geändert, wenn das Sequenzprotokoll erstellt wird. Um den Vorgang abzuschließen, klicken Sie auf die blaue Schaltfläche "Add Priority Application" (Prioritätsanmeldung hinzufügen).
Schritt 3: Anmelder/Erfinder
Um dem Projekt Daten zu einem neuen Anmelder oder Erfinder hinzuzufügen, klicken Sie im Abschnitt "General Information" (Allgemeine Angaben) der Ansicht "Project Details" (Projektdetails) auf die Schaltfläche "Add Inventor" (Erfinder hinzufügen) bzw. auf "Add Applicant" (Anmelder hinzufügen). Die Schritte für beide Aktionen sind identisch, sodass nachfolgend nur allgemeine Anweisungen aufgeführt sind. Dieser Vorgang muss jedoch zweimal wiederholt werden, wenn sowohl ein Anmelder als auch ein Erfinder dem Projekt hinzugefügt werden sollen, selbst wenn der Anmelder auch der Erfinder ist.
Wenn Sie auf die Schaltfläche klicken, öffnet sich ein Overlay mit zwei Optionsfeldern. Bei der Auswahl "Existing applicant/inventor" (Bestehender Anmelder/Erfinder) können Sie aus einem Dropdown-Feld wählen, das aktuell gespeicherte Personen und Organisationen innerhalb der lokalen Instanz des Desktop-Tools auflistet.
Bei der Auswahl "New applicant/inventor” (Neuer Anmelder/Erfinder) müssen Sie alle erforderlichen Angaben machen, die beim Anlegen einer neuen Person/Organisation verlangt werden.
Sobald die Angaben vollständig sind, klicken Sie auf die blaue Schaltfläche "Add Applicant" (Anmelder hinzufügen) bzw. "Add Inventor" (Erfinder hinzufügen).

Hinweis: Nur ein Anmelder ist erforderlich, damit das Sequenzprotokoll als gültig angesehen wird. Daher muss ein Anmelder und/oder Erfinder als Hauptanmelder gekennzeichnet werden. Dies ist der Anmelder/Erfinder, der in dem generierten Sequenzprotokoll erscheint.
Schritt 4: Bezeichnungen der Erfindung
Im letzten Schritt wird im Abschnitt "General Information" (Allgemeine Angaben) unter "Invention Title" die Bezeichnung der Erfindung hinzugefügt.
Gehen Sie folgendermaßen vor. um eine neue Bezeichnung der Erfindung hinzuzufügen:
- Klicken Sie auf die Schaltfläche "Add Invention title" (Bezeichnung der Erfindung hinzufügen).
- Es öffnet sich ein Overlay. Tragen Sie Sie hier die Bezeichnung der Erfindung ein und geben Sie außerdem an, in welcher Sprache die Bezeichnung bereitgestellt wird.
- Klicken Sie auf die blaue Schaltfläche "Add Invention title".

Gemäß WIPO ST.26 ist es für ein Sequenzprotokoll obligatorisch, dass die Bezeichnung der Erfindung in der Sprache der Einreichung angegeben wird. Ein Projekt kann jedoch optional auch mehr als eine Bezeichnung der Erfindung in zusätzlichen Sprachen enthalten, doch nur eine Bezeichnung der Erfindung pro Sprache. Jede neue Bezeichnung der Erfindung kann mittels der oben erwähnten Schritte hinzugefügt werden.
Sequenzdaten
Geben Sie im Abschnitt "Sequences" (Sequenzen) der Ansicht "Project Details" (Projektdetails) die technischen Informationen zu den Sequenzen selbst an. WIPO Sequence bietet verschiedene Möglichkeiten, Sequenzdaten in ein Projekt einzufügen, beispielsweise durch manuelles Erstellen, Importieren und Einfügen einer Sequenz. In den folgenden Unterabschnitten erhalten Sie weitere Informationen zu den Schritten, die zur Durchführung dieser Aktionen erforderlich sind.
Erstellen einer Sequenz
So erstellen Sie eine Sequenz in einem Projekt
- Klicken Sie auf die Schaltfläche "Create new sequence" (Neue Sequenz erstellen). Dies richtet den Fokus auf das Bedienfeld "Sequenz".
- Weisen Sie der Sequenz einen Namen zu, indem Sie den gewünschten Namen in das Feld "Sequence Name" eingeben. Alternativ können Sie das Feld "Sequence Name" leer lassen und WIPO Sequence vergibt einen Standardnamen für die neue Sequenz. Standardnamen beginnen mit "Seq" und einer darauf folgenden, iterativ aufsteigenden Nummer ("Seq_1", "Seq_2", "Seq_3").
Hinweis:: Der "Sequence Name" soll es dem Benutzer erleichtern, zwischen den Sequenzen in einem Projekt zu unterscheiden und erscheint nicht in der XML-Datei, die das Sequenzprotokoll enthält.
- Verwenden Sie das Dropdown-Feld "Molecule Type*" (Molekülart), um einen der drei in WIPO ST.26 zulässigen Molekülarten auszuwählen ("DNA", "RNA" oder "AA"). Wenn Sie eine Sequenz mit sowohl "DNA" als auch "RNA" Segmenten erstellen möchten, muss "DNA" als Hauptmolkülart ausgewählt werden.
- Geben Sie die Sequenzreste in das Feld "Residues*" ein oder fügen Sie sie dort ein.
- Geben Sie den Namen des Organismus in das Feld "Organism name*" ein oder fügen sie ihn dort ein. Um einen Organismusnamen aus der vordefinierten Liste von Organismusnamen auszuwählen, können Sie einfach mit der Eingabe des Namens des Organismus beginnen. Es erscheint eine Dropdown-Liste. Wählen Sie den gewünschten Namen des Organismus aus, falls vorhanden. Andernfalls werden Sie möglicherweise vom Tool aufgefordert, den Organismus der Datenbank für benutzerdefinierte Organismen hinzuzufügen, die lokal gespeichert ist.
- Lautet die Molekülart "DNA" oder "RNA", müssen Sie aus dem Dropdown-Feld "Qualifier Molecule Type" eine spezifische Molekülart auswählen, die automatisch mit den für die in Schritt 3 ausgewählte Molekülart geeigneten Werten gefüllt wird. Beachten Sie, dass für die Molekülart "AA" der Molekülart-Qualifier "Qualifier Molecule Type" automatisch mit dem Wert "protein" gefüllt wird.
- Wenn das Kästchen "Mark as an intentionally skipped sequence" (Als eine absichtlich übersprungene Sequenz markieren) aktiviert ist, wird die Sequenz in die resultierende XML-Datei, die das Sequenzprotokoll enthält, als absichtlich übersprungene Sequenz aufgenommen, d. h. sie hat das in WIPO ST.26 Absatz 58 angegebene Format. Beachten Sie, dass das Kästchen "Mark as an intentionally skipped sequence" (Als eine absichtlich übersprungene Sequenz markieren) aktiviert sein muss, wen Sie eine Sequenz mit weniger als 10 Nicht-"n"-Nukleotidresten oder weniger als 4 Nicht-"X"-Aminosäureresten speichern möchten. Wenn eine Sequenz absichtlich übersprungen wird, werden im Bedienfeld "Sequenz" alle Einschränkungen für die Angabe von Werten für Pflichtelemente aufgehoben und die resultierende gespeicherte Sequenz wird bei der Validierung des Projekts ignoriert.
- Wenn die Molekülart der Sequenz "DNA" ist, erscheint ein Feld mit der Aufschrift "The sequence contains both DNA & RNA fragments" (Die Sequenz enthält sowohl DNA- als auch RNA-Fragmente). Enthält die Sequenz sowohl DNA- als auch RNA-Fragmente, können Sie mithilfe dieses Kontrollkästchens die in WIPO ST.26, Absatz 55, geforderten Merkmale zur Beschreibung von DNA/RNA-Hybriden leicht hinzufügen. Aktivieren Sie das Kontrollkästchen "The sequence contains both DNA & RNA fragments" (Die Sequenz enthält sowohl DNA- als auch RNA-Fragmente), um das Bedienfeld "Sequence panel" zu erweitern und Felder hinzuzufügen, in denen Sie jedes DNA- und RNA-Fragment mit dem Merkmalschlüssel "misc_feature" und der entsprechenden Lage beschreiben können. Wählen Sie für jedes Fragment "DNA" oder "RNA" aus dem Dropdown-Menü im Feld "Molecule Type" (Molekülart) aus und geben Sie anschließend im Feld "Location" eine Lage an. Optional können Sie weiteren Text in dem Feld "Further text" hinzufügen. Sie können so viele dieser Merkmale wie nötig erstellen. Verwenden Sie dazu auf die Schaltfläche "Add new ‘misc_feature’ feature" (Neues 'misc_feature'-Merkmal hinzufügen). Beachten Sie, dass die gesamte Länge der Sequenz durch "misc_feature"-Merkmalschlüssel abgedeckt sein muss, und zwar so, dass jeder einzelne Rest entweder mit "DNA" mit "RNA" angezeigt wird.
- Um die Sequenz abzuschließen und zu speichern, klicken Sie auf die graue Schaltfläche "Create sequence" (Sequenz erstellen) oder auf die blaue Schaltfläche "Create & Display Sequence" (Sequenz erstellen und anzeigen). Wenn Sie auf die blaue Schaltfläche "Create & Display sequence" (Sequenz erstellen und anzeigen) klicken, öffnet sich, nachdem die Sequenz erstellt wurde, eine zusammenklappbare Sequenzanzeige, in der Sie alle Werte überprüfen können. Sei finden Sie in der Ansicht "Project Details" (Projektdetails), unterhalb des Abschnitts "Sequences" (Sequenzen).
Hinweis: Wenn es sich bei der Sequenz um eine Nukleotidsequenz ("DNA" oder "RNA") handelt, sind nur die in WIPO ST.26, Anhang I, Tabelle 1 aufgeführten Symbole zulässig (a, c, g, t, m, r, w, s, y, k, v, h, d, b, n). Beachten Sie, dass das Symbol "u" nicht zulässig ist. Symbole, die als Großbuchstaben eingegeben werden, werden in Kleinbuchstaben umgewandelt. Alle eingegebenen Symbole, die keine Buchstaben sind, werden automatisch entfernt (Leerzeichen, Zahlen, *, -, usw.).
Wenn es sich bei der Sequenz um eine Aminosäuresequenz ("AA") handelt, sind nur die in WIPO ST.26, Anhang I, Tabelle 3 aufgeführten Symbole zulässig (A, R, N, D, C, Q, E, G, H, I, L, K, M, F, P, O, S, U, T, W, Y, V, B, Z, J, X). Beachten Sie, dass nur einbuchstabige Aminosäurensymbole zulässig sind. Wenn Ihre Aminosäuresequenz durch dreibuchstabige Aminosäurensymbole wiedergegeben wird (z. B. Met-Arg-Leu-Trp-Ile), muss die Sequenz in einbuchstabige Symbole umgewandelt werden, bevor Sie sie in das Feld "Residues" (Reste) eingeben oder einfügen.p>
Die neu erstellte Sequenz befindet sich an der letzten Position in der Liste der Sequenzen mit der nächsten verfügbaren Sequenz-ID-Nummer. Um die Reihenfolge einer Sequenz in der Liste zu ändern, befolgen Sie diese Schritte.
Sequenz importieren
Sequenzen können auch direkt aus Dateien in ein Projekt importiert werden. Die akzeptierten Dateiformate sind in Abschnitt 3 aufgeführt. Bei Auswahl der Eingabedatei erkennt das WIPO Sequence Desktop-Tool automatisch das in der Datei verwendete Format.
- Klicken Sie zunächst auf die Schaltfläche "Import sequence" (Sequenz importieren).
- Klicken Sie auf "Upload file [.txt, .xml]" (Datei hochladen [.txt, .xml]). Nach Öffnen des Dialogfelds wählen Sie die Datei mit den zu importierenden Sequenzdaten aus. Das Desktop-Tool erkennt das verwendete Format und führt beim Import einige Validierungsprüfungen durch. Fünf Formate werden vom Tool zum Importieren von Sequenzen akzeptiert: RAW, Multi-Sequence, FASTA, ST.26 und ST.25.
- Bei Auswahl einer Datei im WIPO ST.25- oder ST.26-Format erscheint zunächst ein Kontrollkästchen "Select Range Sequences" (Sequenzbereich auswählen). Wird dieses Kästchen aktiviert, öffnet sich eine Tabelle mit den Sequenz-ID-Nummern jeder Sequenz in der Datei und der Reihenfolge, in der sie an die Liste der Sequenzen im Projekt angehängt werden. Wenn Sie nicht alle Sequenzen in das Projekt importieren möchten, können Sie den gewünschten Bereich von Sequenz-ID-Nummern angeben. Es kann sowohl eine einzelne Sequenz als auch eine Liste von durch Kommas getrennten Sequenzen oder ein Sequenzbereich in der Schreibweise x-y eingetragen werden, z.B. "1, 3, 7, 13-20, 30-50".
- Beim Importieren einer Datei im Multi-Sequence-Format wird das Kontrollkästchen "Select Range Sequences IDs" (Sequenzbereich-IDs auswählen) angezeigt, das bei Aktivierung eine Vorschautabelle mit den Sequenz-ID-Nummern der entsprechenden Sequenzen in der Datei sowie die Beschreibung für jede Sequenz anzeigt, einschließlich Sequenzname, Molekülart und Organismusname. Sie müssen den Bereich der Sequenz-ID-Nummern auswählen, den Sie in die Liste der Sequenzen innerhalb des Projekts importieren möchte. Standardmäßig wird die Gesamtzahl der Sequenzen der ausgewählten Sequenzprotokolldatei als ein Bereich angezeigt.
- Die letzten beiden Formate, die vom Prozess des Sequenzenimports akzeptiert werden, sind die Dateiformate RAW und FASTA. Diese Formate definieren nur eine einzelne Sequenz pro Datei. Wird eine RAW- oder FATA-Datei für den Import ausgewählt, zeigt das Tool das entsprechende Bedienfeld an. Fahren Sie fort, indem Sie alle Pflichtfelder ausfüllen.
- Nach dem Import navigiert das Tool zur Ansicht "Import Report" (Importbericht).
Sequenz einfügen
Zum Einfügen einer Sequenz an eine bestimmte Position in der Liste der Sequenzen klicken Sie auf Schaltfläche "Insert Sequence" (Sequenz einfügen). Ein Overlay mit einem Bedienfeld wird angezeigt. Zusätzlich zum Ausfüllen aller für die Erstellung einer Sequenz erforderlichen Informationen müssen Sie oben links im Bedienfeld die Position eingeben, an der die Sequenz in der Sequenzliste erscheinen soll. Zum Abschluss können Sie auf die Schaltflächen "Insert sequence" (Sequenz einfügen) oder "Insert & Display Sequence" (Sequenz einfügen & anzeigen) klicken.

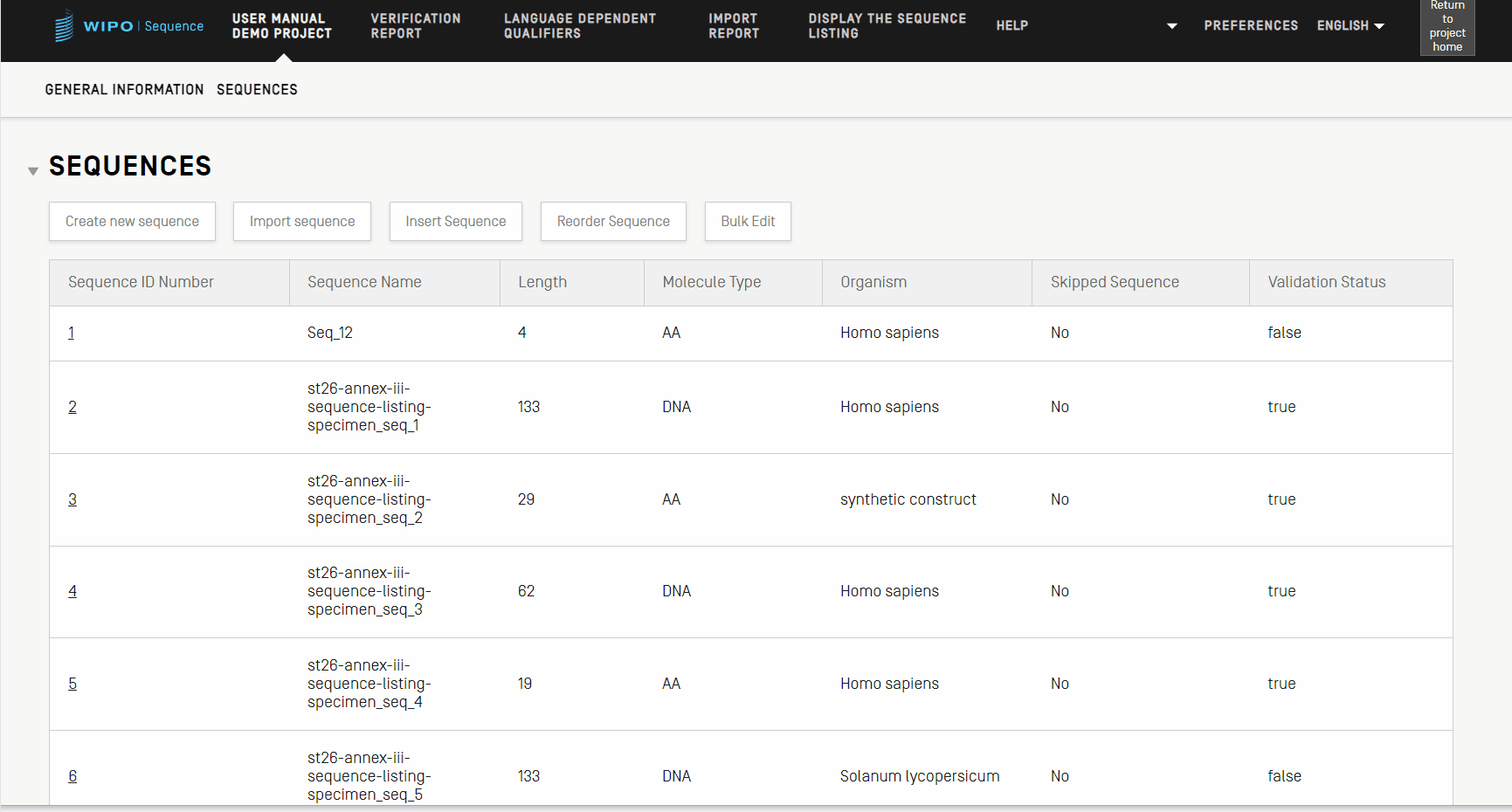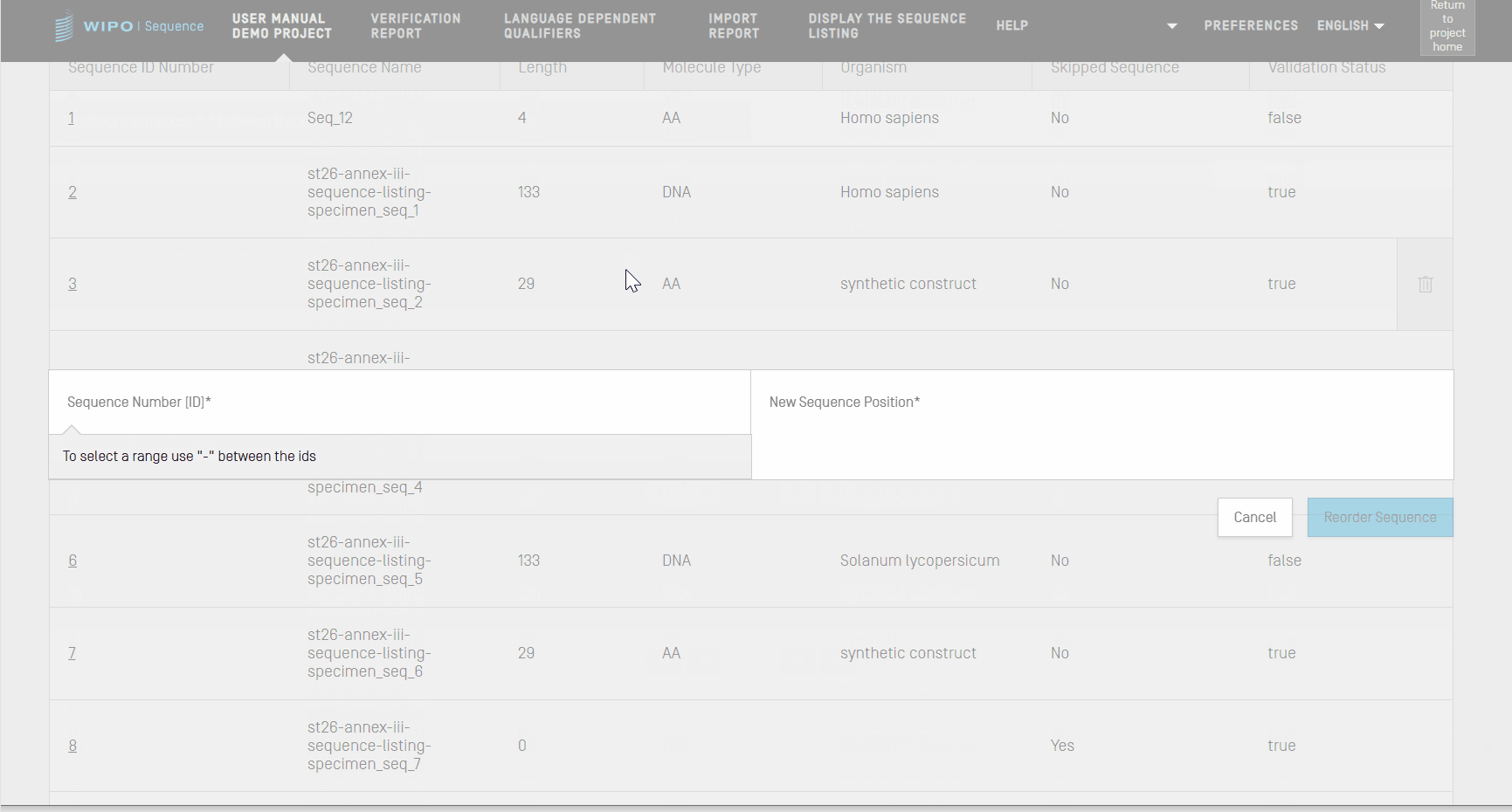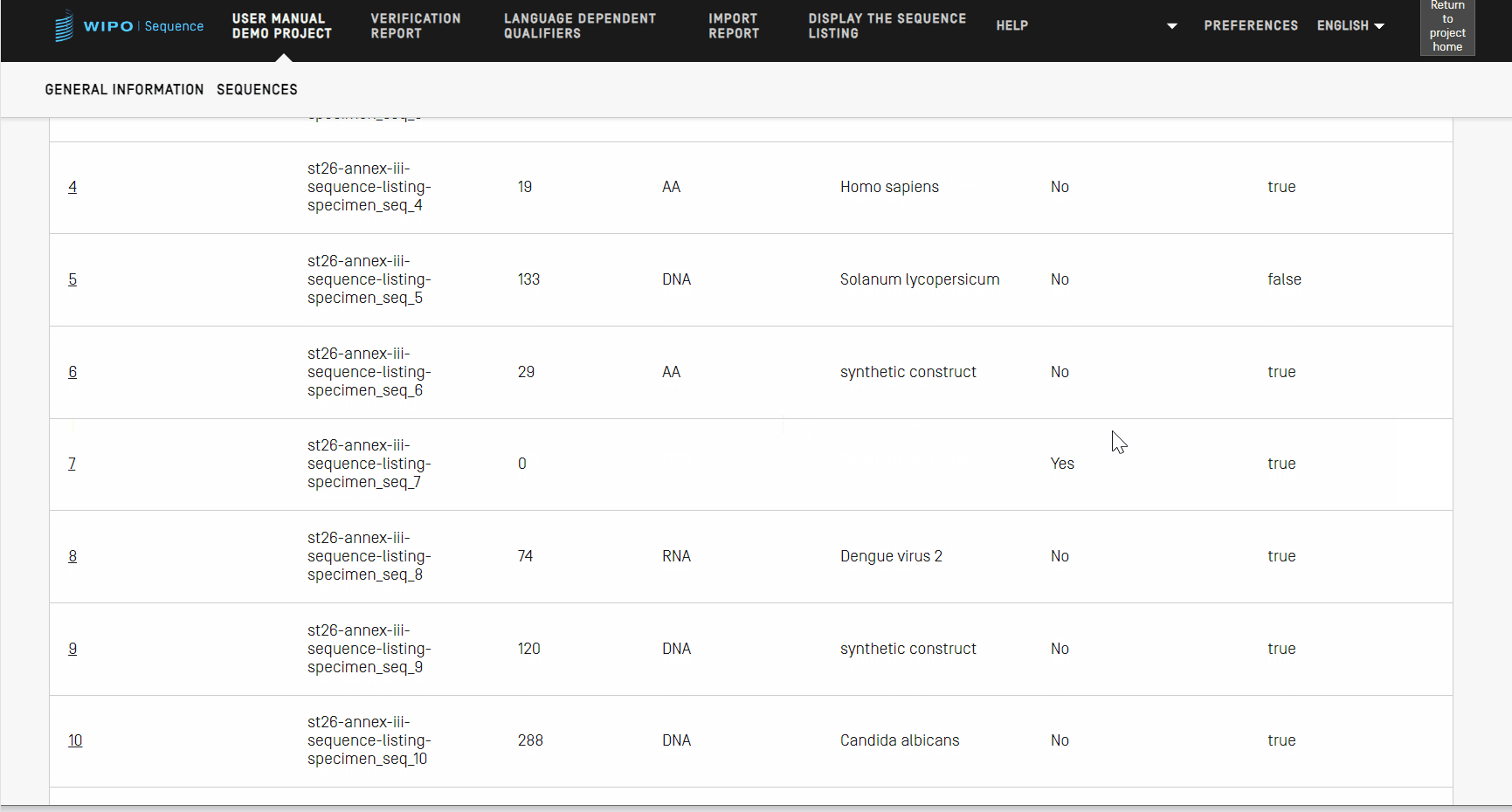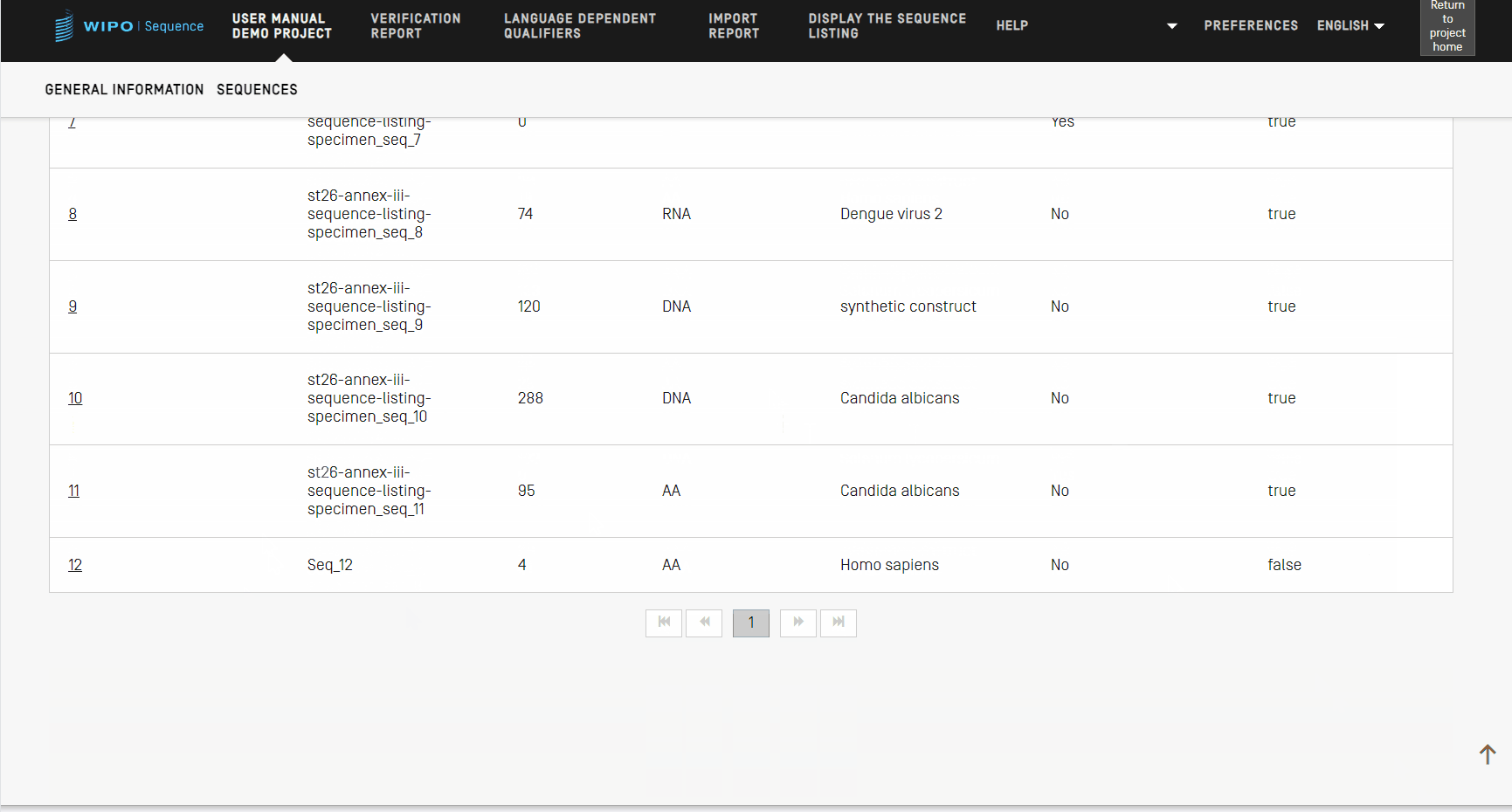
Sequenzen neu ordnen
Mit den folgenden Schritten können Sie eine Neuordnung vornehmen, d.h. die Reihenfolge der Sequenzen innerhalb der Liste der in einem Projekt bereitgestellten Sequenzen festlegen.
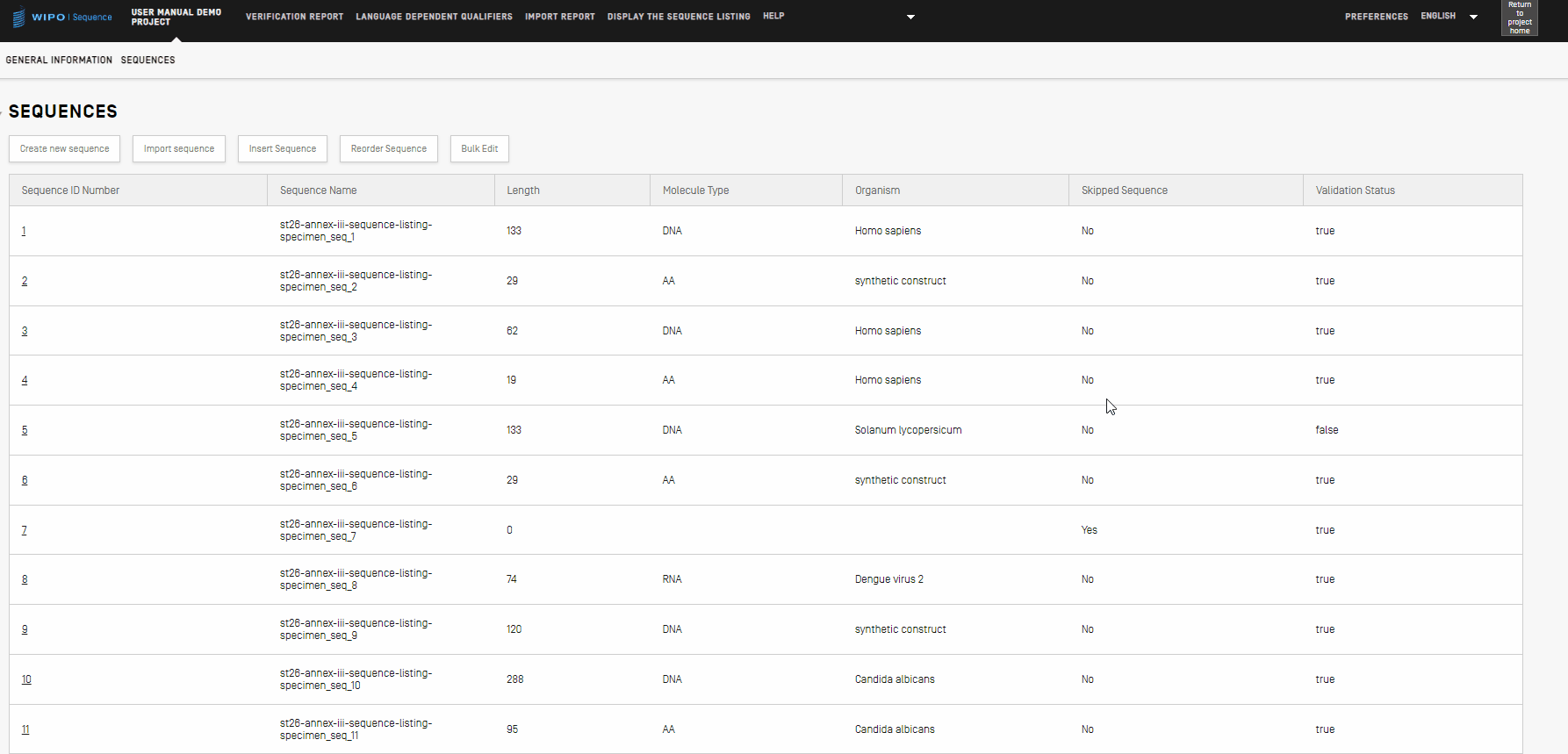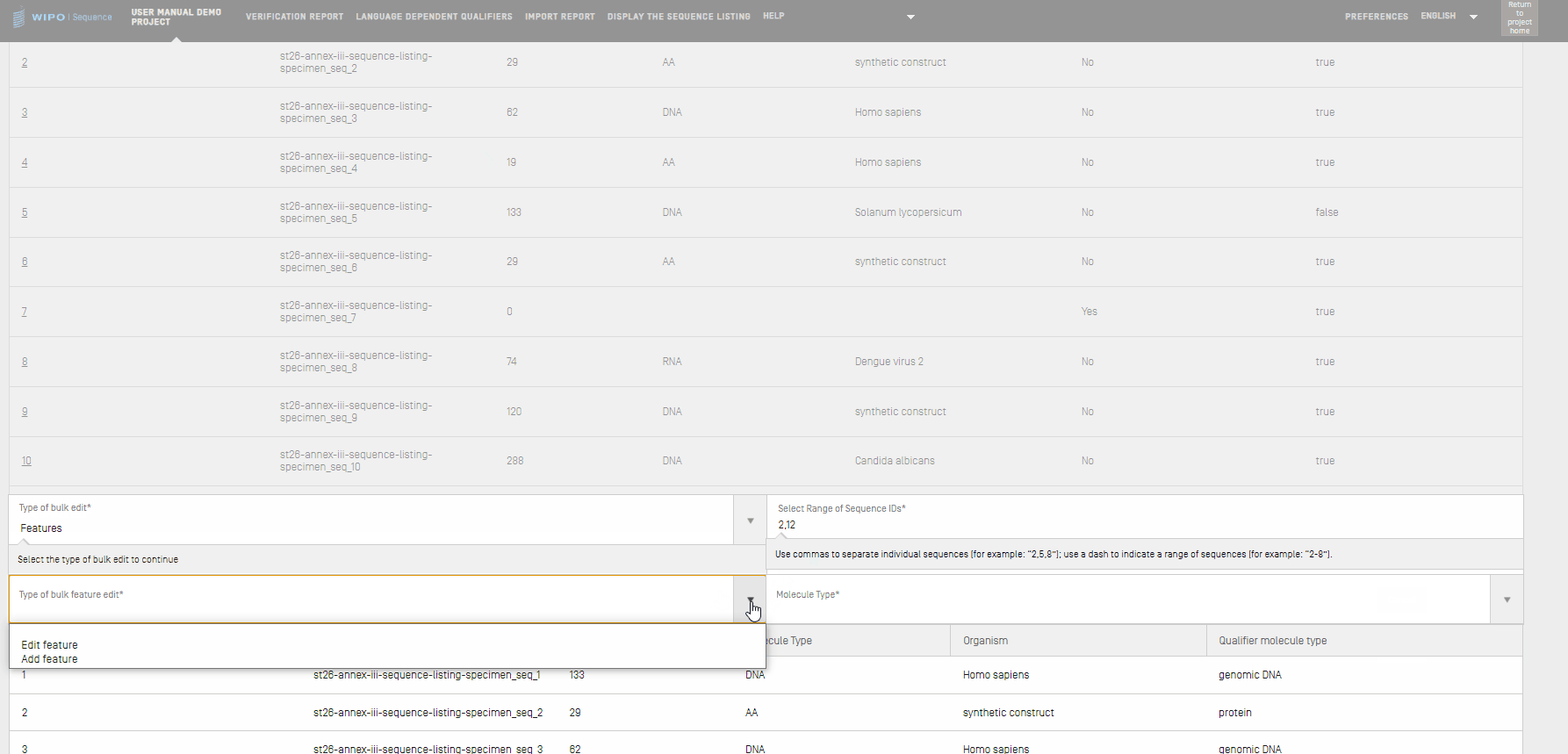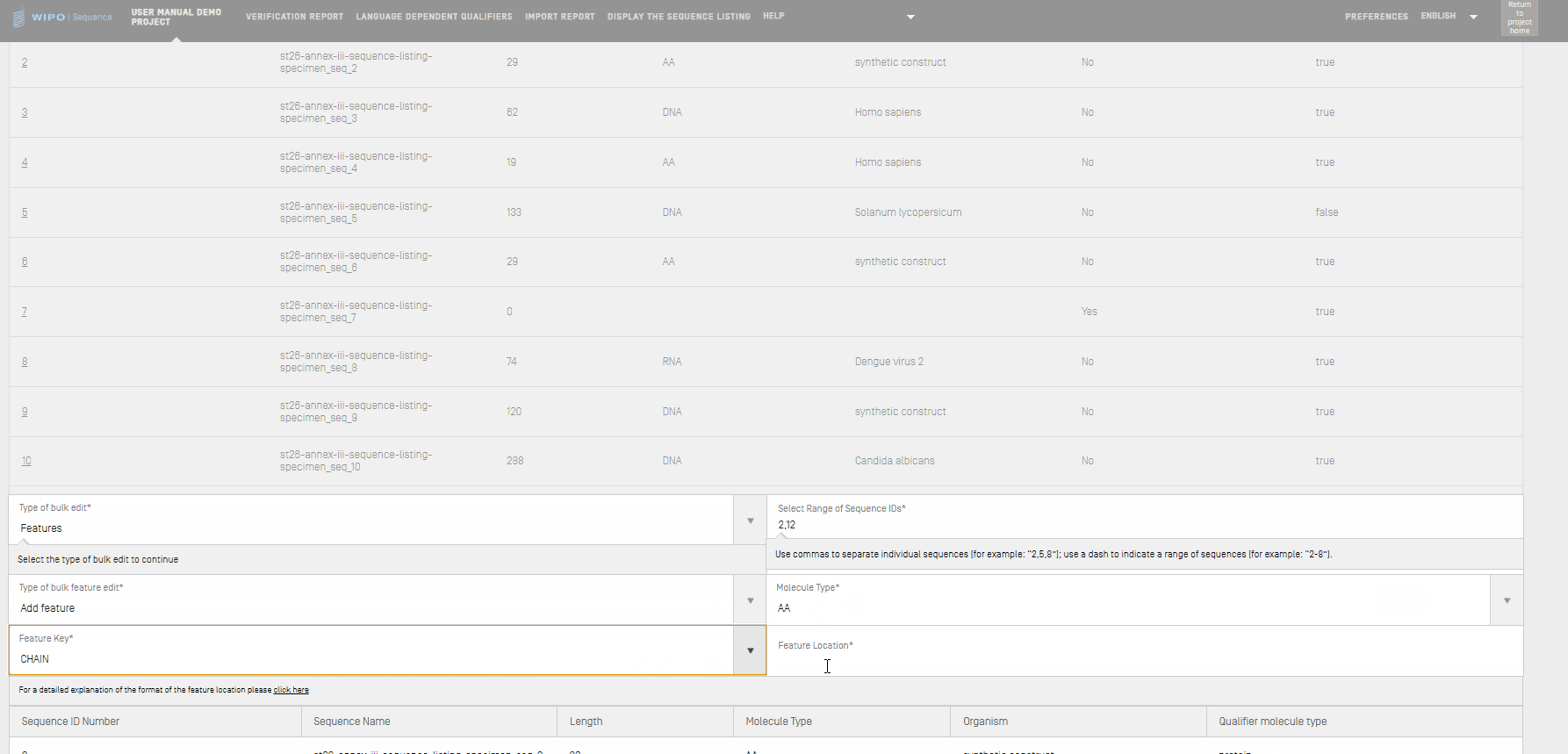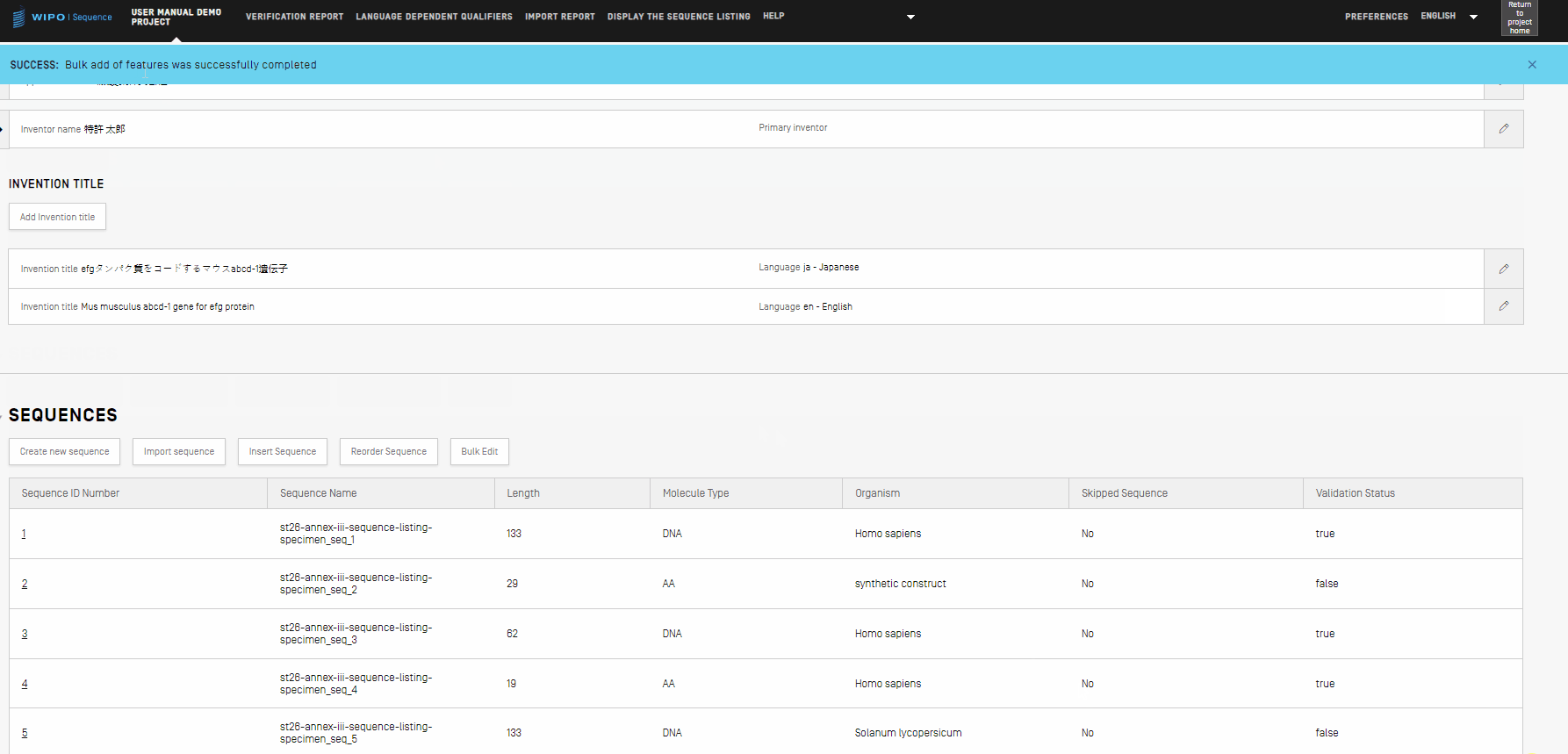
Bulk-Bearbeitung
Während jede der Sequenzen einzeln durch Klicken auf das Bleistiftsymbol bearbeitet werden kann, können Sie die Funktion zur Bulk-Bearbeitung verwenden, wenn Änderungen für mehrere Sequenzen erforderlich sind. Sie können zwar Sequenzen einzeln bearbeiten, dies wäre jedoch für Projekte mit einer großen Anzahl von Sequenzen nicht machbar. Es gibt eine Reihe von Feldern, die auf diese Weise bearbeitet werden können. Weitere Einzelheiten finden Sie weiter unten.
- Klicken Sie zunächst auf die Schaltfläche "Bulk-Bearbeitung"
- Wählen Sie unter "Art der Bulk-Bearbeitung" die Art der Mengenbearbeitung aus.
- Bei Auswahl von "Qualifier molecule Type" (Molekülart-Qualifier) werden Sie aufgefordert, die Art der Nucleinsäuresequenzen auszuwählen, auf die die Bulk-Bearbeitung angewendet werden soll. Das System warnt auch davor, dass der Qualifier "mol_type" für Sequenzen, in denen Organismus = "synthetic sequence" ist, "other DNA" oder "other RNA" sein muss. Wenn Sie diese Werte ändern, wird bei der Projektvalidierung ein Fehler generiert. Das System zeigt eine Vorschau der Sequenzen für die Bulk-Bearbeitung mit den angegebenen Merkmalen an. Während der Bearbeitung informiert Sie das System den Benutzer darüber, dass NUR bei Nucleinsäuresequenzen der Wert des Qualifiers "mol_type" bearbeitet werden kann (da der gleiche Wert für die Aminosäuresequenzen vom System automatisch auf "protein" gesetzt wird).
- Bei Auswahl von "Organism" (Organismus) müssen Sie den Bereich der zu bearbeitenden Sequenz-IDs eingeben. Haben Sie beispielsweise entschieden, den Wert des Organismus durch "synthetic construct" zu modifizieren, teilt Ihnen das System mit, dass der Qualifier "mol_type" je nach Molekülart automatisch zu "other DNA" oder "other RNA" geändert wird.
- Bei Auswahl von "Merkmale" müssen Sie angeben, ob Sie vorhandene Merkmale bearbeiten oder neue hinzufügen möchten. Sie müssen die "Molecule Type" (Molekülart-Qualifier) und den Bereich der zu bearbeitenden Sequenz-IDs eingegeben.
- Wenn Sie you unter "Art der Bulk-Bearbeitung" "Edit feature" (Merkmale) auswählen, beispielsweise indem Sie den Wert für die Lage eines CDS-Merkmals durch "complement(join(1..30,61.. 90))" modifizieren, sollten Sie zunächst Folgendes in das Textfeld "Select Range of Sequence IDs" (Bereich der Sequenz-IDs auswählen) eintragen: die relevanten Sequenz-IDs, die Molekülart unter "Molocule Type", sowie den relevanten Merkmalschlüssel unter "Feature Key" sowie seine Merkmals-Lage unter "Feature Location". Wählen Sie zum Abschluss "Edit sequences" (Sequenzen bearbeiten).
- Wenn Sie you unter "Type of bulk edit" (Art der Bulk-Bearbeitung) "Add feature" (Merkmale) auswählen, beispielsweise indem Sie ein neues Merkmal "CHAIN" mit der Merkmals-Lage "1..4" hinzufügen, sollten Sie zunächst Folgendes in das Textfeld "Select Range of Sequence ID" (Bereich der Sequenz-IDs auswählen) eintragen: die relevanten Sequenz-IDs, die Molekülart unter "Molocule Type", sowie den relevanten Merkmalschlüssel unter "Feature Key" sowie seine Merkmals-Lage unter "Feature Location".
- Bei Auswahl von "Bulk-Überspringen" müssen Sie im Textfeld "Select Range of Sequence IDs" (Bereich der Sequenz-IDs auswählen) die Sequenzbereich-IDs angeben, die das System überspringen soll.
- Bei Auswahl von "Bulk-Löschen" müssen Sie im Textfeld "Select Range of Sequence IDs" (Bereich der Sequenz-IDs auswählen) die Sequenzbereich-IDs angeben, die das System löschen soll.
- Bei jeder Bulk-Bearbeitung informiert Sie das Tool, nachdem Sie die Schaltfläche "Edit sequences" (Sequenzen bearbeiten) klicken, durch die Anzeige eines blauen Banners über den Erfolg der Operation. Ein Beispiel ist unten dargestellt:
Merkmaldaten eingeben
Gemäß WIPO-Standard ST.26 MUSS jede Sequenz mindestens ein Merkmal haben, das mit ihr verknüpft ist. Jedes "source"-Merkmal muss zwei obligatorische Qualifier aufweisen: "organism" und "mol_type".
Die Merkmaltabelle umfasst drei Spalten: Merkmalschlüssel, Lage des Merkmals innerhalb der genetischen Sequenz und die mit einem einzelnen Sequenzmerkmal verknüpften Qualifier.
Die Merkmals-Lage gibt an, in welchem Segment der Sequenz das Merkmal vorhanden ist. Die zulässigen Formate zum Festlegen der Merkmals-Lage sind in WIPO ST.26 beschrieben und sind wie folgt:
- Einzelne Restenummer: x
- Restenummern, die ein Sequenzintervall begrenzen: x..y
- Reste vor der ersten oder nach der letzten angegebenen Restenummer: < x, >x, <x..y, x..>y, <x..>y
- Eine Stelle zwischen zwei benachbarten Nukleotiden: x^y
- Restenummern, die durch eine ketteninterne Quervernetzung zusammengeführt werden: x..y
Lage-Operatoren können verwendet werden, um komplexe Lagebeschreibungen zu bilden:
- "join (location, location, … location)": Die Lagen werden zu einer zusammenhängenden Sequenz verbunden (von einem Ende zum anderen).
- "order (location, location, … location)": Die Elemente werden in der angegebenen Reihenfolge gefunden, aber es ist keine Aussage, ob das Verbinden dieser Elemente sinnvoll ist.
- "complement (location)": Zeigt an, dass sich das Merkmal auf dem Strang befindet, der komplementär zu dem durch den Lagedeskriptor angegebenen Sequenzintervall ist, wenn es in 5'- zu 3'-Richtung oder in der Richtung gelesen wird, die die 5'- zu 3'-Richtung nachahmt.
Um der Sequenz ein neues Merkmal hinzuzufügen, klicken Sie im Abschnitt "Features" (Merkmale) der ausgewählten Sequenz auf "Add feature" (Merkmal hinzufügen). Qualifier können in dieser Phase ebenfalls dem Merkmal hinzugefügt werden und sind im nächsten Abschnitt beschrieben.
CDS-Merkmale
Die CDS-Merkmalsart wird verwendet, um die Codierungssequenz für ein Protein zu beschreiben. Ein CDS-Merkmal kann optional die Aminosäuretranslation des Segments der Sequenz enthalten, zu der es gehört. Es wird, sofern die erforderliche Mindestlänge erfüllt ist, als eine separate Sequenz innerhalb des Projekts angezeigt. Innerhalb des CDS-Merkmals der Originalsequenz ist eine Referenz zu der Sequenz-ID der translatierten Aminosäuresequenz im Qualifier "protein_id" angegeben.
Beim Erstellen eines "CDS"-Merkmals für eine Sequenz kann der Qualifier "translation" (mit dem Standardwert 1 – "Standard Code" des genetischen Codes) automatisch zum CDS-Merkmal mit einem Qualifier-Wert, der dem translatierten Resteanteil der Sequenz entspricht (wie durch die Merkmals-Lage angegeben), hinzugefügt werden. Sie können eine zugehörige "protein-ID" und eine separate Aminosäuresequenz können auch dadurch generieren, dass Sie das Kontrollkästchen in den "Basic Information" (Allgemeinen Informationen) oben auf der Seite der Projektdetails aktivieren. Dieser Qualifier ist jedoch nicht obligatorisch und kann nach der Generierung gelöscht werden. Sie können einen Qualifier "translation" und "protein_id" auch manuell erstellen, der auf die zugehörige translatierte Sequenz-ID verweist, die Sie gleichermaßen erstellt haben.
Hinweis: Ab Version 2.1.0 ist das Kontrollkästchen "Automatically add a translation qualifier" (Automatische Hinzufügung eines Translations-Qualifiers) standardmäßig aktiviert.

Die automatische Erstellung eines CDS-Merkmal-Qualifiers umfasst die folgenden Schritte:
- Klicken Sie in der jeweiligen Sequenzanzeige auf die Schaltfläche "Add feature" (Merkmal hinzufügen) und wählen Sie "CDS" als Merkmalschlüssel aus. Wenn das Kontrollkästchen "Automatically add a translation qualifier" (Automatische Hinzufügung eines Translations-Qualifiers) in "Basic Information" aktiviert ist, wird automatisch ein "translation"-Qualifier, sein Wert und ein "protein_id"-Qualifier sowie die zugehörige separate Aminosäuresequenz (falls zutreffend) hinzugefügt, wenn ein CDS-Merkmal einer Nucleotidsequenz hinzugefügt wird.
- Sie können einen Translations-Qualifier auch manuell erstellen.
- Nachdem Sie die Bearbeitung des Merkmals und der zugehörigen Qualifiers abgeschlossen haben, müssen Sie auf "Create Feature" (Merkmal erstellen) klicken, um es zu speichern. Ein resultierendes CDS-Merkmal wird zu der zugeordnet Sequenz dargestellt.
Erfüllt der Wert des Translations-Qualifiers die Mindestlängenanforderungen, erstellt das Tool eine neue Sequenz für das Projekt mit den folgenden Attributen:
- Sequenz-ID-Nummer = Der nächste verfügbare Wert für die Sequenz-ID-Nummer
- Länge = Länge der translatierten Sequenz
- Sequenzname = Der im Feld "Sequence Name" des Qualifiers "translation" angegebene Wert. Wenn kein Name angegeben wurde, wird der Standard-Sequenzname angegeben ("Seq_#").
- Molekülart = "AA"
- Organismusname = Derselbe Wert, der für die Originalsequenz angegeben wurde
- Molekülart-Qualifier = "protein"
- Sequenzreste = Translatierte Werte der Originalsequenz
Hinweis: Betreffend die Erstellung der translatierten Sequenz wird die separate translatierte Sequenz nur dann erstellt, wenn sie über mindestens 4 spezifisch definierte Reste verfügt (z.B. "AXTG" zählt als 3 Zeichen). Wenn bei der Modifizierung des Qualifiers "translation" der Qualifier-Wert weniger als 4 spezifisch definierte Reste enthält, wird die zugehörige Sequenztranslation entfernt, ebenso wie der Qualifier "protein_id".
Hinweise zu CDS-Merkmalen bei der Aufnahme eines Qualifiers "pseudo" oder "pseudogen":
Stellen Sie sicher, dass die automatische Translation deaktiviert ist, wenn Sie einem CDS-Merkmal einen Qualifier "pseudo" oder "pseudogen" hinzufügen. Ist die automatische Translation nicht abgeschaltet wenn einem CDS-Merkmal ein Qualifier "pseudo" oder "pseudogen" hinzugefügt wird, dann wird bei der Aktualisierung des CDS-Merkmals automatisch ein Qualifier "translation" hinzugefügt. Um diesen Fehler zu korrigieren, deaktivieren Sie die automatische Translation für das Projekt, öffnen Sie das CDS-Merkmal, löschen die Qualifier "translation" und "protein-id" und aktualisieren Sie dann das Merkmal.
Möchten Sie den Translations-Qualifier automatisch generieren, können der Wert der Translationstabelle und der Sequenzname im Bearbeitungsbedienfeld des Qualifiers eingestellt werden. Bei der Erstellung des Merkmals führt das Tool die Translation durch und fügt dann dem Merkmal einen "protein_id"-Qualifier sowie eine neue Sequenz mit dem Translationswert hinzu.
Die Translation wird nur dann ein weiteres Mal durchgeführt, wenn die Merkmals-Lage oder einer der Qualifier "transl_table", "transl_except" oder "codon_start" seine Werte ändert. In diesem Fall wird die verknüpfte Sequenz aktualisiert.
Hinweis: Wenn der Translationswert geändert wird, wird der Wert der verknüpften Sequenz automatisch aktualisiert. Wird jedoch die verknüpfte Nukleotidsequenz modifiziert, ändert sich der Wert des Translations-Qualifiers nicht. Wird der "protein_id"-Qualifier nach der Erstellung modifiziert, verliert die verknüpfte Sequenz ihre Verknüpfung zur Originalsequenz.
Hinweise zur Verwendung des Stopp-Codons:
Normalerweise sollten Stopp-Codons nur am Ende eines CDS-Merkmals zu finden sein, um den Endpunkt der kodierten Aminosäuresequenz anzuzeigen. Sie sollten niemals in der Mitte eines CDS-Merkmals zu finden sein, es sei denn es liegt ein Qualifier "transl_except" vor, der angibt, dass das Stopp-Codon in eine bestimmte Aminosäure translatiert werden soll.
Wird ein Stopp-Codon in der Mitte eines CDS-Merkmals gefunden (unten in gelb hervorgehoben), und es liegt kein Qualifier "transl_except" vor, der angibt, dass das Stopp-Codon in eine bestimmte Aminosäure translatiert werden soll, sollte das Tool die Translation an dieser Stelle beenden und in einem roten Banner die Fehlermeldung anzeigen, dass keine Translation generiert wird.
Im Validierungsbericht sollte ein Fehler aufgeführt werden, um Sie darauf hinzuweisen, dass es ein Problem mit seiner Kodierungssequenz gibt.
Qualifier-Daten eingeben
Qualifier werden verwendet, um Informationen über Merkmale bereitzustellen, die über die durch den Merkmalschlüssel und die Merkmals-Lage vermittelten Angaben hinausgehen. Es gibt drei Formatarten für Werte, um verschiedene Arten von Informationen zu berücksichtigen, die durch Qualifier übermittelt werden:
- Freitext
- kontrolliertes Vokabular oder Aufzählungswerte (z. B. eine Zahl oder ein Datum)
- Sequenzen
Um die Qualifier für ein Merkmal anzuzeigen, müssen Sie zunächst das Merkmal aus der Merkmaltabelle der entsprechenden Sequenz auswählen.
Bereits vorhandene Qualifier können durch Klicken auf das Stiftsymbol rechts neben jeder Zeile bearbeitet werden oder Sie können durch Klicken auf die Schaltfläche "Add qualifier" (Qualifier hinzufügen) einen neuen Qualifier zum aktuell ausgewählten Merkmal hinzufügen.

Beim Bearbeiten oder Hinzufügen eines Qualifiers werden diese beiden Felder angezeigt: der "Qualifier Name", der aus einer Dropdown-Liste ausgewählt werden kann, und der "Qualifier Wert".
Das Feld "Qualifier Value" (Qualifier-Wert) verhält sich je nach Art des Qualifiers unterschiedlich:
- Qualifier mit vordefinierten Werten: Das Wertefeld ist ein Dropdown-Feld, in dem Sie einen der vordefinierten Werte für den Qualifier auswählen können.
- Qualifier mit Freitext: Das Wertefeld ist ein Freitextfeld. Zusätzlich zum Qualifier-Namen und dem Qualifier-Wert, der nur den englischen Wert enthält, werden zwei weitere Felder angezeigt, mit denen Sie sowohl den Sprachcode (z.B. "ru") als auch den entsprechenden Sprachwert im "Non English Qualifier Value", also im nicht-englischen Freitext-Sprachcode, angeben können. Dem Feld "Language code" (Sprachcode) wird derselbe Wert zugewiesen wie dem Feld "Non English free text language code" (Nicht-englischer Freitext-Sprachcode) in den Projektdetails. Sie können eine Reihe von nicht-englischen Werten für jede ausgewählte Sprache entweder durch manuelle Eingabe oder durch Importieren der richtigen zugehörigen Sprache aus einer XLIFF-Datei bereitstellen.
- Qualifier mit vordefiniertem Format: Das Wertefeld ist ein Freitext-Feld, aber der eingegebene Wert wird validiert, um sicherzustellen, dass er den spezifischen Regeln in WIPO ST.26 Anhang I, Abschnitt 6 entspricht.
- Qualifier ohne zulässigen Wert: Das Feld für den Qualifier-Wert kann nicht bearbeitet werden.
Zum Schluss müssen Sie auf die blaue Schaltfläche "Create Qualifier" (Qualifier erstellen) klicken, um den neu erstellten Qualifier hinzuzufügen, oder auf "Save" (Speichern), um die Änderungen an einem bereits vorhandenen Qualifier zu speichern. Im letzten Schritt, nachdem der/die Qualifier hinzugefügt/modifiziert wurde(n), müssen Sie auf die Schaltfläche "Update feature" (Merkmal aktualisieren) am unteren Rand des Merkmal-Overlays klicken, um fortzufahren.
2.3 Ansicht Personen & Organisationen
Diese Ansicht verwaltet alle lokal gespeicherten Personen und Organisationen.
Person oder Organisation erstellen
Um eine neue Person oder Organisation zu erstellen, muss der Benutzer in der Ansicht Personen & Organisationen beginnen. Klicken Sie auf den oben in der Ansicht befindlichen Link "CREATE NEW PERSON OR ORGANIZATION" (Neue Person oder Organisation erstellen).

In der neuen Ansicht müssen Sie zumindest die Pflichtfelder (mit einem Sternchen "*" gekennzeichnet) ausfüllen, die den Angaben zur neuen Person/Organisation entsprechen. Für den Anmelder/Erfinder ist dies nur der Name (sofern in lateinischen Buchstaben angegeben) und die Sprache.
Liegt der Name der Person oder Organisation nicht in lateinischen Schriftzeichen vor, sollte die lateinische Version des Namens im Feld "Name Latin" angegeben werden. Werden diese Informationen nicht angegeben, so wird das Projekt nicht validieren, wenn das ST.26-Sequenzprotokoll validiert oder generiert wird.
2.4 Ansicht Benutzerdefinierte Organismen
Um benutzerdefinierte Organismen zu erstellen, bearbeiten, importieren, exportieren oder löschen, müssen Sie in der Ansicht Organismen beginnen.
Benutzerdefinierten Organismus erstellen

Zum Erstellen eines neuen benutzerdefinierten Organismus klicken Sie auf den in der Ansicht oben rechts befindlichen Link "CREATE NEW ORGANISM" (Neuen Organismus erstellen). Geben Sie im folgenden Bildschirm den Namen des neuen Organismus ein und klicken Sie auf "Speichern". Wenn eine Beschreibung dieses benutzerdefinierten Organismus erforderlich ist, kann diese optional wie abgebildet hinzugefügt werden. Um die Details zu bearbeiten, klicken Sie auf den Namen des Organismus.
Benutzerdefinierte Organismen exportieren
Alle benutzerdefinierten Organismen und deren Beschreibung, die im Tool gespeichert sind, können exportiert und in einer Textdatei gespeichert werden, um sie außerhalb des Tools zu ändern oder zu einem späteren Zeitpunkt zu importieren. Um diese Liste zu exportieren, wählen Sie zunächst "EXPORT CUSTOM ORGANISMS" (Benutzerdefinierte Organismen exportieren), wie unten hervorgehoben.
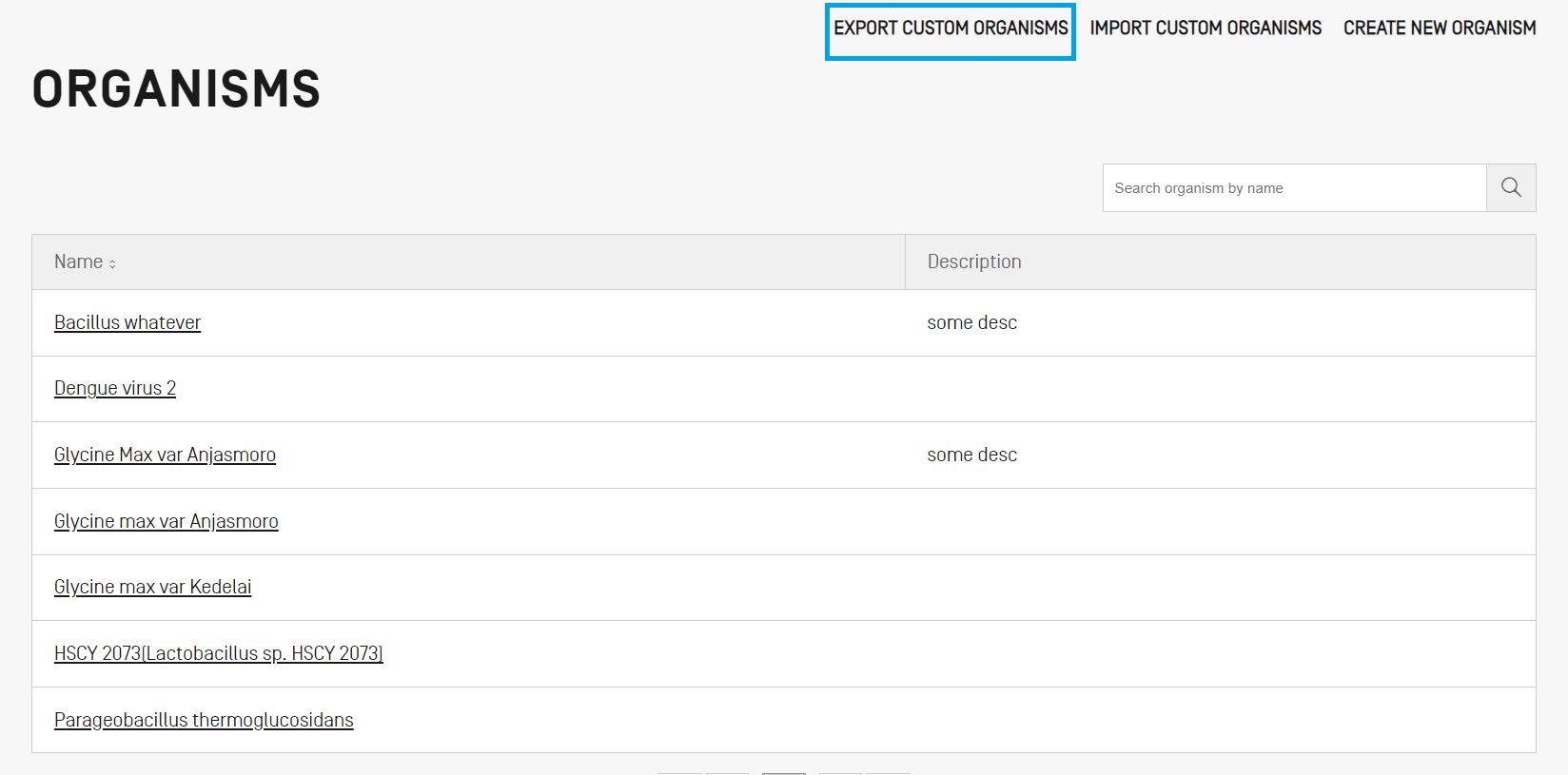
Als Nächstes öffnet ein Dialogfeld, in dem Sie den Namen der Datei und den gewünschten Speicherort auswählen können.

Die exportierte Datei ist eine Text-Datei, die sowohl den Namen als auch die Beschreibung des Organismus enthält, der bearbeitet und in das Tool importiert werden könnte. Ein Beispiel herunterladen.
Benutzerdefinierte Organismen importieren
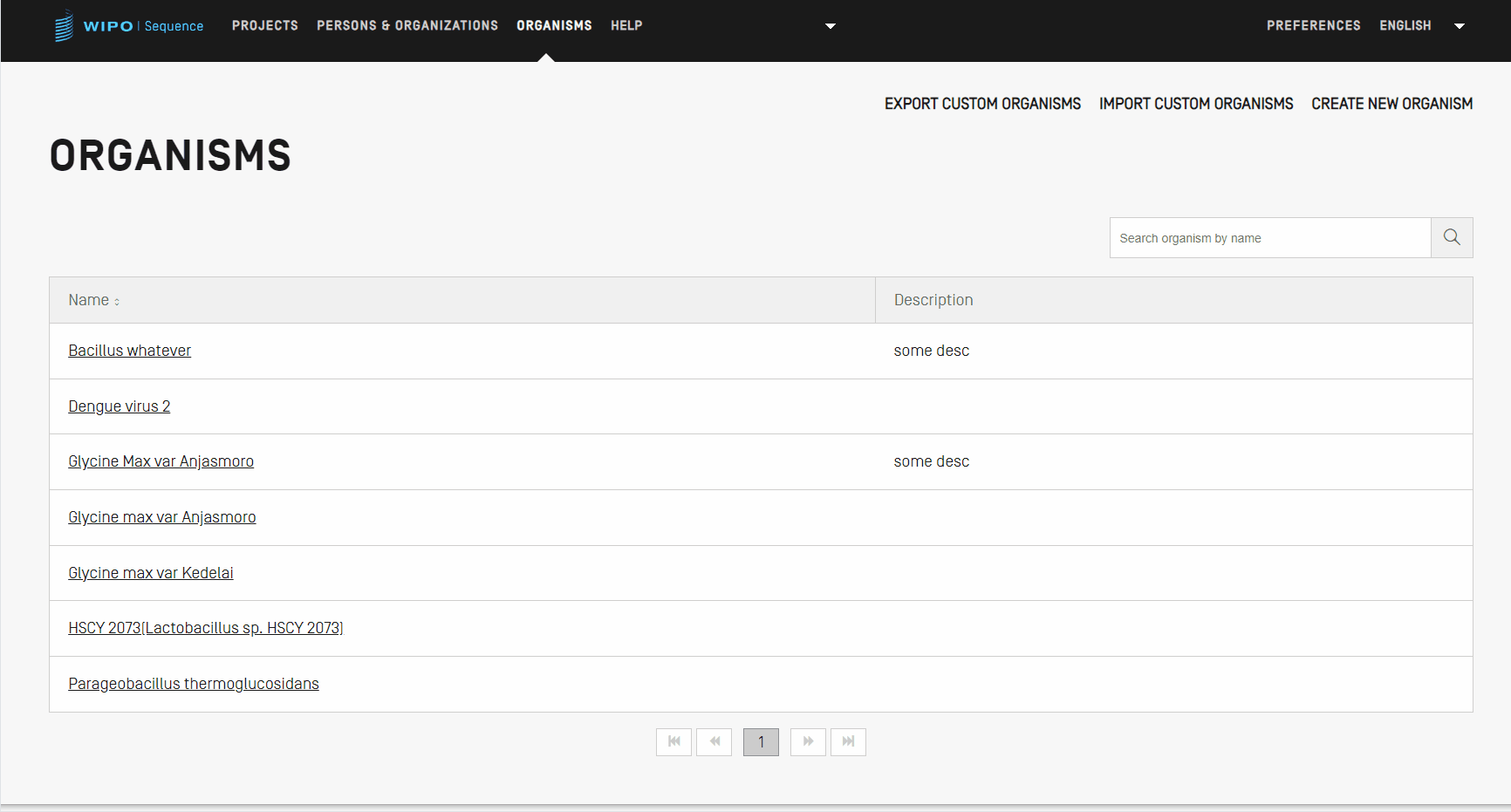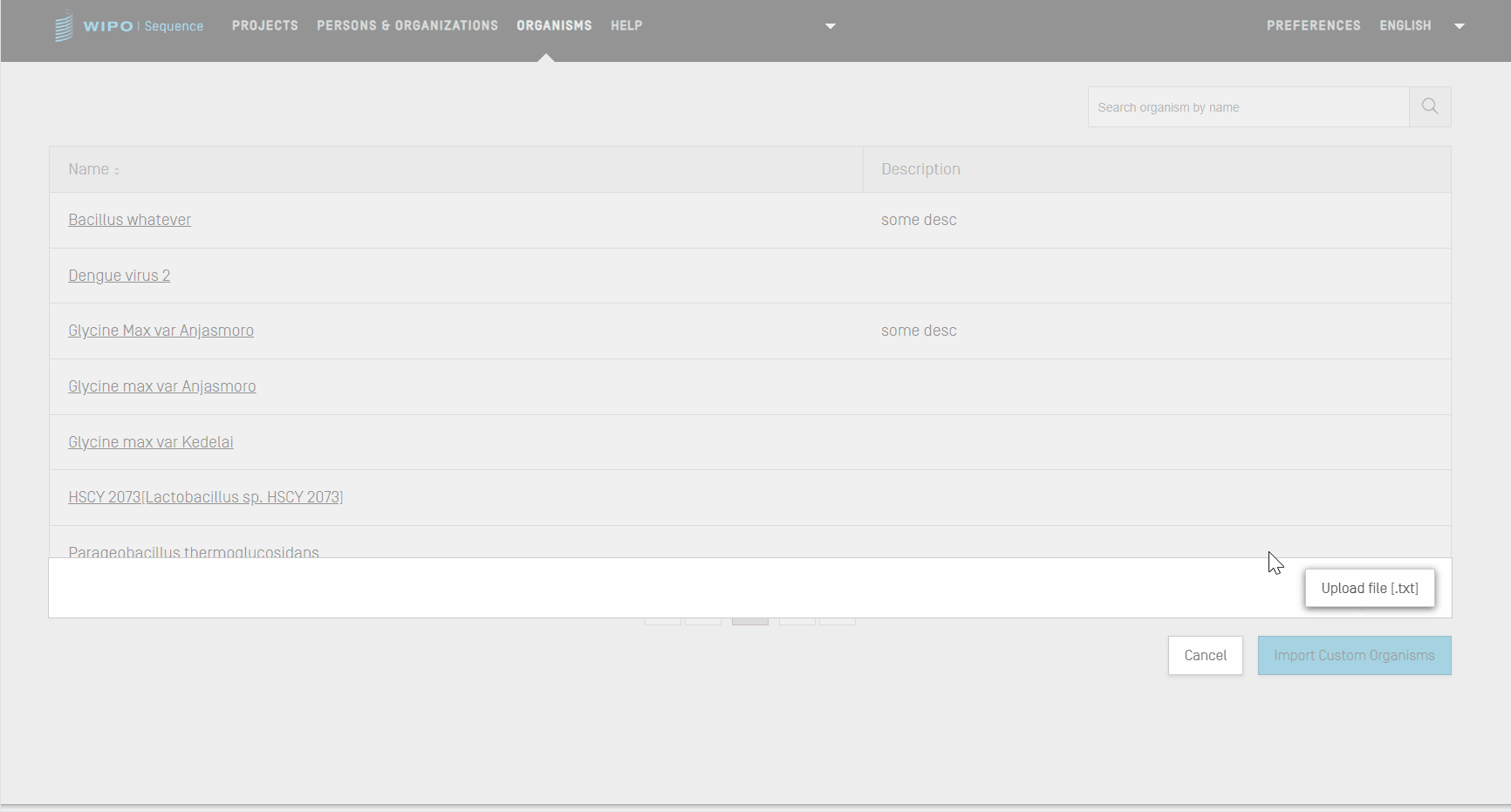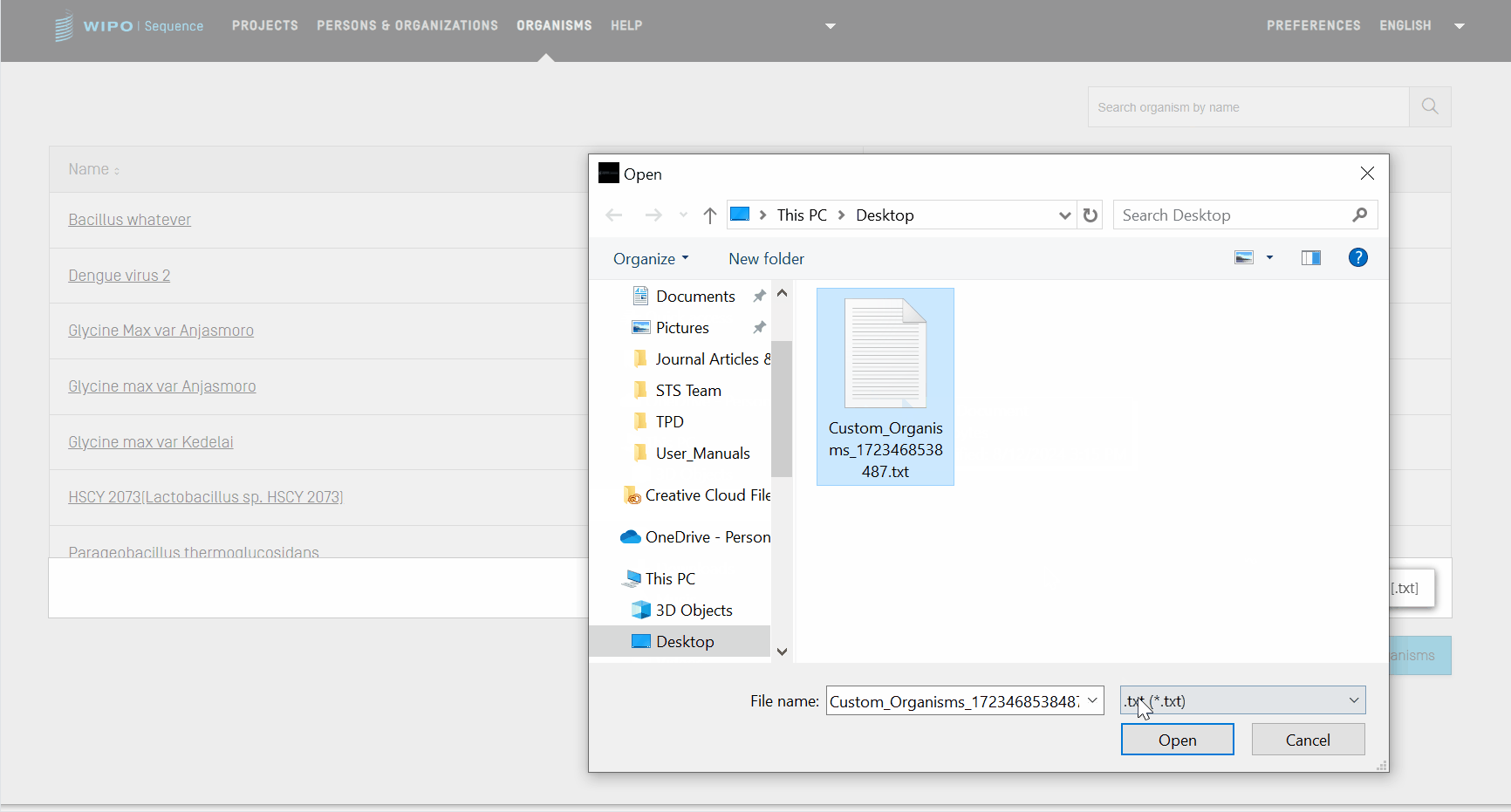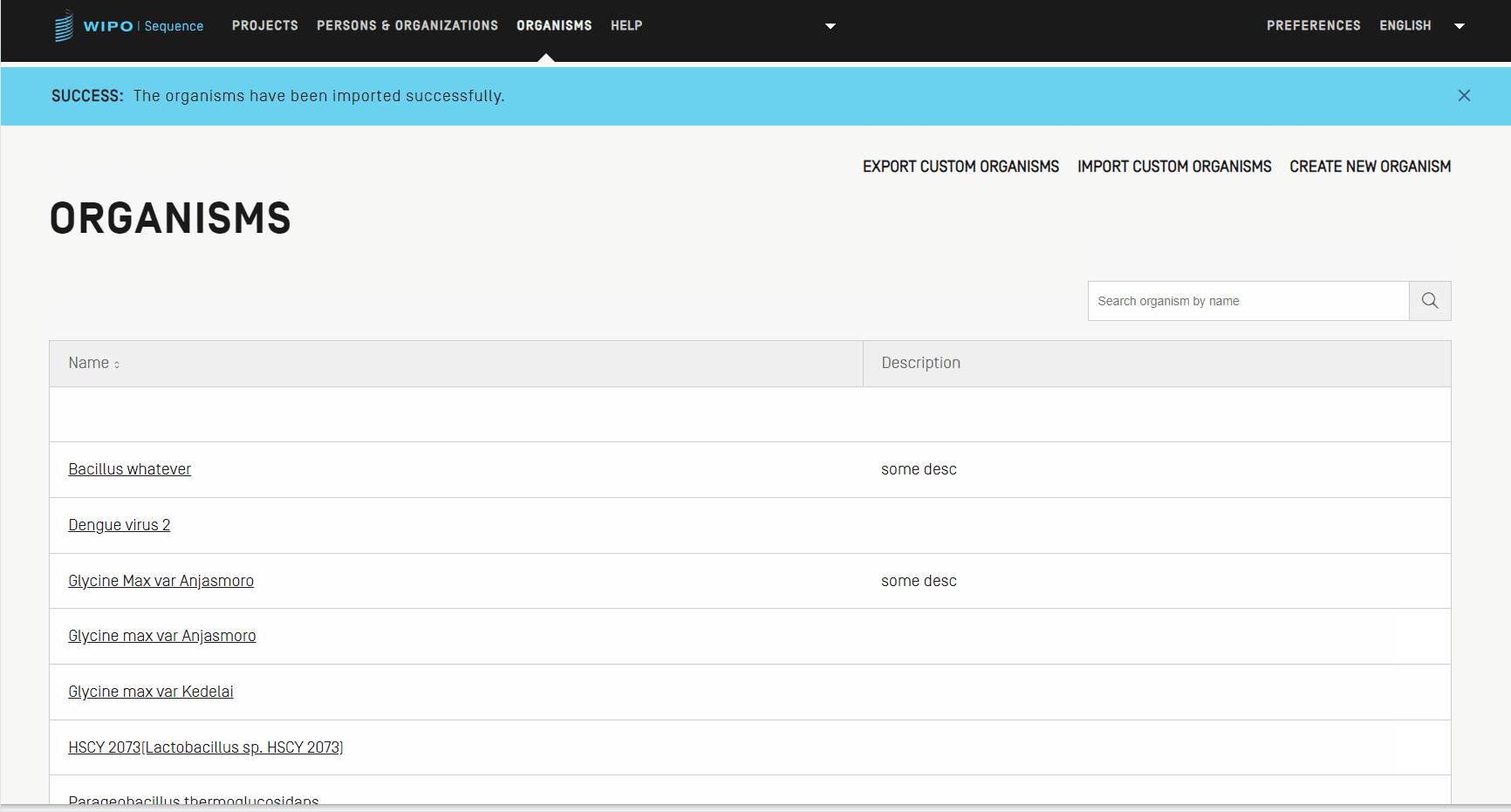
Zum Importieren einer Liste von benutzerdefinierten Organismen müssen Sie zunächst auf den in der Ansicht oben befindlichen Link "IMPORT CUSTOM ORGANISMS" (Benutzerdefinierte Organismen importieren) klicken. Dadurch wird ein Overlay unterhalb der Tabelle der Organismen geöffnet.
- Klicken Sie auf die Schaltfläche "Upload file [.txt]" (Datei [.txt] hochladen).
- Wählen Sie die Datei mit den benutzerdefinierten Organismusnamen aus dem Dialogfeld aus.
- Klicken Sie abschließend auf die blaue Schaltfläche "Import Custom Organisms" (Benutzerdefinierte Organismen importieren).
Hinweis: Die zu importierende Datei ist eine Textdatei (*.txt) mit einer Liste von benutzerdefinierten Organismusnamen in Klartext (UTF-8), wobei jedes Element in einer neuen Zeile steht.
2.5 Ansicht Einstellungen
Die Ansicht Einstellungen ermöglicht die Änderung mehrerer Konfigurationsparameter von WIPO Sequence. Diese Parameter gelten für jedes vom Tool erstellte oder bearbeitete Projekt.
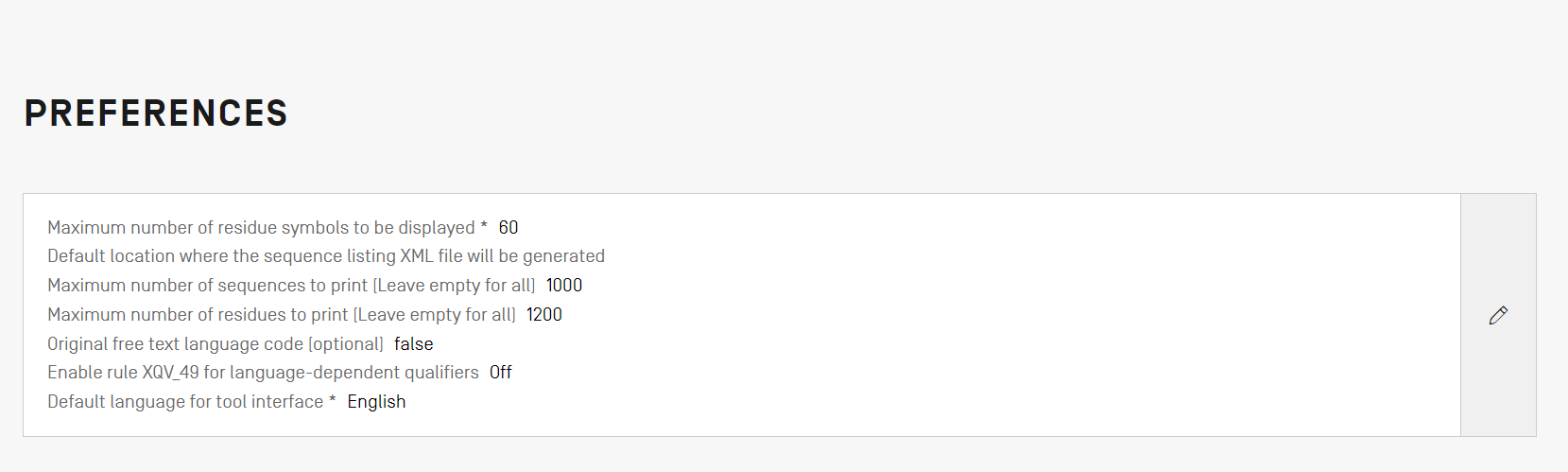
Zum Ändern der Einstellungen klicken Sie auf das oben gezeigte Stiftsymbol, um das Bearbeitungsbedienfeld zu öffnen.
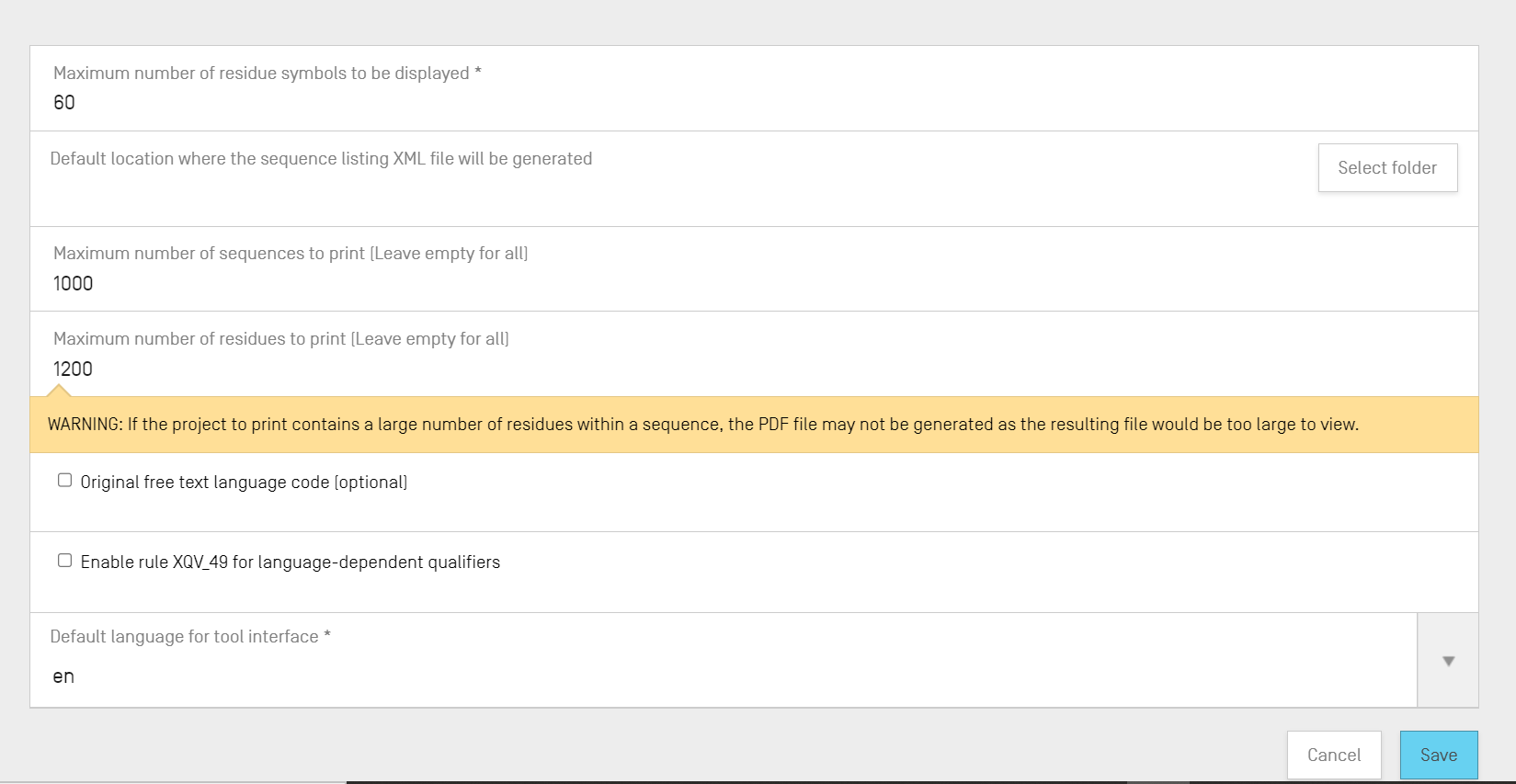
Die Liste der Konfigurationselemente, die in dieser Ansicht (in der Reihenfolge) geändert werden können, umfasst:
- Maximale Anzahl von anzuzeigenden Rest-Symbolen: Dieser Parameter legt die Anzahl der Reste fest, die bei der Anzeige einer Sequenz pro Zeile angezeigt werden: Die Standardeinstellung ist 60.
- Standardort, wo die XML-Datei des Sequenzprotokolls erstellt wird Diese Angabe ist nicht obligatorisch.
- Maximale Anzahl der zu druckenden Sequenzen (alle, wenn Sie das Feld leer lassen): die Standardeinstellung ist 1000.
- Maximale Anzahl der zu druckenden Reste (alle, wenn Sie das Feld leer lassen): die Standardeinstellung ist 1200.
- Originaler Freitext-Sprachcode (optional): Wenn dieses Kontrollkästchen aktiviert ist, wird bei der Validierung eine Warnung ausgegeben, wenn der originale Freitext-Sprachcode nicht angegeben ist. Diese Option ist standardmäßig deaktiviert.
- Regel XQV_49 für sprachabhängige Qualifier aktivieren: Wenn dieses Kontrollkästchen aktiviert ist, wird eine Warnung angezeigt, wenn kein englischer Wert für einen sprachabhängigen Freitext-Qualifier angegeben ist. Standardmäßig ist diese Funktion deaktiviert.
- Standard-Schnittstellensprache: Dies ist die Sprache, in der die Benutzeroberfläche beim Starten von WIPO Sequence angezeigt wird. Standardmäßig ist dies Englisch.
Hinweis: Der dritte und vierte Parameter sind relevant, wenn das Projekt als PDF gedruckt wird. Bei sehr umfangreichen Sequenzprotokollen kann das resultierende PDF mehrere tausend Seiten umfassen und nicht angezeigt werden.
3. Dateiformat
WIPO-Standard ST.25
Weitere Informationen zum Format der WIPO Standard ST.25-Dateien finden Sie unter Standard.
Ein Beispiel ist in Anhang III des Standards enthalten.
RAW (Rohformat)
Dieses Format kann nur eine Sequenz beschreiben. Der genetische Code ist in seiner Grundform ohne zusätzliche Informationen geschrieben. Beim Import müssen Molekülart, Merkmale und Name mit Hilfe des Tools zur Sequenz hinzugefügt werden.
Beispiel:
aggatatagatagtatatgatagtatgatatgatgatgtatgtatagtgtagttatga
Multi-Sequenz
Das Multi-Sequenz-Format kann eine oder mehrere Sequenzen beschreiben, zusammen mit ihrem Namen, der Molekülart und dem Namen des Organismus. Es ist eines der zulässigen Formate für den Import mit "PatentIn". Die erste Zeile mit nicht-leerem Text ist die Kopfzeile und besteht aus den folgenden Komponenten:
<SequenceName; SequenceType; OrganismName>
Die Informationen in der Kopfzeile werden von WIPO Sequence wie folgt interpretiert:
| Header entry (Eintrag in Kopfzeile) | Allowable Input (Zulässige Eingabe) | Interpreted as (Interpretiert als) |
|---|---|---|
| Sequenzname | Name der Sequenz (Freitext) | Name der Sequenz in der WIPO-Projektdatei (wird nicht Teil der XML-Datei sein) |
| Sequenzart | Eine der folgenden:
|
Beachten Sie, dass, je nach Eingabe des Organismus, der mol_type für DNA und RNA näher definiert werden muss (ST.26, Absätze 75-84) |
| Name des Organismus | Name des Organismus (Freitext) | "Source/organism note"-Hinweis: Handelt es sich bei der Eingabe um ein synthetisches Konstrukt, wird der "mol_type" für DNA und RNA automatisch als "andere DNA" oder "andere RNA" identifiziert (ST.26, Absatz 84 Buchstabe (a)). |
Die Sequenzdaten beginnen in der Zeile nach der Kopfzeile. Eine neue Sequenz wird durch eine neue Zeile in der Datei, nach dem Ende der Sequenzinformation der vorherigen Sequenz, beschrieben. Zwischen dem Ende einer Sequenz und dem Beginn der nächsten Kopfzeile können eine oder mehrere Leerzeilen stehen. Aminosäuresequenzen müssen in einem Buchstabencode angegeben werden (Anhang I, Abschnitt 3, Tabelle 3). Zulässige Nukleotidsymbole sind in Anhang I, Abschnitt 1, Tabelle 1 aufgeführt. Beachten Sie, dass "u" in RNA-Sequenzen nicht automatisch in "t" umgewandelt wird, wie in ST.26 Absätze 14 und 19 gefordert, und nach dem Import manuell eingegeben werden muss. Es wird empfohlen, "u" in "t" umzuwandeln, bevor Sie eine Datei mit RNA-Sequenzen im Multi-Sequence-Format importieren.
Im Folgenden ein Beispiel für einen Satz von drei Sequenzen, die im Multi-Sequence-Format definiert sind.
Beispiel:
<First Sequence; RNA; Albies alba> uuuucuuauuguuucuccuacugcuuaucauaaugauugucguaguggcuuccucaucgucucccccaccgccuaccacaacgacugccgcagcggauuacuaauaguaucaccaacagcauaacaaaaagaaugacgaagaggguugcugauggugucgccgacggcguagcagaaggaguggcggagggg
<Second Sequence; DNA; synthetic construct> attgacgtcagtgacgcggtactgacgtcagctgcagtactgacgtaccaaccacgtggtgagctctcgacatgcaactgactcgtcgctattgacgtcagtgacgcggtactgacgtcagctgcagtactgacgtaccaaccacgtggtgagctctcgacatgcaactgactcgtcgctcagt
<Third Sequence; AA; Mus musculus>
SPPGKPQGPPPQGGNQPQGPPPPPGKPQGPPPQGGNRPQGPPPPGKPQGPPPQGDKSRSPR
FASTA
Dieses Format enthält sowohl Reste also auch Beschreibung. Beim Importieren haben Sie die Möglichkeit, die Beschreibung als ein "Note"-Qualifier (Hinweis-Qualifier) zu speichern.
Beispiel:
AJ011880.1 Artificial oligonucleotide sequence SSR primer (CAC13R)
CTCAACAATCTGAAGCATCG
4. Fehlerbehandlung
WIPO Sequence bietet eine Hilfe-Funktion, die über das Hauptmenü aufgerufen werden kann.
- Die verschiedenen Hilfeoptionen führen zu weiteren Informationen. Dort finden Sie:
- Einen Link zum Benutzerhandbuch
- Einen Link zur WIPO ST.26-Wissensdatenbank
- Einen Link zum Kontaktformular für das WIPO Sequence Support-Team
- Einen Link zur aktuellen Version von WIPO Standard ST.26
- Allgemeine Informationen zur Version des WIPO-Sequence-Desktop-Tools
Häufige Probleme und Workarounds
Der Importbericht oder der Validierungsbericht zeigt an, dass das Projekt mehrere Qualifier-IDs enthält.
Workaround: Löschen Sie das Merkmal, das eine doppelte Qualifier-ID enthält. Erstellen Sie das Merkmal an derselben Stelle neu, aber fügen Sie nicht den Qualifier vor dem Speichern hinzu. Bearbeiten Sie als Nächstes das Merkmal, um den entsprechenden Qualifier hinzuzufügen, und aktualisieren Sie das Merkmal.
Der vorgeschlagene Dateiname mit der Linux-Distribution ist falsch.
Es besteht ein bekanntes Problem bei der Verwendung der Linux-Distribution: In der Standardeinstellung wird zusätzlich "\" beim Vorschlag eines Dateinamens angezeigt. Um dieses Problem zu beheben, entfernen Sie bitte manuell das zusätzliche "\" vor dem Speichern.
Weitere Antworten finden Sie in der WIPO-Sequenz-Wissensdatenbank.
Beim Versuch, das Sequenzprotokoll im HTML-Format anzuzeigen, wird ein Fehler angezeigt.
Ist das generierte Sequenzprotokoll im XML-Format größer als 100 MB, wird anstelle des Sequenzprotokolls im HTML-Format ein Fehler angezeigt, der darauf hinweist, dass das Sequenzprotokoll zu groß ist, um im HTML-Format dargestellt zu werden.
Bei weiteren Fragen wenden Sie sich bitte an uns.