Manuel de l’utilisateur de WIPO Sequence
Le présent document a pour but d’expliquer aux utilisateurs comment utiliser les fonctions de base du logiciel WIPO Sequence. En général, les utilisateurs sont des déposants ou leurs représentants qui souhaitent présenter une demande de brevet comportant un listage des séquences.
Ce manuel d'utilisation concerne la version 3.0.0.
1. Résumé des fonctionnalités
Ce tableau résume toutes les fonctionnalités disponibles dans la version actuelle du logiciel, avec des liens vers les sections correspondantes :
2. Fonctionnalités du logiciel
2.1 Vue Project Home (accueil du projet)
On trouvera dans la présente section une description détaillée des différentes options disponibles dans Project Home (accueil du projet).
Un projet est la structure objet employée par le logiciel pour stocker les données nécessaires à la génération des listages des séquences. Le logiciel utilise les données stockées dans le projet, une fois que la conformité de ces données à la norme ST.26 de l’OMPI a été validée, pour alimenter les valeurs du listage des séquences généré.
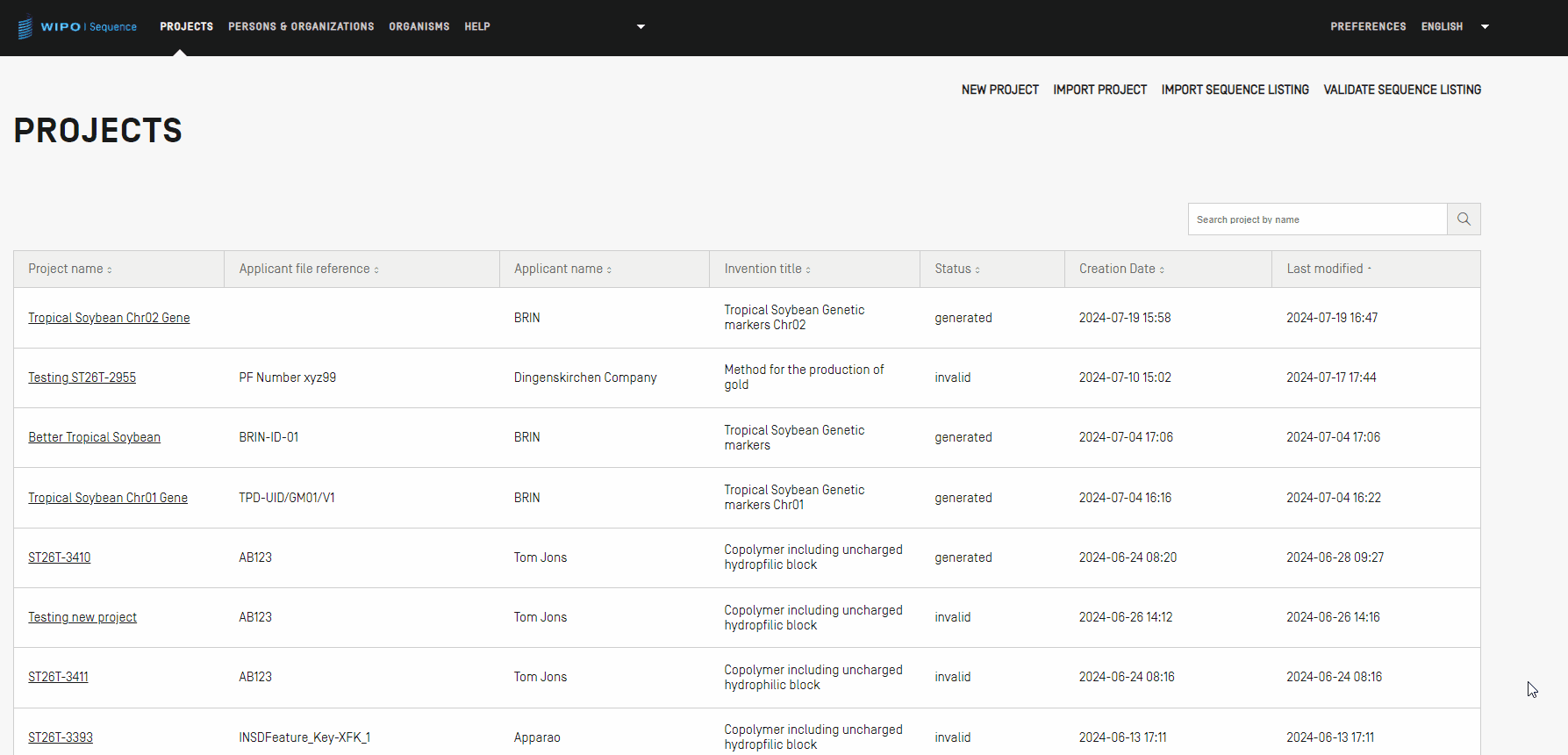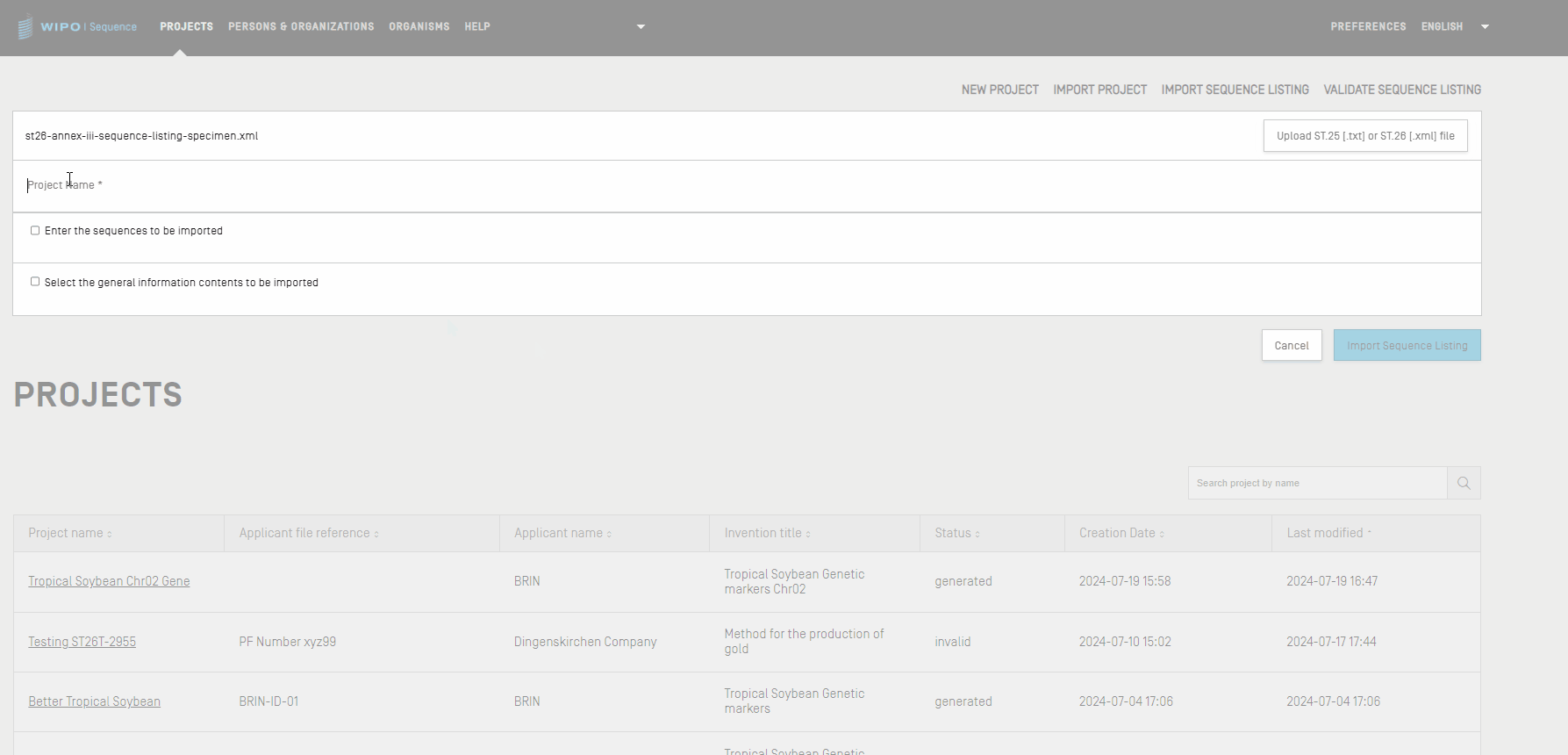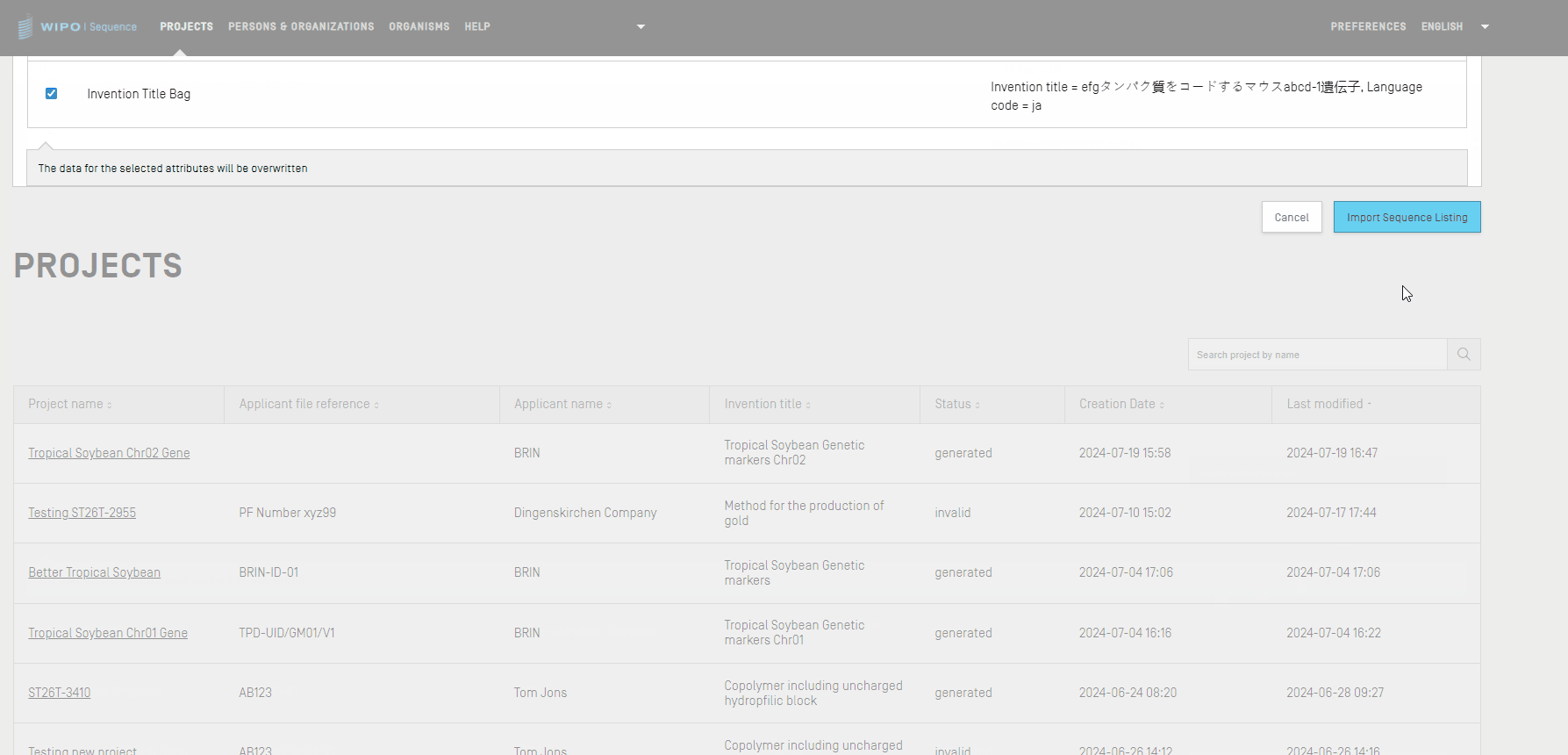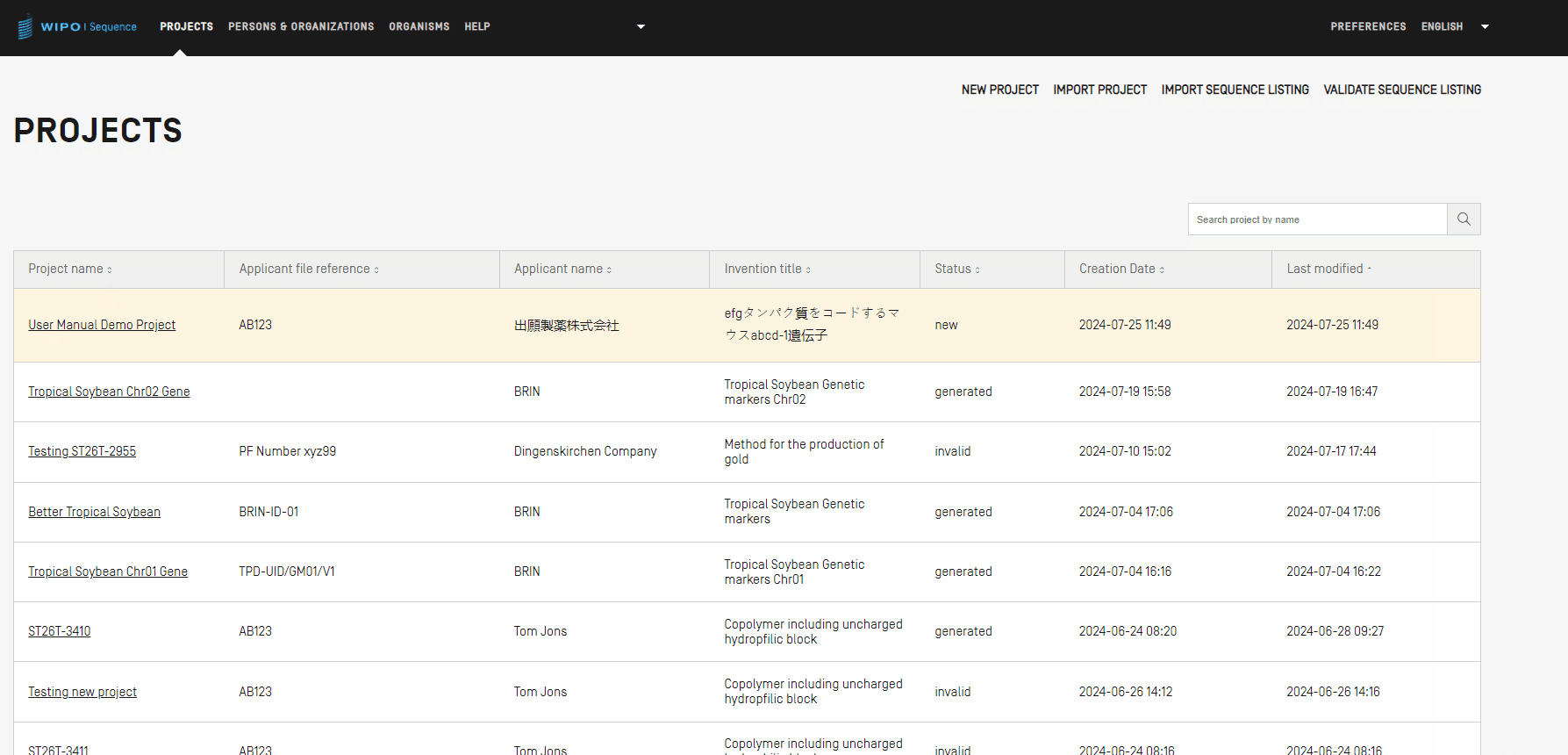
Dans cette vue, la liste des projets créés s’affiche, ce qui vous permet de trier ou d’utiliser la fonction de recherche pour filtrer par nom de projet, référence du dossier du déposant, nom du déposant, titre de l’invention, statut ou date de création.
Note : Le logiciel affiche un maximum de 1000 projets. Si un projet n’est pas affiché dans la vue de l’accueil du projet, vous devez utiliser la fonction de recherche pour identifier le projet par son nom, car il est toujours stocké localement, mais n’est pas visible sur cette page.
Créer un projet
Pour créer un nouveau projet, vous devez partir de la vue initiale de l’accueil du projet représenté ci-dessous.

1) Cliquez sur le lien “NEW PROJECT” (NOUVEAU PROJET) en haut de la page. Comme indiqué, le logiciel va demander un nom (obligatoire) et une description (facultative).

2) Lorsque le champ de nom est renseigné, le bouton “Save” (Sauvegarder) devient actif et vous permet d’enregistrer le nouveau projet. La liste des projets comprendra désormais ce nouveau projet sur la page de l'accueil du projet.
Importer un projet
Cette fonctionnalité permet d'importer dans le logiciel un projet précédemment exporté. Pour importer un fichier de projet, vous devez commencer par afficher la vue de l'accueil du projet.
Cliquez sur le lien “IMPORT PROJECT (IMPORTER UN PROJET) en haut de la page comme indiqué, puis suivez les étapes de la vidéo ci-dessous :
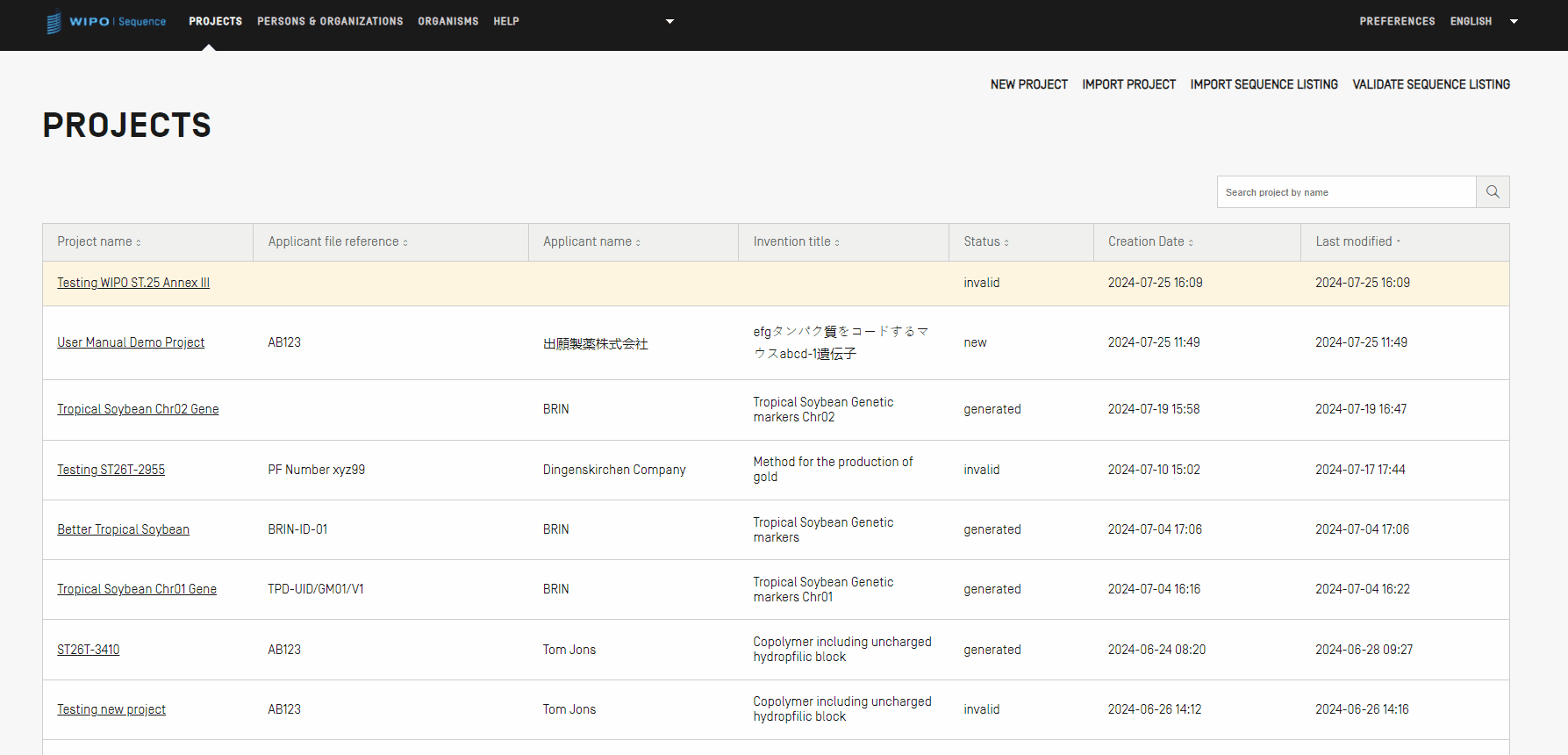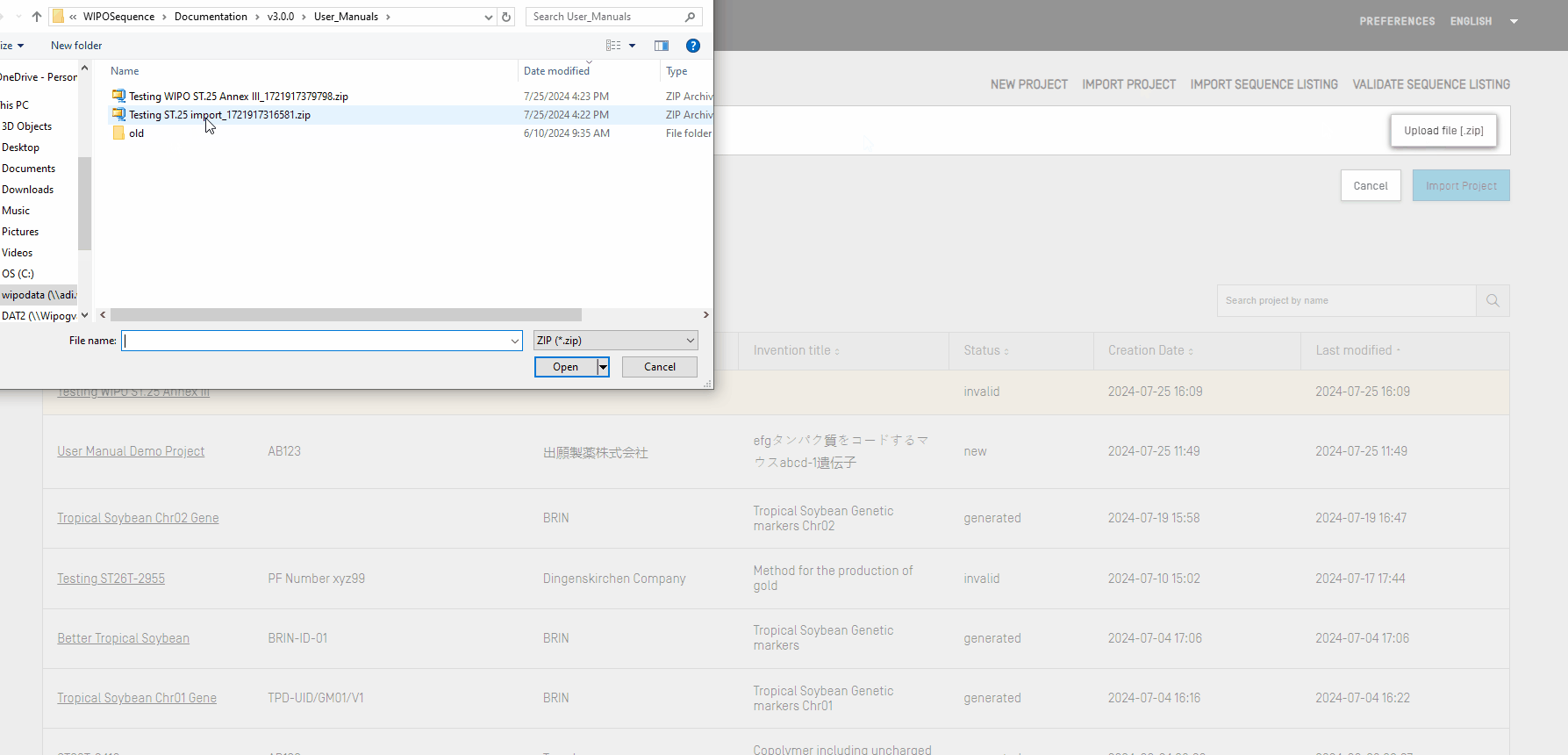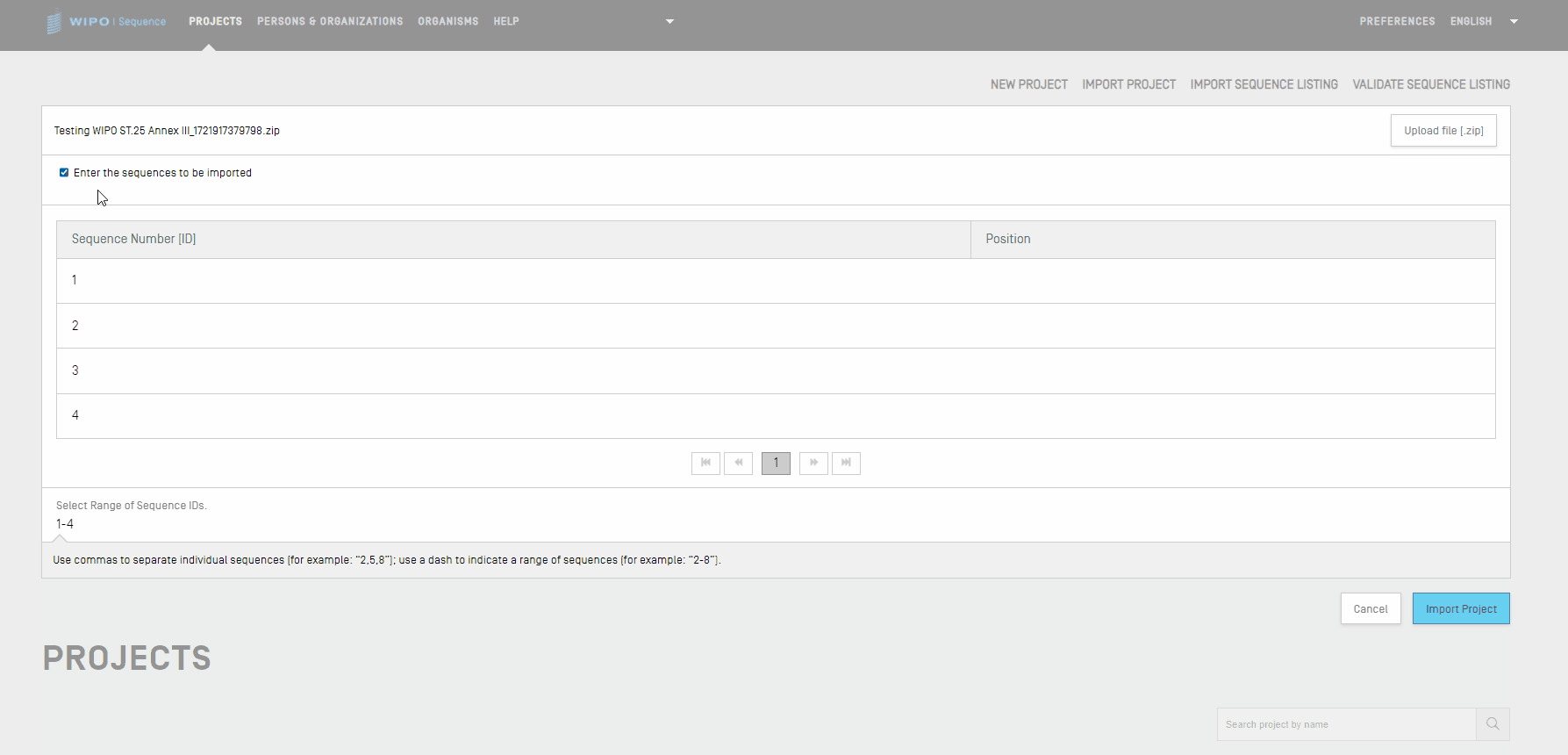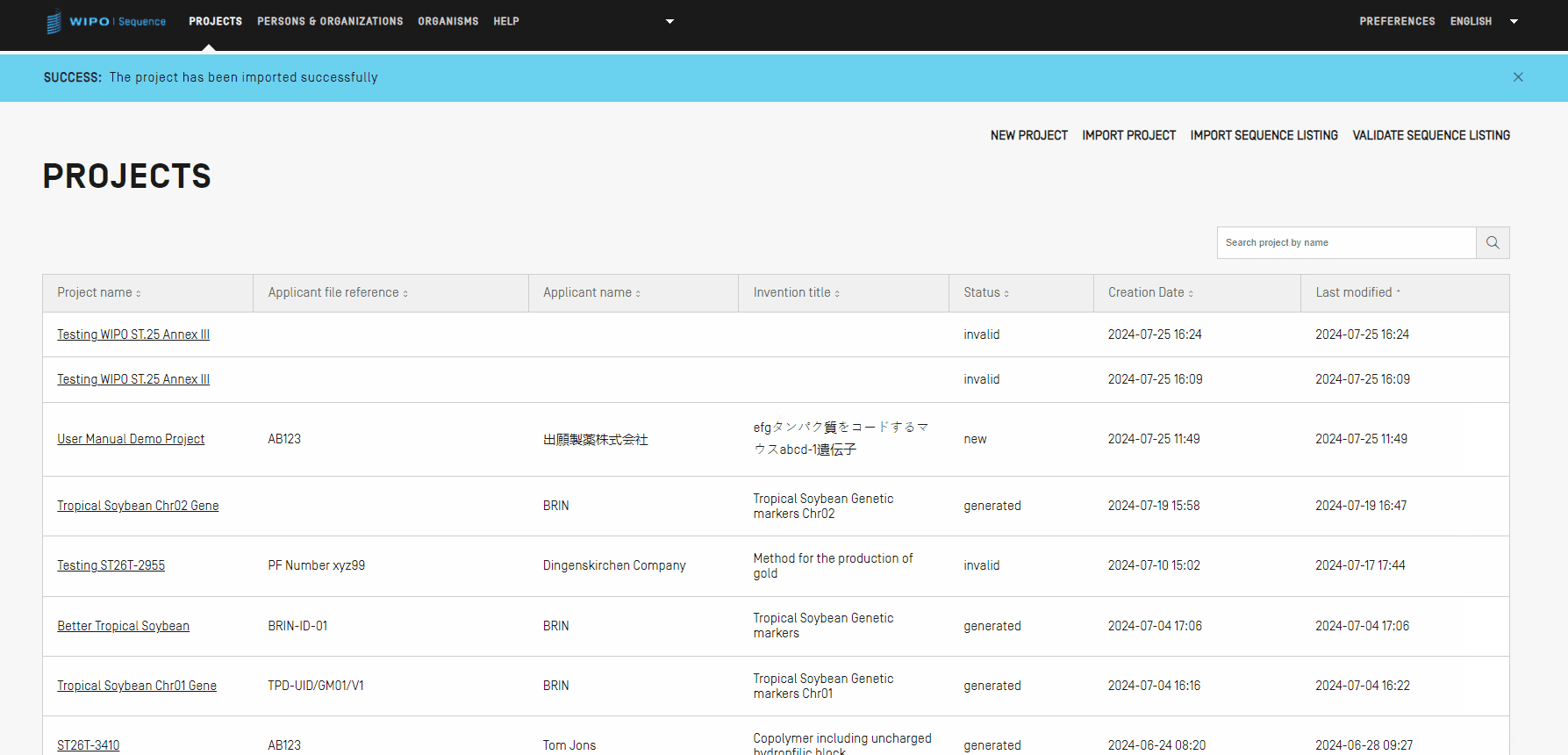
Si la case “Select Range Sequences” (Choisir une fourchette de séquences) reste non cochée, toutes les séquences seront importées. Si vous souhaitez choisir des séquences particulières à importer dans le projet, vous devez cocher la case “Select Range Sequences” (Choisir une fourchette de séquences) et saisir le numéro d’identifiant (ID) des séquences souhaitées dans le champ pertinent. Vous pouvez saisir une seule séquence, ou un listage de séquences en les séparant par une virgule, ou encore une fourchette de séquences sous la forme x-y. Par défaut, le nombre total de séquences du projet importé sera affiché sous forme de fourchette, c'est-à-dire 1-total des séquences.
Exemple : “1, 3, 7, 13-20, 30-50”
Si l'importation du projet réussit, le bandeau bleu et le message illustrés ci-dessous apparaissent en haut de la vue.
Note : Vous devez vérifier que le projet zip importé correspond à la base de données actuelle. Si le projet a été exporté dans une version antérieure à la version 3.0.0, il ne fonctionnera pas correctement; le projet importé doit utiliser la même base de données. Ceci est dû à la nouvelle mise en œuvre de la base de données à partir de la version 3.0.0.
Importer un listage des séquences
À partir de la vue Project Home (accueil du projet),vous ne pouvez importer que des informations issues d’un listage des séquences conforme à la norme ST.26 ou ST.25. Les formats de chacun de ces fichiers doivent être *.xml pour les fichiers ST.26 et *.txt pour les fichiers ST.25. Pour plus de détails sur les étapes à suivre, regardez la vidéo ci-dessous.
Note : Lors de l'importation d'un listage des séquences, les fonctionnalités et les qualificateurs sont sensibles à la casse et doivent être conformes aux valeurs indiquées à l’annexe I de la norme ST.26 de l’OMPI.
Il importe également de noter que les listages des séquences conformes à la norme ST.25 de l’OMPI doivent être valides, faute de quoi la fonctionnalité du logiciel WIPO Sequence ne peut être garantie durant l'importation.
Le tableau du rapport d’importation n’est affiché que lorsqu'une erreur survient pendant l'importation d’un fichier et il contient alors les colonnes suivantes :
- "Type de note" : "INDIVIDUAL" (INDIVIDUELLE) pour un message concernant une séquence particulière, ou "GLOBAL" (GLOBALE) pour un message concernant une ou plusieurs séquences de manière générale
- “Data element code” (Code d’élément de donnée) : valeur reprise du fichier source aux fins des listages des séquences ST.25
- “Message text” (Texte du message) : message détaillé contenant des informations sur le problème détecté et sur les modifications à apporter pour le régler (le cas échéant)
- “Detected sequence” (Séquence détectée) : numéro de la séquence importée liée au message (lorsque le type est “INDIVIDUAL”, sinon ce champ reste vide).

Si le fichier était au format ST.25, la vue “Import Review” (Rapport d’importation) contient tout d’abord un rapport d’importation, puis le rapport sur les données modifiées. Ce rapport sur les données modifiées affiche toutes les données ayant subi des transformations ou des modifications au cours du processus d’importation. Les données suivantes sont présentées dans un tableau récapitulatif :
- “Origin Tag” (Balise source) : code de l’élément de donnée correspondant au type d’élément lors de l'importation de listages des séquences ST.25
- “Origin Element Name” (Nom de l’élément source) : nom correspondant au type d’élément
- “Origin Element Value” (Valeur de l’élément source) : valeur correspondant à l’élément original dans le fichier source
- “Target Element Name” (Nom de l’élément cible) : nom de l’élément équivalent dans le format ST.26 si l’information va être stockée dans le projet
- “Target Element Value” (Valeur de l’élément cible) : valeur qui sera attribuée au nom de l’élément cible dans le projet
- “Transformation” : description des modifications ou des transformations subies par l’élément
- “Sequence ID Number” (Numéro ID de la séquence) : numéro d’identifiant de la séquence correspondant à l’élément transformé dans le projet

À ce stade, vous pouvez revenir à la vue Project HOme (accueil du projet) ou imprimer un rapport sur ces modifications au format PDF. Pour savoir comment télécharger/imprimer le fichier PDF, reportez-vous à la section Display sequence listing (Afficher le listage des séquences).
Inversement, le processus d’importation peut échouer si le fichier de listage des séquences contient des erreurs. Dans ce cas, après avoir tenté une importation, vous serez averti par un bandeau rouge indiquant qu’une erreur est survenue pendant l’importation

Note : En outre, le logiciel fonctionne mieux lorsque l’on respecte le seuil limite de 100 000 séquences. Lorsque vous vous trouvez en présence de listages des séquences volumineux, vous pouvez utiliser la solution suivante : décomposer le processus d’importation en une série d’étapes, en choisissant une fourchette précise de séquences à importer, puis en important ces séquences dans un projet fourchette par fourchette. Par exemple, un listage des séquences d’environ 100 000 séquences peut être divisé en une série de 10 x 10 000 séquences et ces séries peuvent ensuite être importées une par une. Les 10 000 premières séquences seront utilisées lors de la création du projet.
Valider le listage des séquences
Vous pouvez valider un fichier de listage des séquences ST.26 en cliquant sur le bouton “VALIDATE SEQUENCE LISTING” (VALIDER LE LISTAGE DES SÉQUENCES) en haut et à droite de la vue Projects (Projets).

Si le listage des séquences est validé, un bandeau s’affiche comme indiqué.

Si la validation échoue, un rapport de vérification s’ouvre dans votre navigateur et la liste des erreurs de validation est présentée dans un tableau, comme indiqué dans l'exemple. L’emplacement du fichier HTML s’affiche à côté du rapport de vérification XML, au cas où vous souhaiteriez copier les fichiers dans un autre emplacement.
Si vous utilisez un navigateur IE, pour que le format se charge correctement, vous devez autoriser l'exécution d'un script interne sur votre ordinateur. Dans le cas contraire, les séquences ne s’afficheront pas dans le format standard et seront moins lisibles.
Veuillez noter que pour que le listage des séquences puisse être validé, le fichier ST.26 doit être conforme aux critères suivants :
-
Le fichier doit être codé en UTF-8 et doit contenir des caractères conformes à la spécification XML 1.0
-
Le fichier doit contenir la ligne DOCTYPE suivante :
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing 1.3//EN" “ST26SequenceListing_V1_3.dtd”>
Le fichier doit être conforme au fichier DTD intitulé ST26SequenceListing_V1_3.dtd.
Supprimer un projet
Pour supprimer un projet, vous devez commencer par afficher la vue Project Home (accueil du projet).
Cliquez sur l'icône représentant une poubelle dans la ligne du tableau de l'accueil du projet que vous souhaitez supprimer.
Dans la boîte de dialogue qui apparaît, cliquez sur le bouton “Delete” (Supprimer) pour confirmer la suppression du projet choisi.

2.2 Vue détaillée du projet
La vue détaillée du projet contient toutes les informations relatives à une seule demande de brevet ou à un seul listage des séquences. Elle est divisée en deux sections : les informations générales et les données relatives aux séquences. En haut de la page se trouve un tableau contenant les informations de base sur le projet, notamment les éléments suivants :
- Nom du projet
- Date et heure de création du projet
- Date et heure des dernières mises à jour apportées au projet
- Statut du projet (valeurs possibles : “new”/“modified”/“generated”/“invalid”/“valid”/“warnings”) - ce n'est pas un champ modifiable!
- Description du projet – facultative
- Nom du fichier importé (si le projet a été importé)
- Code langue du texte libre original
- Nombre de séquences (intitulé "Sequences")
- Case à cocher pour demander l’ajout automatique d’un qualificateur de traduction en cas de création d’une caractéristique CDS (une fonction au niveau du projet)
- Langue du texte libre autre que l'anglais.

Il y a deux niveaux de menus, le premier lié aux données de la vue détaillée du projet (en jaune) et le second un menu de navigation vers d'autres vues liées au projet (en bleu). Vous pouvez quitter le projet en cliquant sur “Return to project home” (Retour à l'accueil du projet).
Pour le menu de navigation, il y a six vues différentes qui sont accessibles à partir de la page détaillée du projet :
- La vue détaillée du projet (actuelle), affichée avec le nom du projet
- La vue “Verification Report” (Rapport de vérification), endroit où il est possible d’accéder au rapport de vérification
- La vue “Language dependent Qualifiers” (Qualificateurs dépendants de la langue) (3), endroit où il est possible d’accéder aux qualificateurs de texte libre dépendants de la langue et de les importer ou les exporter
- La vue “Import Report” (Rapport d’importation), endroit où il est possible d’accéder au rapport d’importation
- La vue “Display Sequence Listing” (Affichage du listage des séquences), qui permet d'accéder à des formats lisibles par une personne pour les 26 listages des séquences générés.
- Le menu d'aide, qui comprend des renvois au Manuel de l'utilisateur, à WIPO Sequence et à la base de connaissances de la norme ST.26.
- La vue “Preferences” (Préférences), qui concerne tous les projets dans cette version de WIPO Sequence.
Imprimer le projet
Pour imprimer un projet, vous devez afficher la vue détaillée du projet souhaité et cliquer sur le bouton “Print” (Imprimer) en haut de la vue.
Deux cases à cocher vont alors s'afficher pour préciser les informations du projet que vous souhaitez imprimer, à savoir la partie consacrée aux informations et/ou aux séquences.
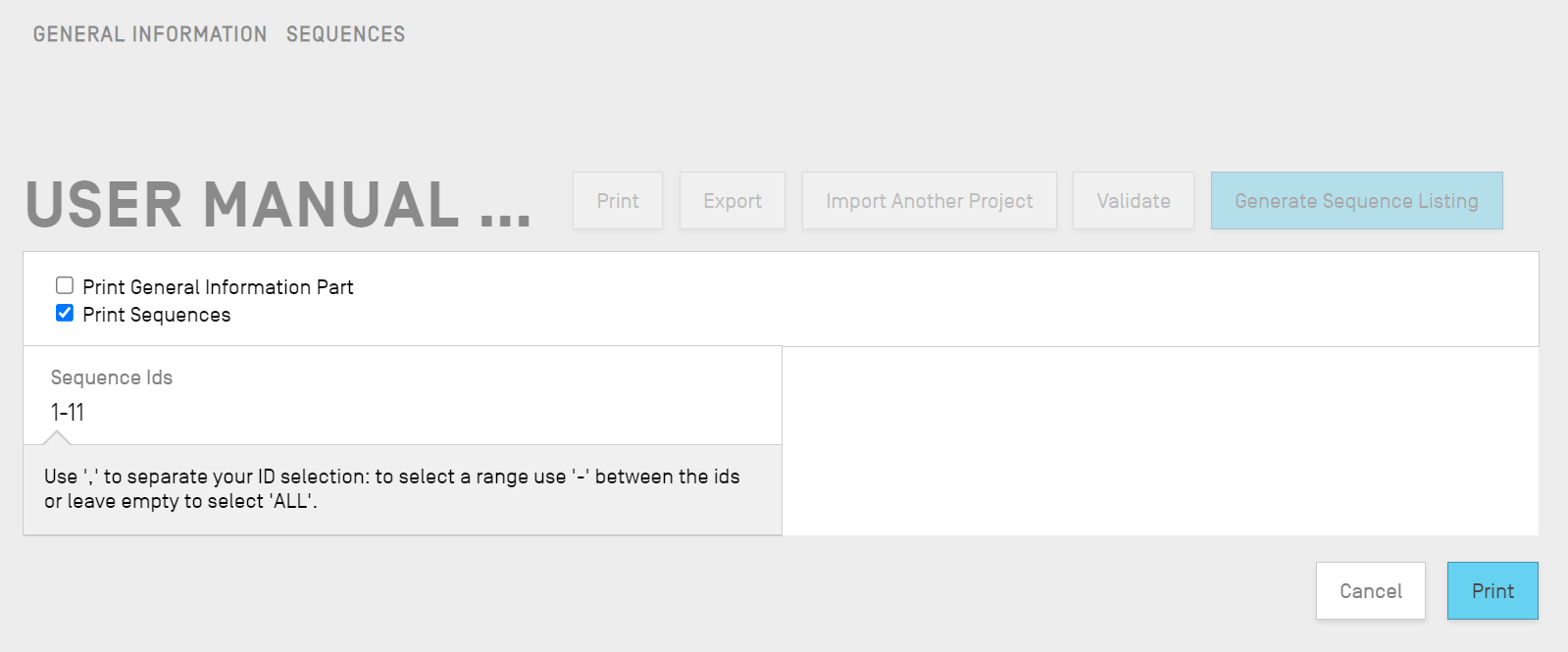
Si vous choisissez la case “Print Sequences” (Imprimer les séquences), vous devez préciser quelles séquences vous souhaitez imprimer en indiquant la fourchette des numéros d’identifiant dans le champ “Sequence IDs” (ID des séquences), ou vous pouvez simplement imprimer toutes les séquences en laissant ce champ vide.
Par défaut, le nombre total de séquences du projet s’affiche sous la forme d’une fourchette.
Exporter le projet
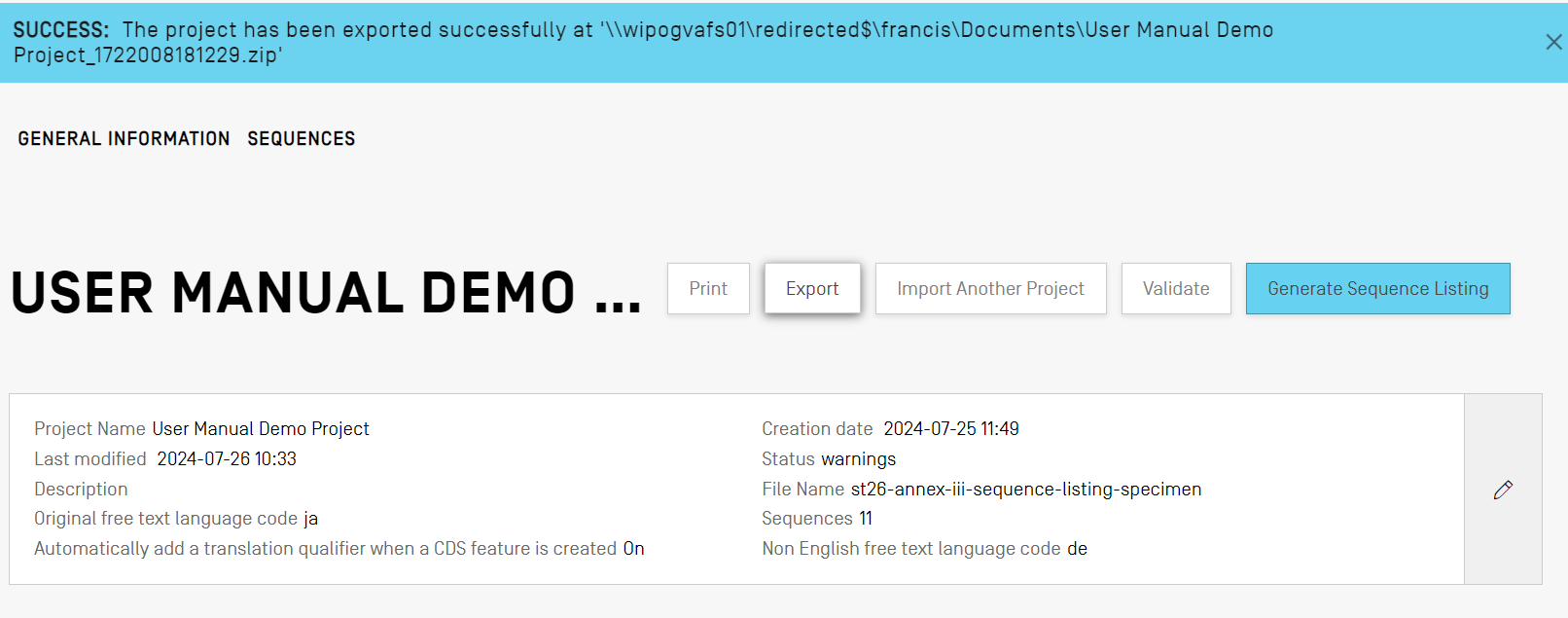
Un projet peut être exporté dans un fichier .zip pour sauvegarder les données du projet ou importer avec un autre ordinateur de bureau sur lequel le logiciel WIPO Sequence est installé. Cliquez simplement sur le bouton “Export” (Exporter) et sélectionnez un emplacement pour enregistrer le fichier .zip Si l'exportation est réussie, vous verrez apparaître le bandeau bleu suivant :
Importer un autre projet dans le projet actuel
Vous pouvez copier dans le projet actuellement ouvert des informations provenant d’autres projets stockés dans le logiciel. Les informations importées peuvent concerner la section “General Information” (Informations générales), la section “Sequences” (Séquences) ou les deux. Les informations générales importées vont remplacer les informations générales déjà présentes dans le projet en cours, tandis que les séquences importées seront ajoutées à la suite du listage des séquences existant du projet.

Vous devez d’abord choisir le projet dont vous souhaitez importer les informations dans le menu déroulant indiqué. Vous pouvez indiquer si vous souhaitez importer certains éléments particuliers des informations générales et également si vous souhaitez importer des séquences en indiquant une fourchette de numéros d’ID de séquences pour préciser quelles séquences doivent être importées dans le projet. Par défaut, le nombre total de séquences du projet s’affiche sous la forme d’une fourchette.
Si la case “General Information” (Informations générales) est cochée, un tableau apparaît et présente toutes les informations générales des deux projets : le projet actuellement choisi (destination) et le projet cible (alternative). Vous devez ensuite indiquer quels éléments des informations générales doivent être remplacés par les informations générales correspondantes du projet importé.
Enfin, une fois que vous avez défini les éléments des informations générales et les séquences à importer dans le projet, vous devez cliquer sur le bouton bleu “Import Project” (Importer un projet). Un bandeau bleu apparaît si les éléments ont été importés correctement.
Valider le projet
Avant de générer le listage des séquences sous forme de fichier XML conforme à la norme ST.26, un projet fera au préalable l’objet d’un processus de validation. Cette étape est toujours effectuée avant la génération du listage des séquences, mais elle peut aussi être effectuée de manière indépendante.
Pour valider un projet, vous devez cliquer sur le bouton “Validate” (Valider) en haut de la vue détaillée du projet.
Une fois la validation achevée, le logiciel présente la vue “Verification Report” (Rapport de vérification), dans laquelle se trouvent tous les messages d’avertissement et d’erreur relatifs à la vérification qui ont pu être générés. En cas de succès de la validation, un bandeau bleu s'affiche.
Si le processus de validation produit des erreurs ou des avertissements, le rapport de vérification généré contient un tableau indiquant les règles et directives de vérification qui n’ont pas été respectées. Chaque ligne du tableau indique s’il s’agit d’une erreur, qui doit alors être corrigée, ou s’il s’agit d’un avertissement qui peut être ignoré.
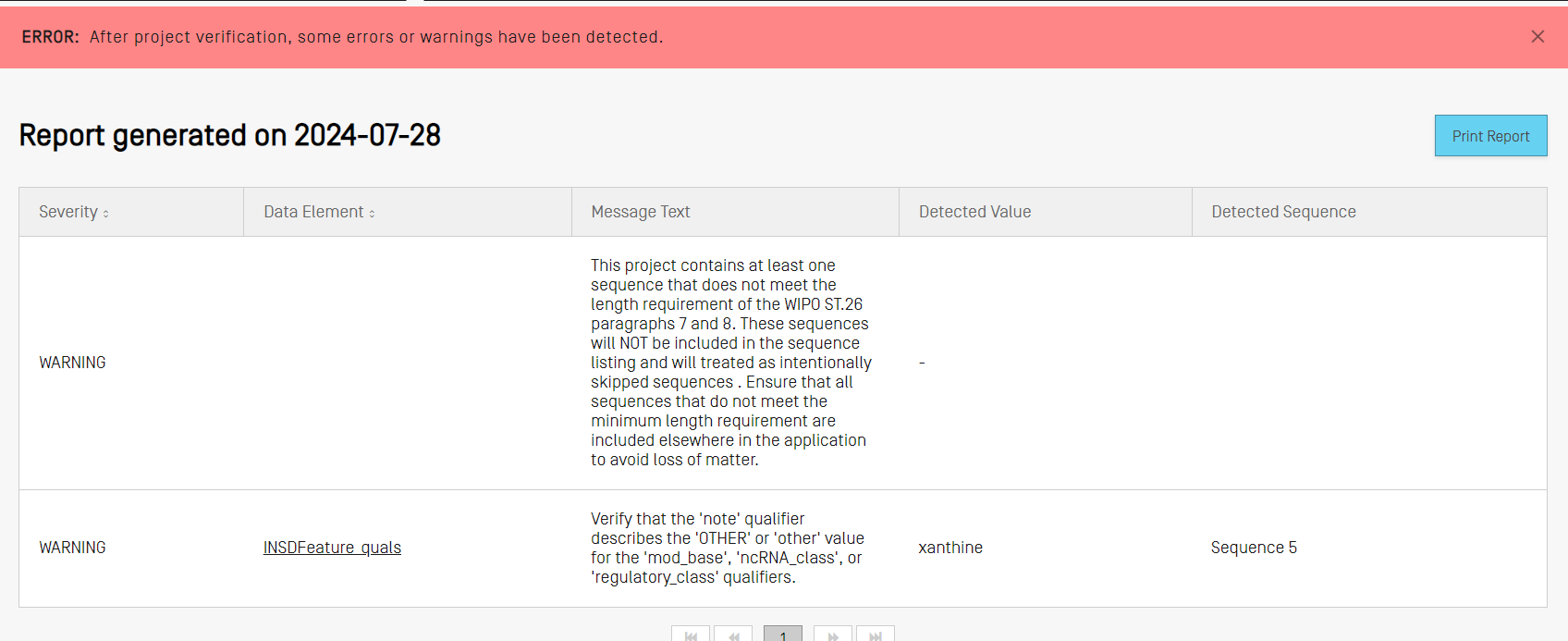
Générer un listage des séquences
La dernière fonction qui puisse être appliquée à un projet, et qui est peut-être la plus importante, consiste à générer le listage des séquences. À cette fin, vous devez cliquer sur le bouton bleu “Generate Sequence Listing” (Générer un listage des séquences) en haut de la vue détaillée du projet. Cette action déclenche automatiquement un processus préalable de validation.
Si le processus de validation du projet réussit, une boîte de dialogue s’ouvre pour vous permettre de choisir l’emplacement où vous souhaitez sauvegarder le listage des séquences (fichier .xml) qui est généré conformément à la norme ST.26.
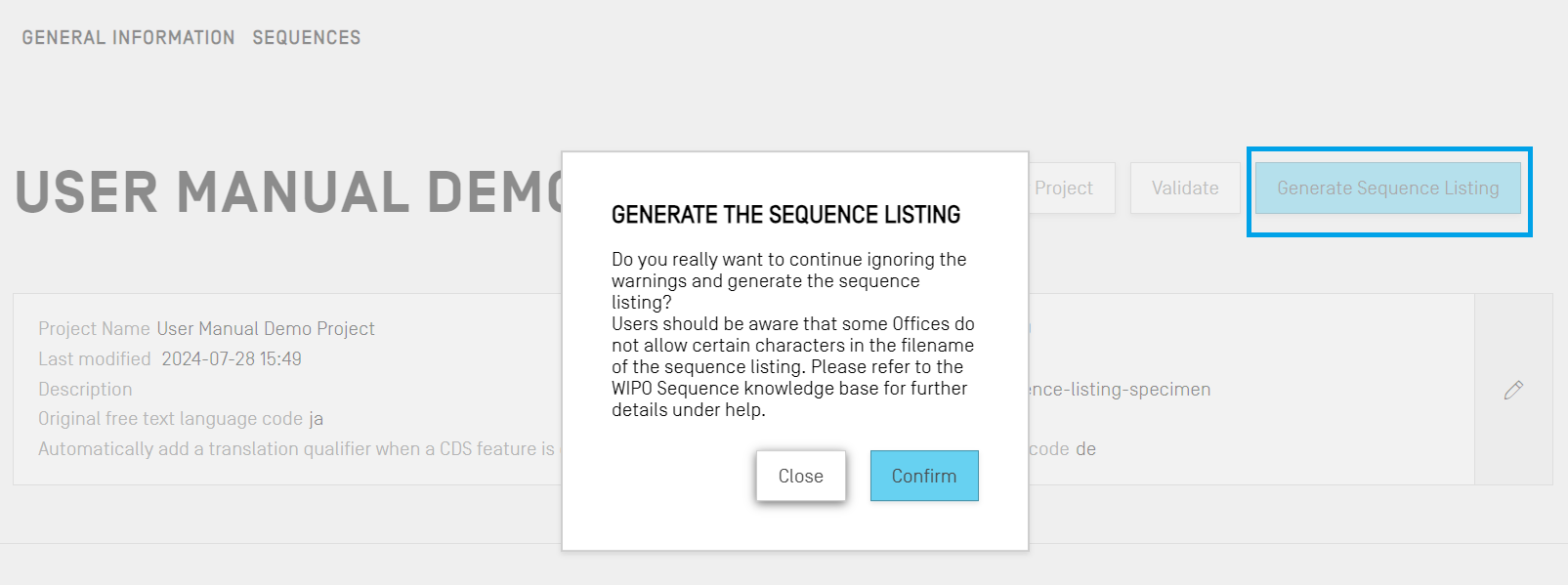
Si la validation du projet échoue, la vue du rapport de vérification s’affiche, ainsi qu’un bandeau rouge. Si le projet est valide, un bandeau bleu s'affiche.
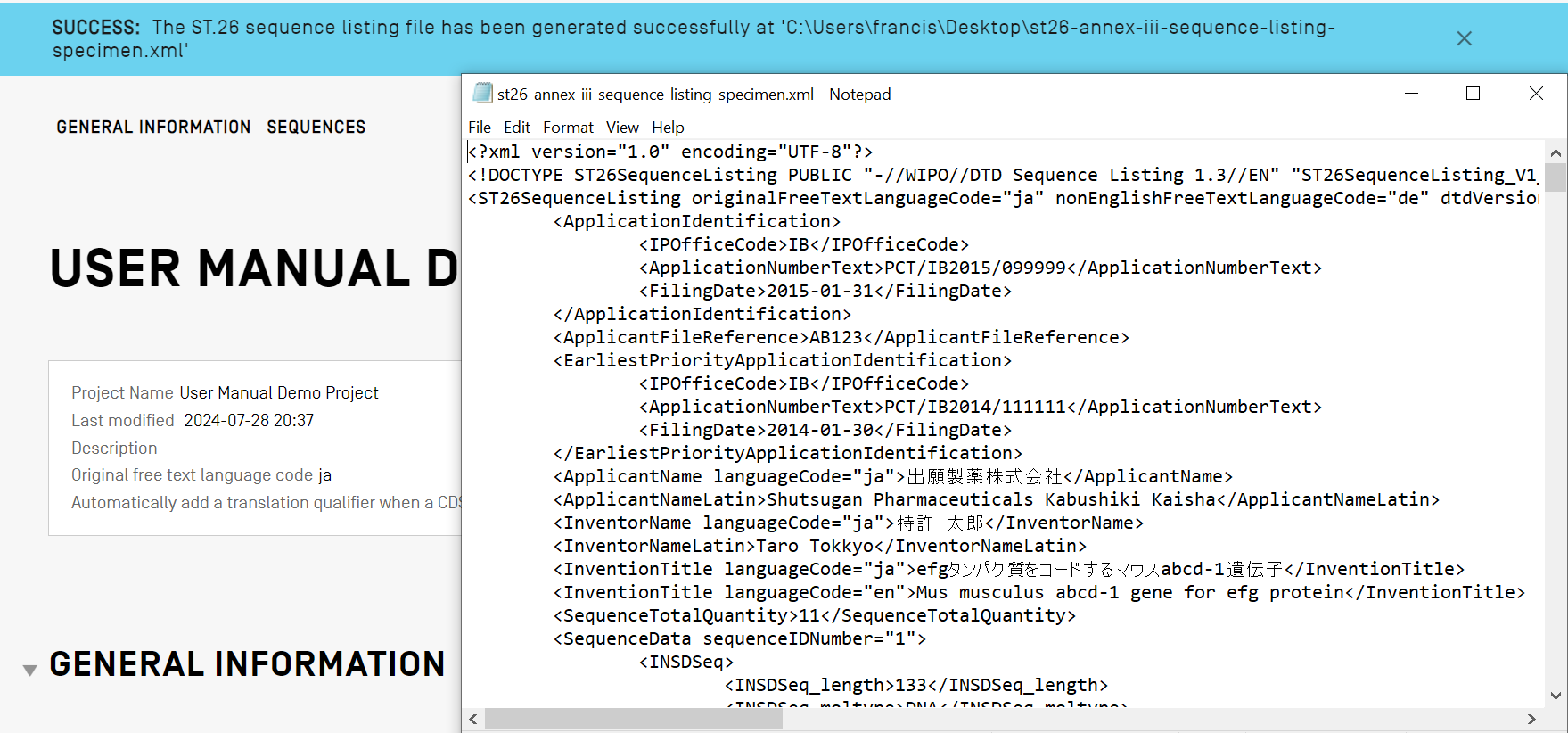
Afficher un listage des séquences
WIPO Sequence permet de générer un listage des séquences sous un format plus facile à lire pour vous que le XML. Une fois le listage des séquences généré, le fichier XML peut être affiché au format HTML ou exporté sous la forme d'un fichier texte. À l’aide de n’importe quel navigateur Internet, vous pouvez également sauvegarder le listage des séquences affiché au format HTML sous forme de fichier PDF. La fonctionnalité d'exportation est accessible à partir de la vue “Display the Sequence Listing” (Afficher le listage des séquences.
Si la génération du listage des séquences a échoué pour un projet donné, la vue de l'affichage du listage des séquences désactivera les boutons “Display Sequence Listing” (Afficher le listage des séquences) et “Export Sequence Listing as .txt file” (Exporter le listage des séquences sous la forme d'un fichier texte) et un message d'erreur va s'afficher.
Qualificatifs de texte libre dépendant de la langue
Les qualificateurs permettant de saisir une valeur en texte libre dans un projet sont présentés plus en détail dans la vue “LANGUAGE DEPENDENT QUALIFIERS” (QUALIFICATEURS DÉPENDANTS DE LA LANGUE) de la page Projets. Lorsqu’un qualificateur dépendant de la langue est ajouté au projet en cours, il s’affiche également dans cette vue.
Vous pouvez modifier une valeur de texte libre traduite associée à un qualificateur en cliquant sur la valeur “Qualifier Name” (Nom du qualificateur), ce qui ouvre un panneau de modification en dessous du tableau.
Vous devez indiquer le code de la langue source et le code de la langue cible pour l’exportation au format de fichier XLIFF des qualificateurs de texte libre. Les valeurs traduites devront être fournies par les traducteurs avant de réimporter le fichier XLIFF.
Veuillez noter que la valeur de qualificateur traduite, apparaissant dans la colonne “Non-English Qualifier Value” (Valeur de qualificateur autre que l'anglais), correspond à la langue sélectionnée spécifiée par le code langue pour texte libre autre que l’anglais.
Si vous cliquez sur le bouton “IMPORT FREE TEXT QUALIFIERS” (IMPORTER DES QUALIFICATEURS DE TEXTE LIBRE), le logiciel ouvrira l’explorateur de fichiers ce qui vous permettra de parcourir les dossiers pour trouver et sélectionner le fichier “XLIFF” à importer. Plusieurs étapes de validation sont prévues afin de s’assurer que les bonnes mises en correspondances entre les valeurs de la langue source et de la langue cible ont été effectuées.
Importer des qualificatifs de texte libre à partir d'un fichier XLIFF
Le fichier sélectionné doit être au format XLIFF et contenir les éléments de données suivants :
- Nom du projet
- Code de la langue cible
- Code de la langue source
- Pour chaque élément unitaire XLIFF :
- Identifiant unique du qualificateur (format : un chiffre précédé de la lettre “q”)
- Valeur du qualificateur dans la balise de la langue source
- Valeur du qualificateur dans la balise de la langue cible.
Une fois que vous avez confirmé le fichier à importer sélectionné, le logiciel vous demandera de vérifier vous voulez vraiment continuer en confirmant une série d’étapes de vérification :
- Le système compare le nom du projet dans le fichier source avec le nom du projet sélectionné.
- Le système vous informera si aucune correspondance n’a été trouvée pour certains qualificateurs.
- Le système vous informera des modifications liées à la langue source et aux valeurs des qualificateurs
- Le système vous informera des modifications liées à la langue cible et aux valeurs traduites associées aux qualificateurs.
Une fois ces étapes terminées, l'utilisateur verra apparaître un bandeau bleu en haut de la page : “SUCCESS: THE FREE TEXT QUALIFIER HAS BEEN IMPORTED SUCCESSFULLY” (OPÉRATION RÉUSSIE : LE QUALIFICATEUR DE TEXTE LIBRE A ÉTÉ IMPORTÉ AVEC SUCCÈS), ainsi qu’un rapport d’importation présentant le détail des valeurs précédentes et des valeurs actuelles importées pour les qualificateurs de texte libre dépendants de la langue. Les utilisateurs peuvent revenir à la vue Qualificateur de texte libre en cliquant sur le bouton “RETURN TO FREE TEXT QUALIFIERS” (RETOUR AUX QUALIFICATEURS DE TEXTE LIBRE).
Exporter des qualificatifs de texte libre au format XLIFF
Si l’utilisateur clique sur le bouton “EXPORT FREE TEXT QUALIFIERS” (EXPORTER LES QUALIFICATEURS DE TEXTE LIBRE) en haut de la vue, puis sélectionne dans la boîte de dialogue le nom du fichier et l’emplacement où enregistrer le fichier texte de qualificateurs, tous les qualificateurs de texte libre du projet seront exportés et enregistrés dans un fichier au format XLIFF.
Ce fichier comprendra :
- La source du projet
- La langue cible du projet
- Les qualificateurs de texte libre
- Les valeurs en texte libre traduites associées aux qualificateurs
- Les informations associées au qualificateur et à la caractéristique communiqués dans le tableau.
Il est possible de consulter, de modifier et d’importer à nouveau ce fichier dans le logiciel après avoir fourni les valeurs traduites appropriées.
Saisir les données d'un projet
Un projet est divisé en deux sections de la même manière que le listage des séquences généré : informations générales et données relatives aux séquences. Vous pouvez couper et coller des données dans un projet ou importer des données d'un autre projet ou d'un listage des séquences. En général, un clic sur l'icône du crayon vous permet de modifier les champs du projet. Les champs obligatoires sont indiqués par un astérisque (“*”).
Informations générales
La section sur les informations générales indique comment vous pouvez saisir des informations concernant la demande de brevet à laquelle sera associé le listage des séquences généré. La première sous-section, Application Identification (Identification de la demande), concerne le statut et les informations relatives à la demande de brevet du projet sélectionné. Les instructions ci-dessous vous guident pas à pas dans les informations à fournir dans cette section.
Étape 1 : Identification de la demande
Pour saisir des informations dans cette sous-section, cliquez sur l’icône représentant un crayon qui est entourée dans la figure à droite de l’écran. Vous devez ensuite fournir des informations en suivant les étapes suivantes :
- Si un numéro a déjà été attribué à la demande, vous devez choisir le code “IP Office” (code de l’Office de propriété intellectuelle) sous lequel la demande a été déposée. Ceci est le code selon la norme ST.3 de l’OMPI.
- Vous devez indiquer si vous avez déjà reçu un numéro de demande ou si vous avez fourni une référence de dossier du déposant, en sélectionnant le bouton radio correspondant.
- Si vous ne disposez pas de numéro de demande, vous DEVEZ indiquer la référence du dossier du déposant dans ce champ.
- Si un numéro a déjà été attribué à la demande, vous devez l’indiquer dans ce champ.
- Choisir la date de dépôt de la demande au moyen du sélecteur de date, si une date a déjà été attribuée.
- Cliquez sur le bouton “Save” (Sauvegarder).
Dans l'exemple présenté, toutes les valeurs optionnelles ont également été saisies :

Note : même si les valeurs obligatoires ont été saisies, un message d'avertissement apparaîtra toujours dans le rapport de vérification indiquant que “The application identification number is absent. Le numéro de demande est obligatoire si le numéro de demande a été attribué.”
Étape 2 : Demande(s) établissant la priorité
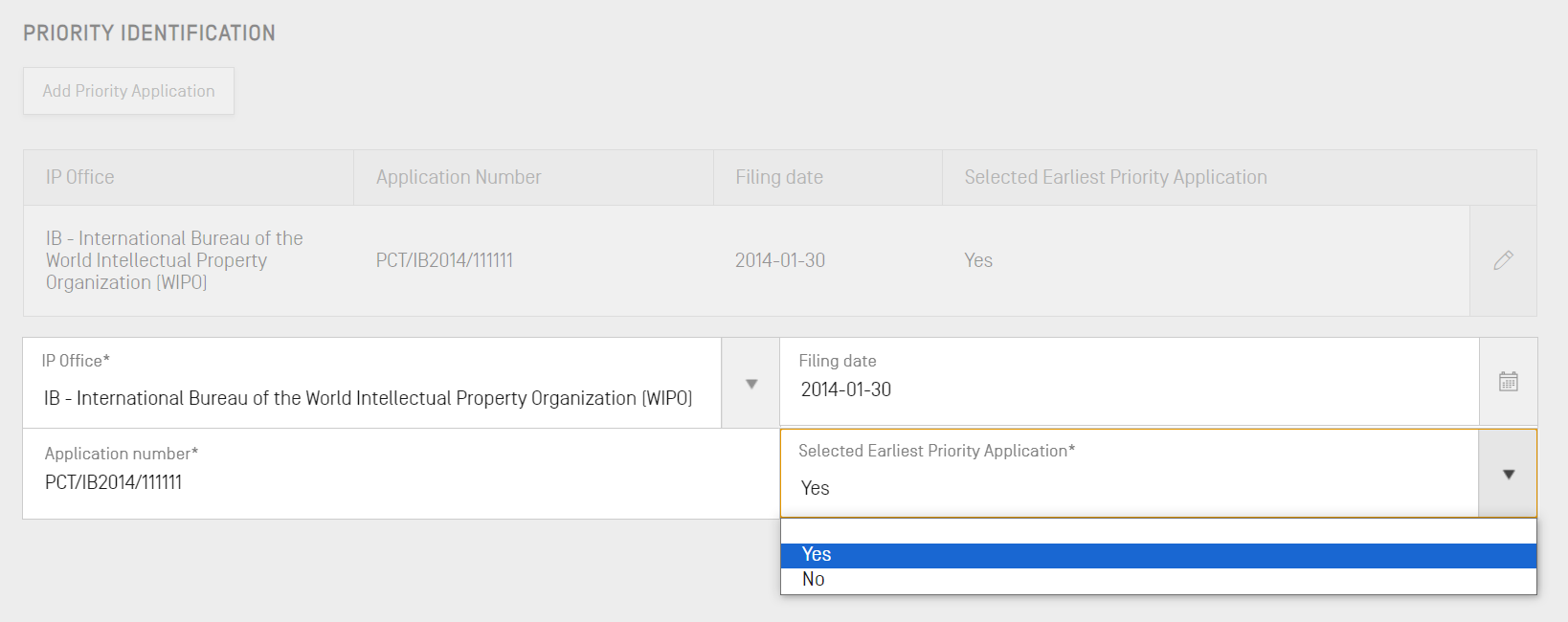
Ensuite, s'il existe une demande établissant la priorité liée à la présente demande, ces détails sont saisis dans la sous-section Demande établissant la priorité. Pour ajouter au projet une demande établissant la priorité , vous devez cliquer sur le bouton "Add Earliest Priority Identification" (Ajouter la demande établissant la priorité) dans la section des informations générales.
Pour indiquer que la demande établissant la priorité est la plus ancienne, vous devez choisir “Yes” (Oui) dans le menu déroulant “Selected Earliest Priority Application” (Demande établissant la priorité la plus ancienne). Cela permet d'indiquer que la demande établissant la priorité est la plus ancienne dans le listage des séquences. Pour finir, cliquez sur le bouton bleu “Add Earliest Priority Application” (Ajouter la demande établissant la priorité la plus ancienne).
Étape 3 : Déposant/Inventeur
Pour ajouter au projet des données concernant un nouveau déposant ou inventeur, vous devez cliquer sur le bouton “Add Inventor” (Ajouter un inventeur) ou “Add Applicant” (Ajouter un déposant) dans la section des informations générales de la vue détaillée du projet. Les étapes permettant d’effectuer ces deux actions étant identiques, seules des instructions générales seront fournies ici; toutefois, le processus doit être effectué deux fois si à la fois un déposant et un inventeur doivent être ajoutés au projet, même si le déposant est aussi l’inventeur.
Lorsque vous cliquez sur le bouton, une zone grisée s'ouvrira avec deux boutons radio. Si “Existing applicant/inventor” (Déposant/inventeur existant) est sélectionné, vous pouvez choisir dans une liste déroulante un nom parmi les personnes et les organisations déjà enregistrées en local dans le logiciel.
Si l'option "Nouveau déposant/inventeur" est sélectionnée, vous devez saisir les informations requises lors de la création d'une nouvelle personne/organisation.
Une fois les informations saisies, cliquez sur le bouton “Add Applicant” (Ajouter un déposant) ou “Add Inventor” (Ajouter un inventeur).

Note : un seul déposant est nécessaire pour que le listage des séquences soit considéré comme valide. Il faut donc qu’un déposant ou un inventeur soit indiqué comme étant le déposant ou l’inventeur principal. C’est ce déposant ou cet inventeur qui apparaîtra dans le listage des séquences généré.
Étape 4 : Titre de l'invention
La dernière étape consiste à ajouter le "Invention Title" (Titre de l'invention) dans la section des informations générales.
Pour ajouter un nouveau titre d'invention :
- Cliquez sur le bouton "Add Invention Title" (Ajouter le titre de l'invention).
- Dans la zone grisée qui apparaît, vous devez saisir le titre de l’invention et préciser la langue dans laquelle le titre est écrit.
- Cliquer sur le bouton bleu "Add Invention Title" (Ajouter le titre de l'invention).

En vertu de la norme ST.26 de l'OMPI, dans un listage des séquences, le titre d'une invention doit obligatoirement être écrit dans la langue de dépôt de la demande. Cependant, un projet peut aussi comporter, à titre facultatif, d’autres titres de l’invention dans d’autres langues, mais un seul titre d’invention pour chaque langue. Tout titre supplémentaire peut être ajouté en suivant les étapes décrites ci-dessus.
Données sur les séquences
La section “Sequences” (Séquences) de la vue détaillée du projet vous permet de fournir les informations techniques concernant les séquences elles-mêmes. WIPO Sequence fournit un certain nombre de moyens pour saisir les données sur les séquences dans un projet, une séquence pouvant être saisie manuellement, importée ou insérée. La sous-section ci-après contient de plus amples détails sur les étapes à suivre pour effectuer ces actions.
Créer une séquence
Les étapes pour créer une séquence dans le projet sont les suivantes :
- Cliquez sur le bouton “Create new sequence” (Créer une nouvelle séquence). Le “panneau des séquences” s’affiche.
- Nommez la séquence en saisissant le nom souhaité dans le champ “Sequence Name” (Nom de la séquence). Vous pouvez également laisser le champ “Sequence Name” vide et WIPO Sequence attribuera un nom par défaut à la nouvelle séquence. Les noms par défaut commencent par “Seq”, suivi d’un nombre itératif croissant (“Seq_1”, “Seq_2”, “Seq_3”).
Note : le champ “Sequence Name” a pour seul but de permettre à l’utilisateur de distinguer plus facilement les séquences au sein du projet; ce nom n’apparaîtra pas dans le fichier XML du listage des séquences.
- Utilisez la liste déroulante “Molecule Type*” (Type de molécule*) pour sélectionner l’un des trois types de molécules autorisés par la norme ST.26 de l’OMPI (“ADN”, “ARN”, “AA”). Si vous souhaitez créer une séquence contenant à la fois des segments d’ADN et d’ARN, veuillez choisir l’ADN comme type de molécule.
- Saisissez ou collez les résidus de la séquence dans le champ “Residues*” (Résidus).
- Saisissez ou collez le nom de l’organisme source dans le champ “Organism name*” (Nom de l’organisme*). Pour sélectionner un nom d’organisme à partir de la liste prédéfinie de noms d’organismes, il suffit de commencer à saisir le nom de l’organisme et une liste déroulante apparaît. Sélectionnez le nom de l’organisme souhaité, si celui-ci apparaît. Dans le cas contraire, l’outil peut vous inviter à ajouter l’organisme à la base de données d’organismes personnalisés stockée localement.
- Si le type de molécule est “ADN” ou “ARN”, vous devez sélectionner un type de molécule spécifique dans la liste déroulante “Qualifier Molecule Type”, qui contiendra les valeurs appropriées pour le type de molécule sélectionné à l’étape 3. Notez que si le type de molécule est “AA”, la case “Qualifier Molecule Type” sera automatiquement remplie avec la valeur “protein”.
- Si la case “Mark as an intentionally skipped sequence” (Marquer comme séquence délibérément omise) est cochée, la séquence sera incluse dans le fichier XML du listage des séquences résultant en tant que “intentionally skipped sequence” (séquence intentionnellement omise); c’est-à-dire qu’elle sera dans le format spécifié au paragraphe 58 de la norme ST.26 de l’OMPI. À noter que la case “Mark as an intentionally skipped sequence” doit être cochée pour enregistrer une séquence comportant moins de 10 résidus de nucléotides non “n” ou moins de 4 résidus d’acides aminés non “X”. Lorsqu’une séquence est intentionnellement omise, toutes les contraintes de renseignement des éléments obligatoires disparaissent du panneau des séquences et la séquence ainsi sauvegardée sera ignorée au moment de la validation du projet.
- Si le type de molécule de la séquence est “ADN”, une case intitulée “La séquence contient à la fois des fragments d’ADN et d’ARN” sera visible. Si la séquence contient à la fois des fragments d’ADN et d’ARN, le fait de cocher cette case permet d’ajouter facilement les caractéristiques requises par la norme ST.26 de l’OMPI, paragraphe 55, pour décrire les hybrides ADN/ARN. Si la case “The sequence contains both DNA & RNA fragments” (La séquence contient à la fois des fragments d’ADN et d’ARN) est cochée, le panneau s’allonge et présente des champs permettant de décrire chaque fragment d’ADN et d’ARN au moyen de la caractéristique “misc_feature” et l’emplacement correspondant. Pour chaque fragment, sélectionnez “ADN” ou “ARN” dans le menu déroulant du champ “Molecule Type” (Type de molécule), puis saisissez un emplacement dans le champ “Location” (emplacement). Du texte supplémentaire peut être ajouté dans le champ “Further text” (texte supplémentaire). Vous pouvez créer autant de ces caractéristiques que nécessaire en cliquant sur le bouton “Add new ‘misc_feature’ feature” (Ajouter une nouvelle caractéristique “misc_feature”). À noter que toute la longueur de la séquence doit être couverte par des clés de caractérisation “misc_feature” de sorte que chaque résidu soit indiqué comme étant de l’“ADN” ou de l’“ARN”.
- Pour terminer et enregistrer la séquence, cliquez sur le bouton gris “Create sequence” (Créer une séquence) ou sur le bouton bleu “Create & Display Sequence” (Créer et afficher une séquence). Si vous cliquez sur le bouton bleu “Create & Display Sequence” (Créer et afficher une séquence), un affichage se déploie après création de la séquence, pour vous permettre de vérifier les valeurs. Il sera visible dans la vue détaillée du projet, sous la section des séquences.
Note : si la séquence est une séquence de nucléotides (“ADN” ou “ARN”), seuls les symboles énumérés dans la norme ST.26 de l’OMPI, annexe I, tableau 1, sont autorisés (a, c, g, t, m, r, w, s, y, k, v, h, d, b, n). À noter que le symbole “u” n’est pas autorisé. Les symboles saisis en lettres majuscules seront convertis en lettres minuscules. Tout symbole non alphabétique saisi sera automatiquement supprimé (espaces, chiffres, *, -, etc.).
Si la séquence est une séquence d’acides aminés (“AA”), seuls les symboles énumérés dans la norme ST.26 de l’OMPI, annexe I, tableau 3, sont autorisés (A, R, N, D, C, Q, E, G, H, I, L, K, M, F, P, O, S, U, T, W, Y, V, B, Z, J, X). À noter que seuls les symboles d’acides aminés à une lettre sont autorisés. Si votre séquence d’acides aminés est représentée par des symboles à trois lettres (par exemple, Met-Arg-Leu-Trp-Ile), elle doit d’abord être convertie en symboles à une lettre avant d’être saisie ou collée dans le champ “Residues”.p>
La séquence qui vient d’être créée peut être consultée à la fin du listage des séquences; elle s’accompagne du numéro d’identifiant de séquence disponible suivant. Si vous souhaitez redéfinir la position d'une séquence dans le listage, veuillez suivre ces étapes.
Importer une séquence
Les séquences peuvent aussi être importées directement depuis des fichiers dans un projet. Les formats de fichier acceptés sont indiqués dans la section 3. Une fois le fichier choisi, le logiciel détecte automatiquement son format.
- Pour démarrer, cliquez sur le bouton “Import sequence” (Importer la séquence).
- Cliquez sur "Upload file [.txt, .xml]" (Télécharger le fichier [.txt, .xml]). Dans la boîte de dialogue qui s’est ouverte, choisissez le fichier contenant les données de séquence à importer. Le logiciel détecte le format du fichier et effectue certaines tâches de validation au moment de l’importation. Le logiciel accepte cinq formats de séquences pour l’importation de séquences dans un projet : texte brut, multiséquence, FASTA, ST.26 et ST.25.
- Si le fichier choisi est au format ST.25 ou ST.26 de l'OMPI, vous verrez tout d’abord une case à cocher intitulée “Select Range Sequences” (Choisir une fourchette de séquences). Une fois cette case cochée, un tableau s’ouvre et présente le numéro d’identifiant de chaque séquence du fichier dans l’ordre dans lequel les séquences ont été ajoutées au listage dans le projet. Si vous ne souhaitez pas importer toutes les séquences dans le projet, vous pouvez définir la fourchette des numéros d’identifiant de séquence souhaités. Vous pouvez saisir une ou plusieurs séquences, ou un listage de séquences séparées par une virgule, ou encore une fourchette de séquences sous la forme x-y. For exemple : “1, 3, 7, 13-20, 30-50”.
- Si le fichier importé est au format multi-séquence, vous verrez s'afficher une case à cocher intitulée "Select Range Sequences" (Choisir une fourchette de séquences), qui une fois cochée affiche un tableau de prévisualisation indiquant les numéros d'identifiant des séquences correspondantes dans le fichier, ainsi que la description de chaque séquence, à savoir le nom de la séquence, le type de molécule et le nom de l'organisme. Vous devez définir la fourchette des numéros d'identifiant de séquence que vous souhaitez importer dans le listage des séquences du projet. Par défaut, le nombre total de séquences du fichier de listages des séquences sélectionné s’affichera sous forme de fourchette.
- Les deux derniers formats acceptés pour le processus d'importation de séquences sont les formats texte brut et FASTA. Ces formats ne permettent de définir qu’une seule séquence par fichier. Lorsqu'un fichier en texte brut ou FASTA est choisi dans le processus d'importation, le logiciel affiche le panneau correspondant. Vous devez poursuivre en complétant les champs obligatoires.
- Une fois l’importation réussie, le logiciel présente l'écran “Import Report” (Rapport d’importation).
Insérer une séquence
Pour insérer une séquence à une position particulière dans le listage, vous devez cliquer sur le bouton “Insert Sequence” (Insérer une séquence). Une zone grise apparaîtra alors. Vous devez fournir toutes les informations nécessaires pour créer une séquence, et indiquer aussi, en haut à gauche du panneau, la position à laquelle la séquence doit apparaître dans le listage. Pour finir, vous pouvez cliquer sur les boutons "Insert sequence" (Insérer une séquence) ou "Insert & Display Sequence" (Insérer et afficher une séquence).

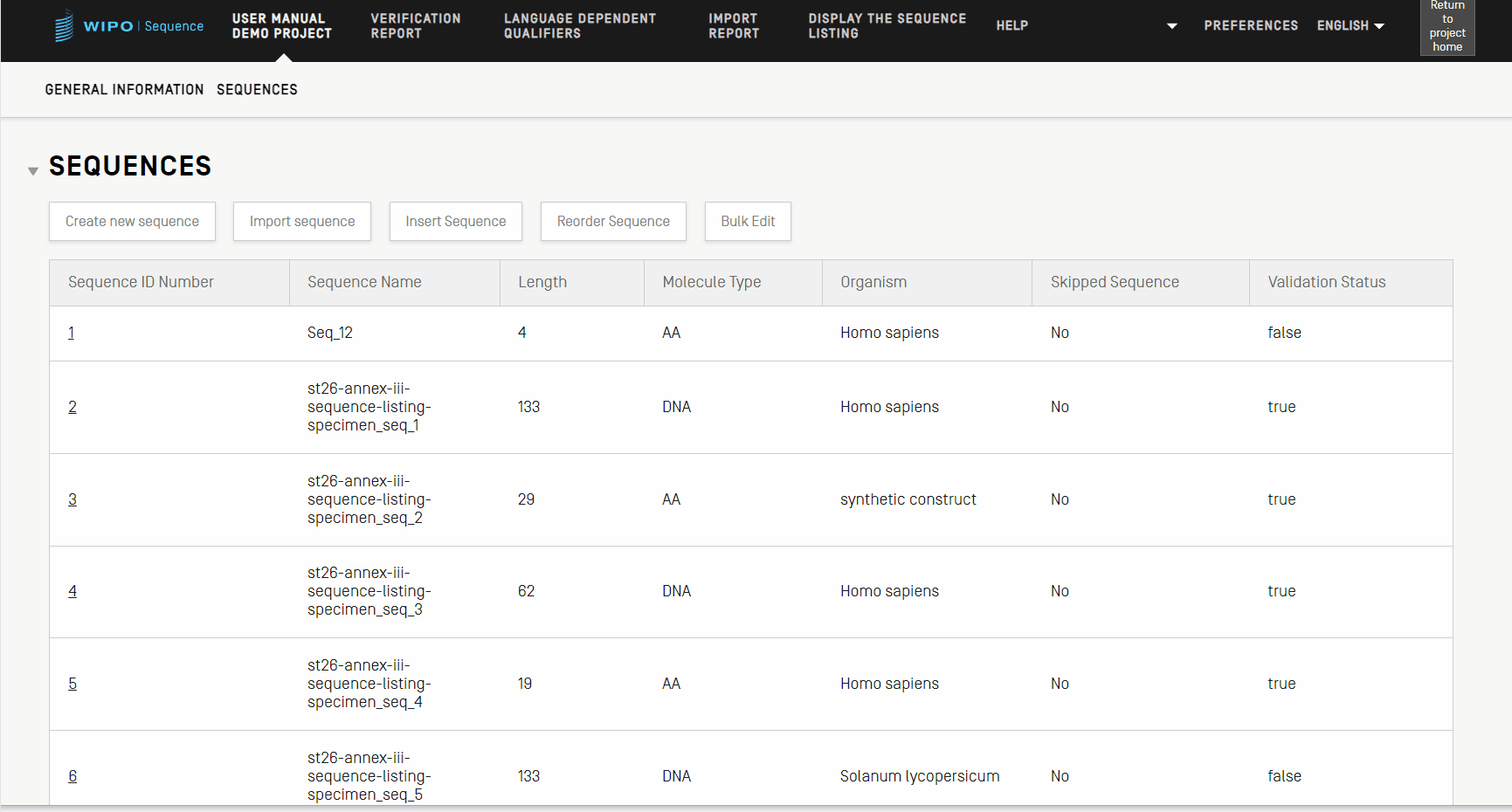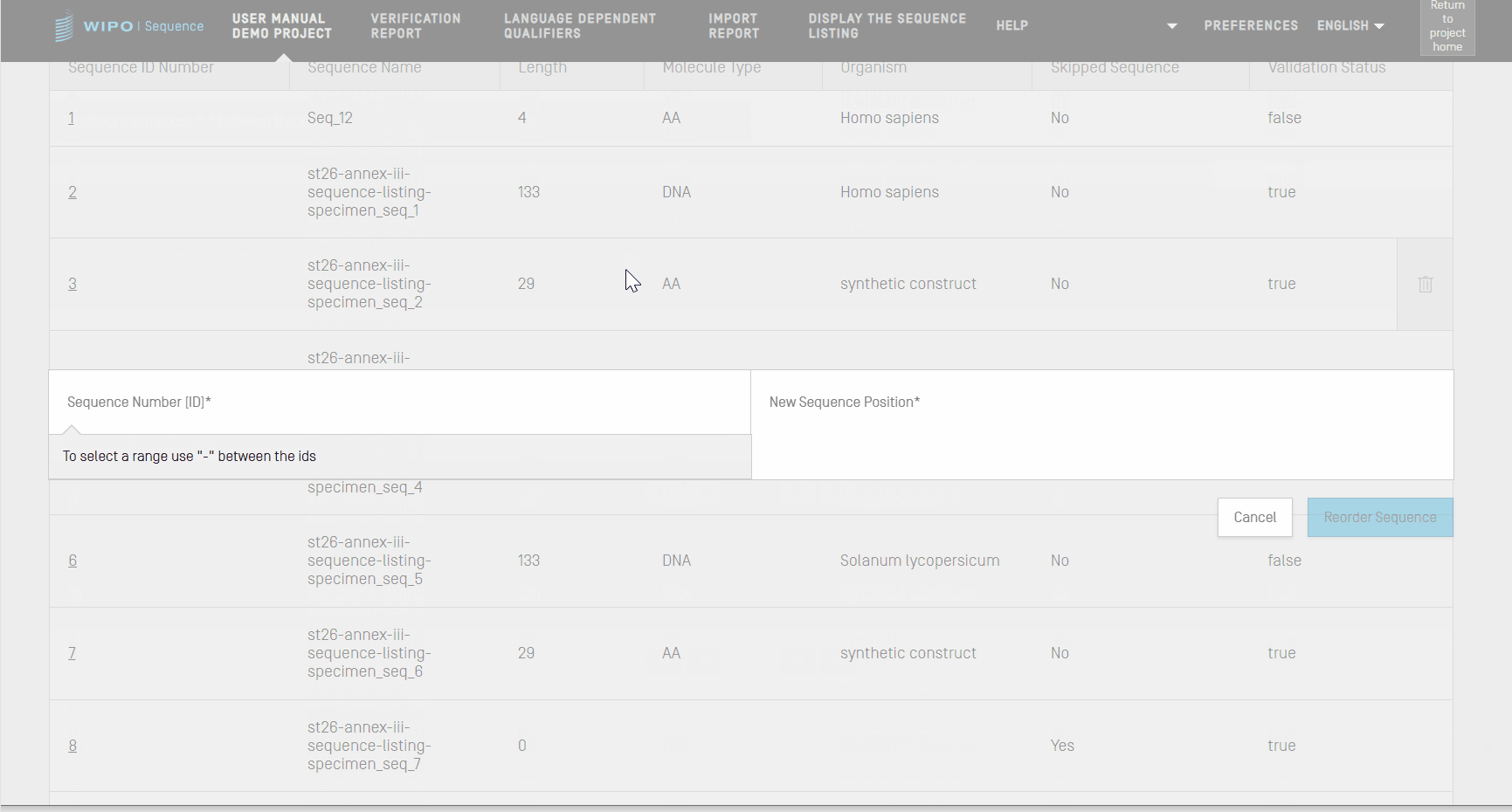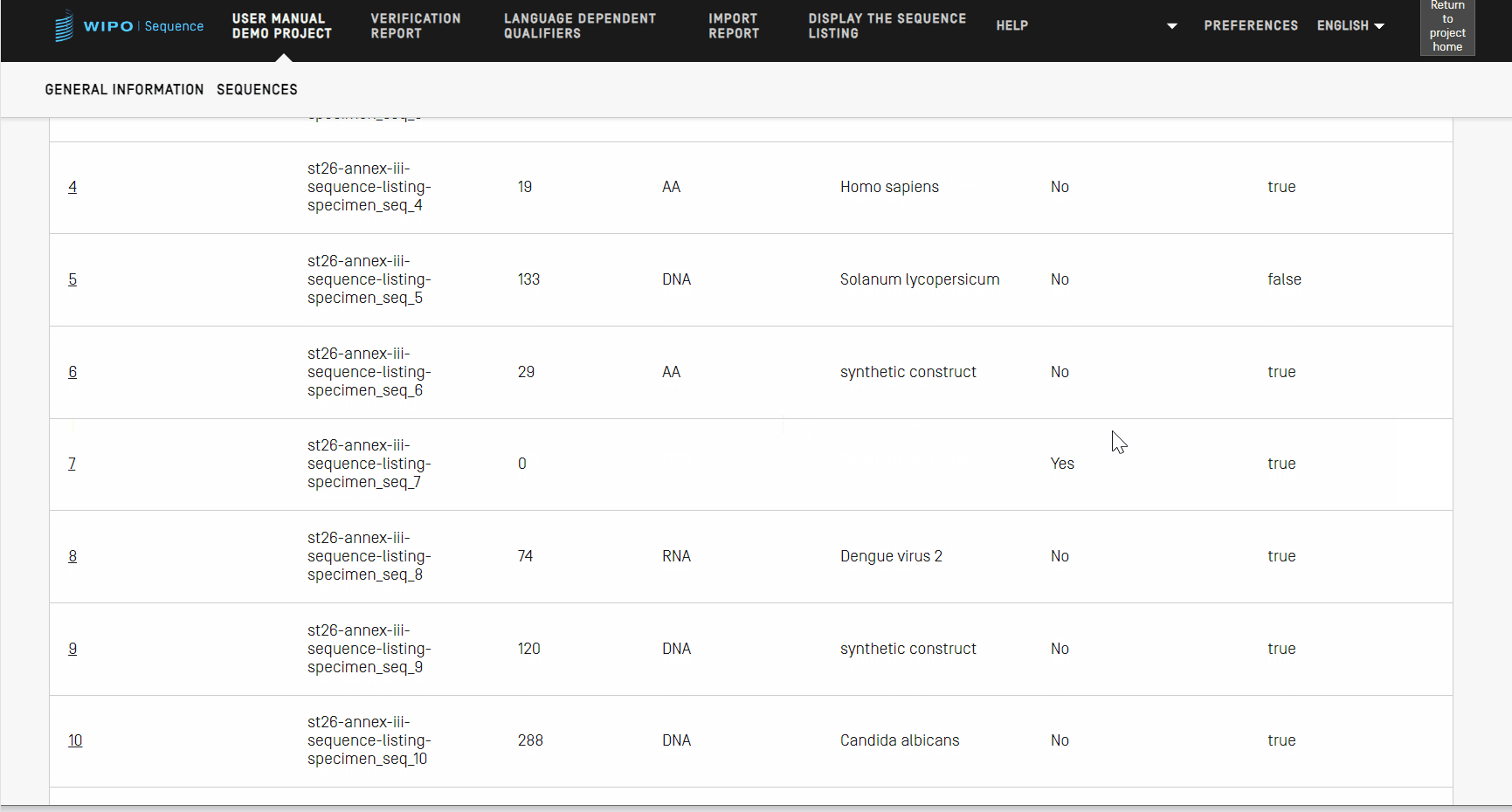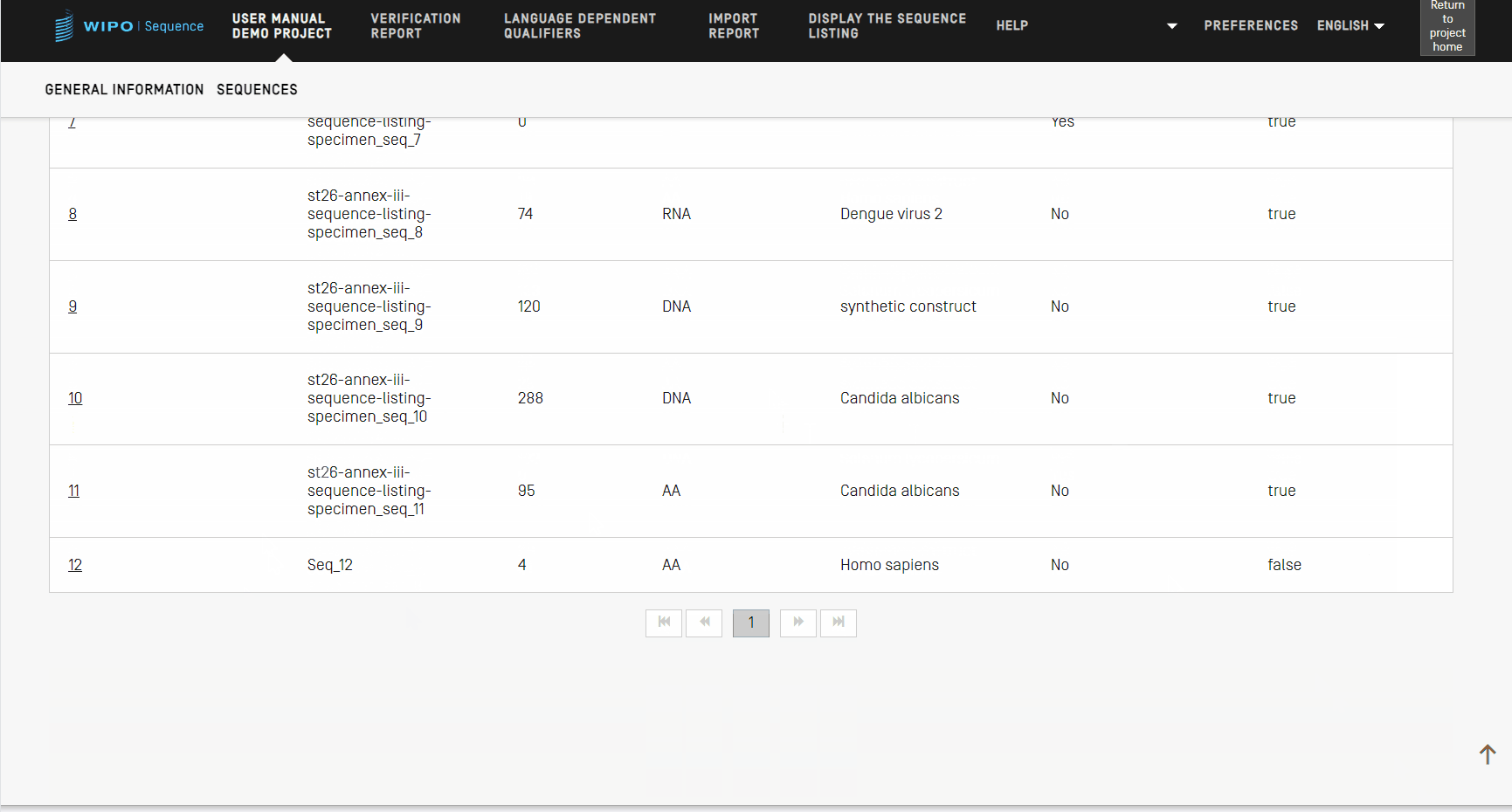
Réordonner une séquence
Vous pouvez réorganiser l’ordre dans lequel les séquences devraient apparaître dans le listage du projet en suivant les étapes présentées dans la vidéo ci-après.
Modifier en bloc
Les séquences peuvent être modifiées une à une en cliquant sur l'icône du crayon, mais vous pouvez utiliser la fonction de modification en bloc lorsque des modifications sont nécessaires pour plusieurs séquences. Bien qu’il soit possible d’accéder aux séquences et de les modifier individuellement, cette méthode n’est pas faisable pour les projets comportant un très grand nombre de séquences. Il existe un certain nombre de champs qui peuvent être modifiés de cette manière, plus de précisions étant données ci-après.
- Commencez par cliquer sur le bouton "Bulk Edit" (Modifier en bloc).
- Choisissez le “Type of bulk edit” (type de modification en bloc) dans la liste déroulante.
- Si l'option "Qualifier molecule type" (Type de molécule du qualificateur) est sélectionnée, le système vous invitera à sélectionner le type de séquences d'acides nucléiques auquel la modification en bloc sera appliquée. Le système vous avertit également que le qualificateur “mol_type” pour les séquences où l’organisme est “synthetic sequence” doit être défini sur “other DNA” ou “other RNA” et que si vous modifiez ces valeurs, une erreur sera générée lors de la validation du projet. Le système affichera un aperçu des séquences à modifier en bloc avec les caractéristiques spécifiées. Durant la modification, le système vous informera que SEULES les séquences d’acides nucléiques peuvent avoir la valeur du qualificateur “mol_type” modifiée (car cette même valeur pour les séquences d’acides aminés est automatiquement définie par le système comme “protéine”).
- Si “Organism” (Organisme) est sélectionné, vous devez saisir la fourchette des identifiants de séquence à modifier. Ensuite, si vous avez choisi de modifier la valeur de l'organisme par “synthetic construct” (construction synthétique), le système vous informera que le qualificateur “mol_type” sera automatiquement modifié par “other DNA” (autre ADN) ou “other RNA”(autre ARN) en fonction du type de molécule de la séquence (par exemple, l'ADN).
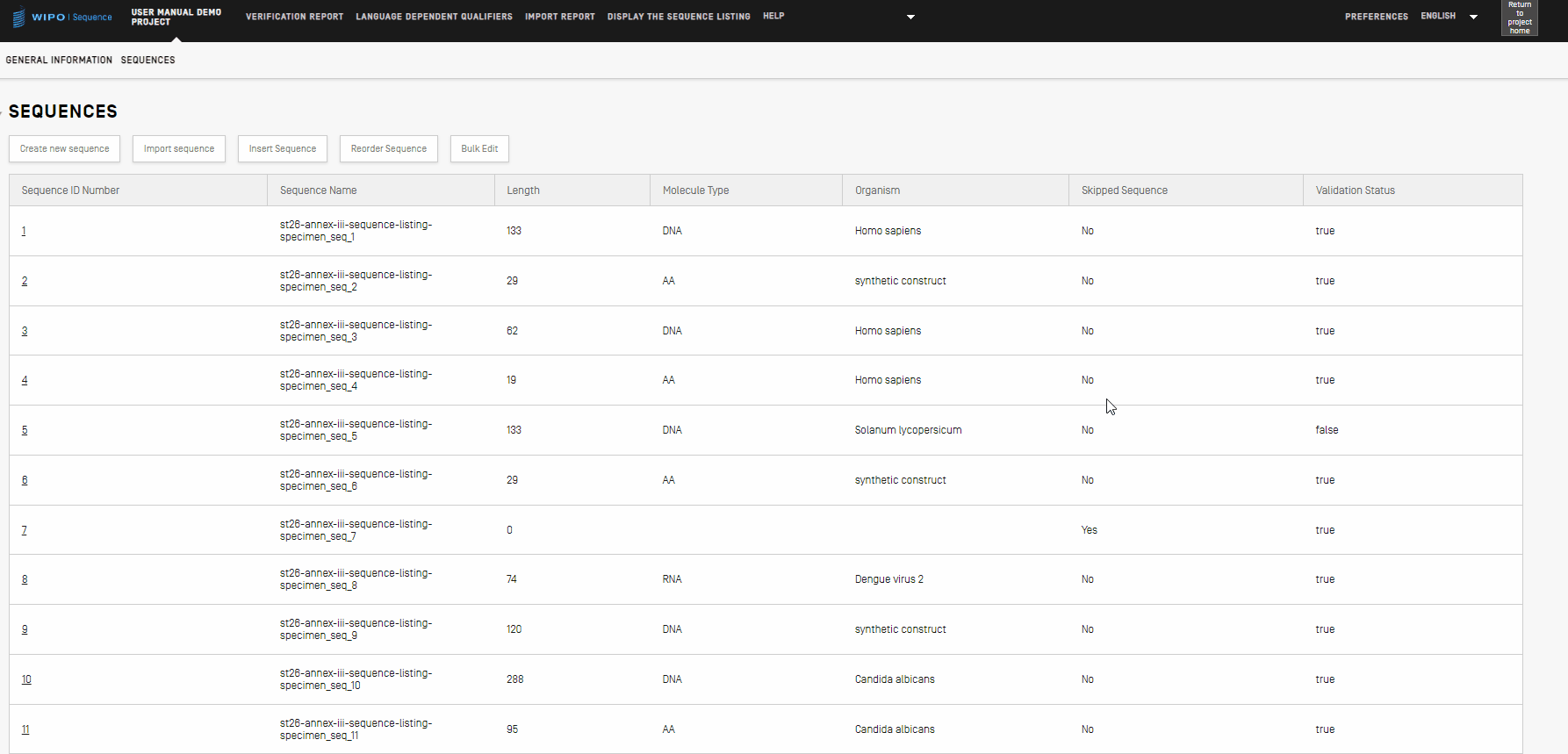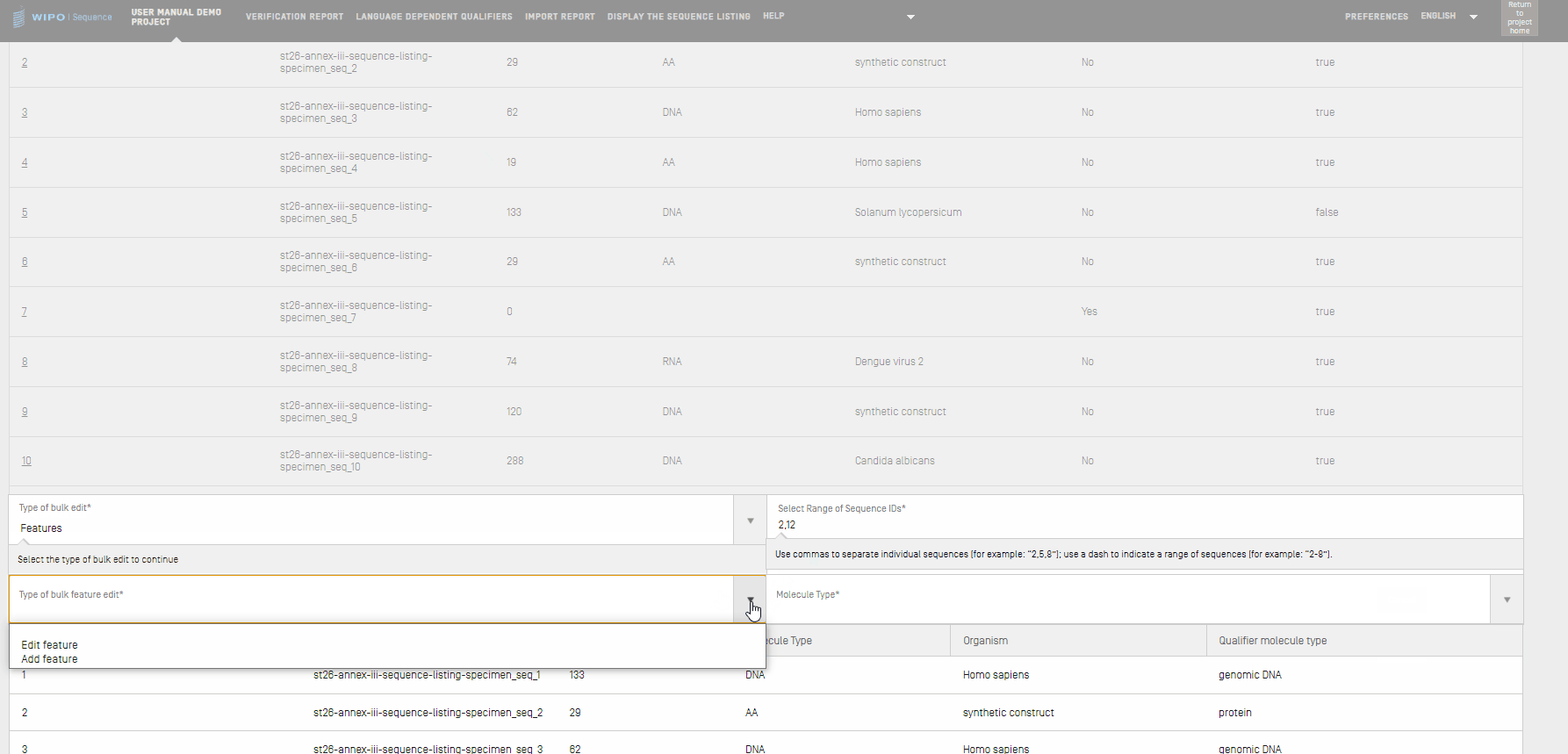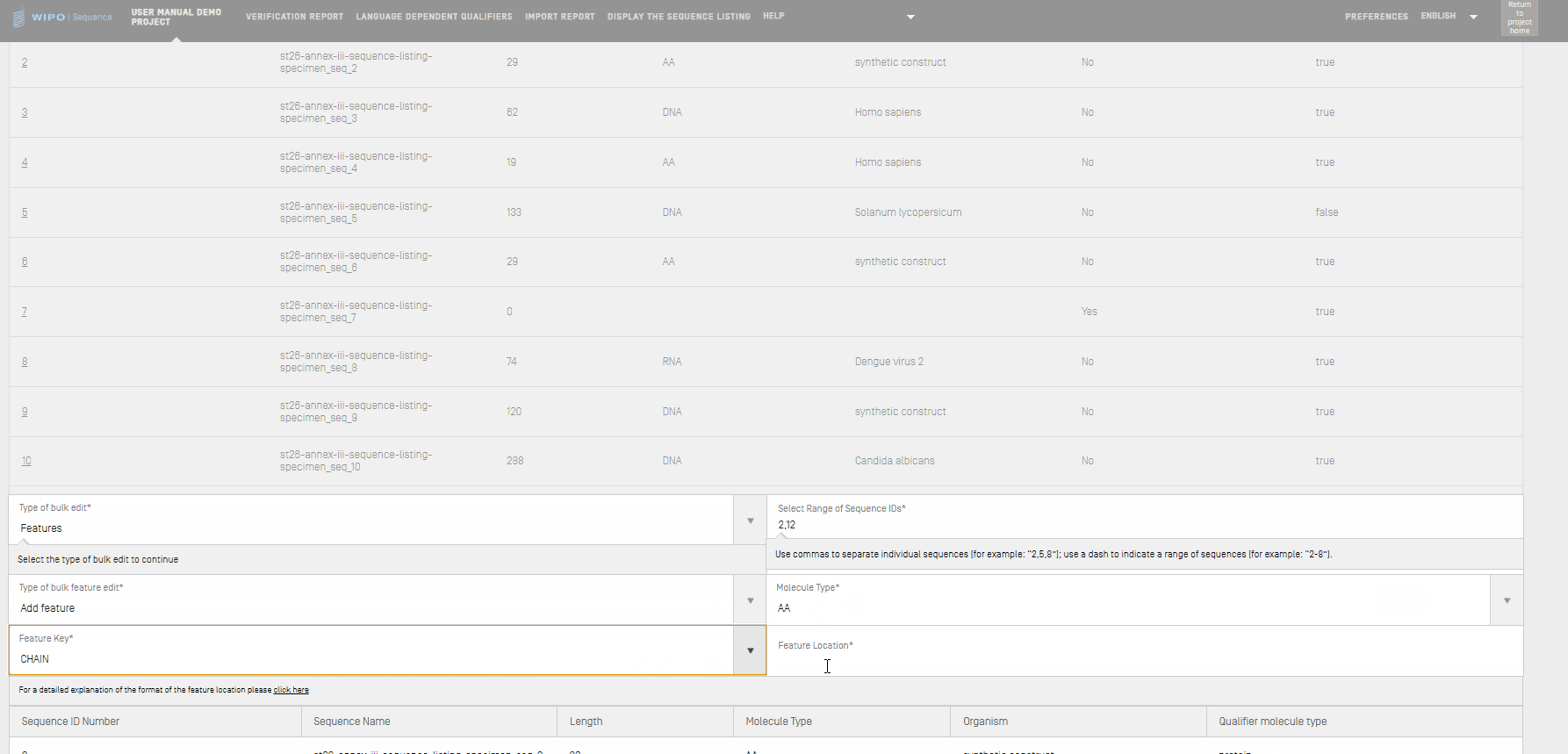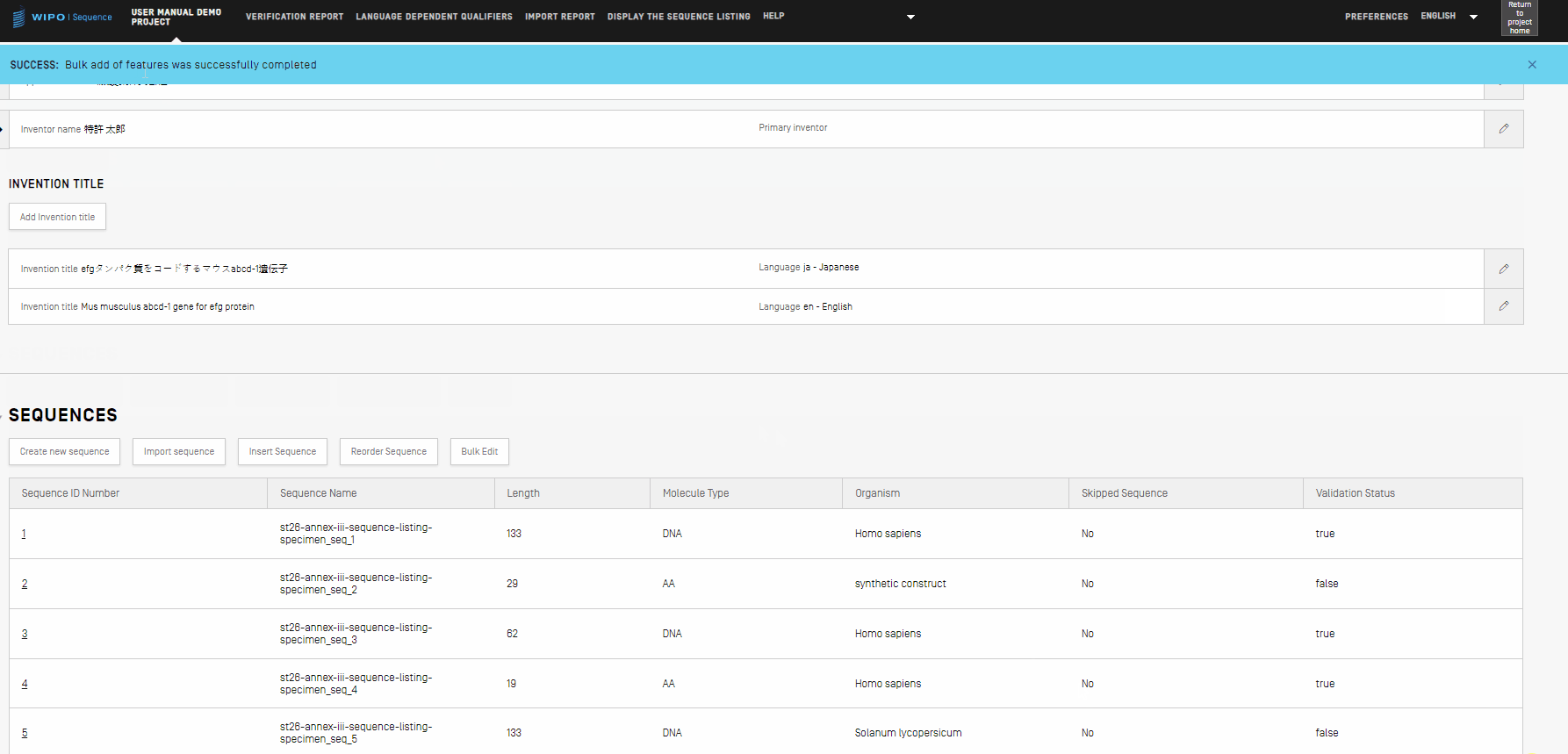
- Si “Features” (Caractéristiques) est sélectionné, vous devez alors préciser si vous souhaitez modifier les caractéristiques existantes ou en ajouter de nouvelles. Vous devez saisir le “Molecule Type” (Type de molécule) et la fourchette des identifiants de séquences à modifier.
- Si vous sélectionnez comme type de modification en bloc “Edit feature” (Modifier une caractéristique), par exemple en remplaçant la valeur de l'emplacement d'une caractéristique CDS par “complement(join(1..30,61..90))”, vous devriez d'abord saisir les éléments suivants dans la zone de texte “Select Range of Sequence IDs” (Sélectionner la fourchette des identifiants de séquence) : les identifiants de séquence correpondants, le “Molecule Type” (Type de molécule), ainsi que la “Feature Key” (Clé de caractérisation) correspondante et son “Feature Location” (Emplacement de la caractéristique). Pour terminer, sélectionnez “Edit sequences” (Modifier les séquences).
- Si vous sélectionnez comme type de modification en bloc “Add feature” (Ajouter une caractéristique), par exemple en ajoutant une nouvelle caractéristique “CHAIN” avec pour emplacement de caractéristique “1..4”, vous devriez d'abord saisir les éléments suivants dans la zone de texte “Select Range of Sequence IDs” (Sélectionner la fourchette des identifiants de séquence) : les identifiants de séquence correpondants, le “Molecule Type” (Type de molécule), ainsi que la “Feature Key” (Clé de caractérisation) correspondante et son “Feature Location” (Emplacement de la caractéristique).
- Si “Bulk skip” (Omettre en bloc) est sélectionné, vous devez alors saisir dans la zone de texte “Select Range of Sequence IDs” (Sélectionner la fourchette des identifiants de séquence) la fourchette des identifiants de séquence que vous souhaitez omettre.
- Si “Bulk delete” (Supprimer en bloc) est sélectionné, vous devez indiquer dans la zone de texte “Select Range of Sequence IDs” (Sélectionner la fourchette des identifiants de séquence) la fourchette de séquences que vous souhaitez supprimer.
- Pour toute opération de modification en bloc, après avoir cliqué sur le bouton “Edit sequences” (Modifier les séquences), le logiciel vous informera de la réussite de l'opération en affichant un bandeau bleu Un exemple est présenté ci-dessous.
Saisie des données relatives aux caractéristiques
En vertu de la norme ST.26 de l’OMPI, toute séquence DOIT s’accompagner d’au moins une caractéristique “source”. Chaque caractéristique source doit avoir deux qualificateurs obligatoires : organisme et mol_type.
Le tableau des caractéristiques comporte trois colonnes: Feature Key (Clé de caractérisation), Location (Emplacement de la caractéristique dans la séquence génétique) et Qualifier (qualificateur associé à une caractéristique de séquence donnée).
L'emplacement de la caractéristique indique dans quel segment de la séquence se trouve la caractéristique. Les formats autorisés pour indiquer l'emplacement de la caractéristique sont définis dans la norme ST.26 de l'OMPI et sont les suivants:
- Numéro de résidu unique : x
- Numéros de résidus définissant une fourchette : x..y
- Résidus se trouvant avant le premier ou après le dernier numéro de résidu défini : <x, >x, <x..y, x.>y, <x..>y
- Site entre deux nucléotides adjacents : x^y
- Numéros de résidu reliés par une liaison intrachaîne : x..y
On peut employer des opérateurs d’emplacement pour former des descriptions d’emplacement complexes :
- “join (location, location, … location)”: Les emplacements sont joints (mis bout à bout) pour former une séquence contiguë.
- “order (location, location, … location)”: Les éléments se trouvent dans l'ordre défini mais rien n'indique que l'on peut raisonnablement les joindre entre eux.
- “complement (location)”: Indique que la caractéristique est située sur le brin de codage complémentaire à la fourchette de séquences définie dans le descripteur d'emplacement lorsque la séquence est lue dans la direction 5' 3', ou dans la direction qui imite la direction 5' 3'.
Pour ajouter une nouvelle caractéristique à la séquence, cliquer sur le bouton “Add feature” (Ajouter une caractéristique) dans la section “Features” (Caractéristiques) de la séquence choisie. À ce stade, il est aussi possible d'ajouter des qualificateurs à la caractéristique; ceux-ci seront traités plus en détail dans la prochaine section.
Caractéristiques CDS
Le type de caractéristique CDS est employé pour décrire la séquence de codage d’une protéine. Une caractéristique CDS peut éventuellement inclure la traduction en acides aminés du segment de la séquence auquel elle appartient. Si cette condition est remplie, l'exigence de longueur minimale apparaîtra comme une séquence distincte dans le projet. Au sein de la caractéristique CDS de la séquence originale, il existe une référence à l’identifiant de la séquence d’acides aminés traduite indiquée dans le qualificateur “protein_id”.
Lorsque l’on crée une caractéristique CDS pour une séquence, il est possible d’ajouter automatiquement le qualificateur “translation” (traduction) (dont la valeur par défaut du “Genetic Code” (Code génétique) est de 1 – “Standard Code” (Code normalisé)) avec une valeur de qualificateur de la traduction du groupe de résidus de la séquence comme indiqué par l’emplacement de la caractéristique. Un identifiant “protein_id” associé et une séquence d’acides aminés distincte peuvent également être générés en cochant la case dans les informations générales (Basic Information) figurant en haut de la page Vue détaillée du projet. Ce qualificateur n'est toutefois pas obligatoire et peut être supprimé après avoir été généré. Vous pouvez aussi créer manuellement des qualificateurs “translation” (traduction) et “protein_id” (identifiant de protéine) qui font référence à l’identifiant de la séquence traduite associée, que vous avez également créé.
Note : À compter de la version 2.1.0, la case “Automatically add a translation qualifier” (Ajouter automatiquement un qualificateur traduction) est cochée par défaut.

Les étapes de création automatique d'un qualificateur de caractéristique CDS sont les suivantes:
- Dans la séquence affichée, cliquez sur le bouton "Add feature" (Ajouter une caractéristique) et choisissez "CDS" comme clé de caractérisation. Si la case “automatically add a translation qualifier” (Ajouter automatiquement un qualificateur de traduction)” dans les informations générales est cochée, l’outil ajoutera automatiquement un qualificateur de traduction, sa valeur et un qualificateur identifiant de protéine et sa séquence d’acides aminés associée (le cas échéant) lorsqu’une caractéristique est ajoutée à une séquence de nucléotides.
- Vous avez également la possibilité de créer un qualificateur de traduction manuellement.
- Lorsque vous avez a fini de modifier la caractéristique et ses qualificateurs connexes, vous devez cliquer sur le bouton “Create Feature” (Créer une caractéristique) pour la sauvegarder. Un exemple de caractéristique CDS est ensuite présenté, associé à la séquence.
Si la valeur du qualificateur de traduction satisfait l'exigence de longueur minimale, le logiciel crée une nouvelle séquence pour le projet avec les attributs suivants :
- Sequence ID Number = valeur disponible suivante d’identifiant de séquence
- Length = longueur de la séquence traduite
- Sequence Name = valeur donnée au champ “Sequence Name” (Nom de la séquence) du qualificateur “translation” (traduction). Si aucun nom n’est indiqué, un nom par défaut (“Seq_#”) est attribué à la séquence.
- Molecule Type (Type de molécule) = “AA”
- Organism Name = le nom de l’organisme est identique à celui qui a été fourni dans la séquence originale
- Qualifier Molecule Type (Type de molécule du qualificateur) = “protein” (protéine)
- Sequence Residues (Résidus de la séquence) = valeurs traduites de la séquence originale
Note : en ce qui concerne la création de la séquence traduite, la séquence traduite distincte n'est créée que si elle comporte au moins quatre résidus spécifiquement définis (par exemple, “AXTG” compte pour trois caractères). Si le qualificateur “translation” (traduction) est modifié et que sa valeur passe en deçà de 4 caractères, la traduction de la séquence associée est alors retirée, de même que le qualificateur “id_protein”.
Recommandations concernant les caractéristiques CDS lors de l’inclusion d’un qualificateur pseudo ou pseudogène :
S’assurer que la fonction traduction automatique (auto-translation) est désactivée lors de l’ajout d’un qualificateur pseudo ou pseudogène à une caractéristique CDS. Si la traduction automatique n’est pas désactivée lors de l’ajout d’un qualificateur pseudo ou pseudogène à une caractéristique CDS, alors, lors de la mise à jour de celle-ci, un qualificateur de traduction sera automatiquement ajouté. Pour corriger cette erreur, désactiver la fonction traduction automatique pour le projet, ouvrir la caractéristique CDS et supprimer les qualificateurs de traduction et “protein_id”, puis procéder à la mise à jour de la caractéristique.
Si vous souhaitez que le qualificateur de traduction soit généré automatiquement, vous pouvez définir la valeur du tableau de traduction et le nom de la séquence depuis le panneau de modification du qualificateur. Lorsque vous créez la caractéristique, le logiciel effectue la traduction puis ajoute un qualificateur “protein_id” à la caractéristique ainsi qu’une nouvelle séquence comportant la valeur de la traduction.
La traduction ne sera effectuée à nouveau que si l'emplacement de la caractéristique ou les valeurs de l'un des qualificateurs "transl_table", "transl_except" ou "codon_start" sont modifiés; dans ce cas, la séquence associée sera mise à jour.
Note : si la valeur de la traduction est modifiée, la séquence associée est mise à jour automatiquement. En revanche, si la séquence de nucléotides associée est modifiée, la valeur du qualificateur de traduction ne change pas. Si le qualificateur “protein_id” est modifié après sa création, la séquence associée perd son association à la séquence originale.
Conseils concernant l’utilisation du codon d’arrêt :
En principe, les codons d’arrêt devraient se trouver uniquement à la fin de la caractéristique CDS, pour indiquer la fin de la séquence d’acides aminés codée. Ils ne devraient jamais se trouver au milieu d’une caractéristique CDS, à moins qu’il n’existe un qualificateur “transl_except” qui indique que le codon d’arrêt doit être traduit dans une séquence d’acides aminés donnée.
Si un codon d’arrêt se trouve au milieu d’une caractéristique CDS (surlignée en jaune ci-dessous) et qu’il n’y a pas de qualificateur “transl_except” indiquant que le codon d’arrêt doit être traduit dans une séquence d’acides aminés donnée, l’outil devrait arrêter la traduction à cet endroit et un bandeau rouge s’afficher pour vous informer qu’aucune traduction ne sera générée.
Une erreur devrait en outre être répertoriée dans le rapport de vérification afin de vous alerter qu’il existe un problème dans le codage de votre séquence.
Saisie les données d'un qualificateur
Les qualificateurs fournissent des informations sur les caractéristiques, pour compléter les informations figurant dans la clé de caractérisation et l’emplacement de caractéristique. Trois formats de valeurs sont autorisés selon les différents types d'informations fournies par les qualificateurs :
- le texte libre
- le vocabulaire contrôlé, les énumérations de valeurs (par exemple, un nombre ou une date) et
- les séquences.
Pour voir les qualificateurs d’une caractéristique, vous devez d’abord choisir celle-ci dans le tableau des caractéristiques de la séquence pertinente, puis cliquer sur l'icône représentant un crayon qui ouvrira une zone grisée.
Les qualificateurs existants peuvent être modifiés en cliquant sur l’icône représentant un crayon à droite de chaque ligne; vous pouvez aussi ajouter un nouveau qualificateur à la caractéristique choisie en cliquant sur le bouton “Add qualifier” (Ajouter un qualificateur).

Lorsque vous modifiez ou ajoutez un qualificateur, vous voyez s'afficher les deux champs ; "Qualifier Name" (Nom du qualificateur, à sélectionner dans une liste déroulante) et "Qualifier Value" (Valeur du qualificateur).
Le champ “Qualifier Value” (Valeur du qualificateur) varie selon le type de qualificateur :
- Qualificateurs à valeurs prédéfinies : le champ de valeur est une liste déroulante dans laquelle vous pouvez choisir l'une des valeurs prédéfinies pour ce qualificateur.
- Qualificateurs de texte libre : le champ de valeur est un champ de texte libre. Outre le nom du qualificateur et la valeur du qualificateur, qui ne peut prendre que la valeur anglais, deux champs supplémentaires apparaissent pour vous permettre de saisir un code langue (p. ex. “ru”) et la valeur de la langue correspondante dans le champ Non English Qualifier Value (Valeur de qualificateur autre que l’anglais). Le champ code langue doit être paramétré avec la même valeur que le “Code langue du texte libre autre que l’anglais” indiqué dans les Informations détaillées du projet. Vous pouvez saisir une série de valeurs autres que l’anglais (Non English) pour chaque langue sélectionnée soit manuellement, soit en important la langue associée appropriée à partir d’un fichier XLIFF.
- Qualificateurs à format prédéfini : le champ de valeur est un champ de texte libre, mais la valeur saisie doit être validée pour garantir sa conformité aux règles spécifiques énoncées dans l’annexe I, section 6 de la norme ST.26 de l’OMPI.
- Qualificateurs sans valeur autorisée : le champ de valeur du qualificateur n'est pas modifiable.
Lorsque vous avez terminé, vous devez cliquer sur le bouton bleu "Create Qualifier" (Créer un qualificateur) pour ajouter le nouveau qualificateur, ou sur "Save" (Sauvegarder) pour enregistrer les changements apportés au qualificateur existant. Pour la dernière étape, après l’ajout ou la modification du ou des qualificateurs, vous devez cliquer sur le bouton “Update feature” (Mettre à jour la caractéristique) en bas de la zone grisée.
2.3 Vue personnes et organisations
Cette vue permet de gérer toutes les personnes et organisations enregistrées localement.
Créer une personne ou organisation
Pour créer une nouvelle personne ou organisation, vous devez commencer par vous placer dans la vue “Persons & Organizations” (Personnes et organisations). Cliquez sur le lien “CREATE NEW PERSON OR ORGANIZATION” (CRÉER UNE NOUVELLE PERSONNE OU ORGANISATION) en haute de la vue, comme indiqué :

Dans la nouvelle vue, vous devez ensuite remplir au moins l’un des champs obligatoires (indiqués par une étoile “*”) correspondant aux détails de la nouvelle personne ou organisation. Pour le déposant/l’inventeur, il s’agit du nom (s’il est indiqué en caractères latins) et de la langue uniquement.
Lorsque le nom de la personne ou de l’organisation n’est pas en caractères latins, il convient d’indiquer ce nom en caractères latins dans le champ “Name Latin” (Nom en caractères latins). Si cette information n’est pas fournie, le projet ne pourra pas être validé au moment de valider ou générer le listage des séquences ST.26.
2.4 Vue organismes personnalisés
Pour créer, modifier, importer, exporter ou supprimer des organismes personnalisés, vous devez commencer par vous placer dans la vue “Organisms” (Organismes).
Créer un organisme personnalisé

Pour créer un nouvel organisme personnalisé, cliquez sur le lien “CREATE NEW ORGANISM” (CRÉER UN NOUVEL ORGANISME) en haut de la vue. Dans l’écran suivant, saisissez le nom du nouvel organisme et cliquez sur “Save” (Sauvegarder). Si une description de cet organisme personnalisé est nécessaire, elle peut être ajoutée à titre facultatif comme le montre la figure suivante. Pour modifier les détails, cliquez sur le nom de l'organisme.
Exporter les organismes personnalisés
Tous les organismes personnalisés et leur description qui sont stockés dans le logiciel peuvent être exportés et enregistrés dans un fichier texte pour être modifiés en dehors du logiciel ou être importés ultérieurement. Pour exporter cette liste, commencez par choisir "EXPORT CUSTOM ORGANISMS" (EXPORTER LES ORGANISMES PERSONNALISES), comme indiqué ci-dessous.
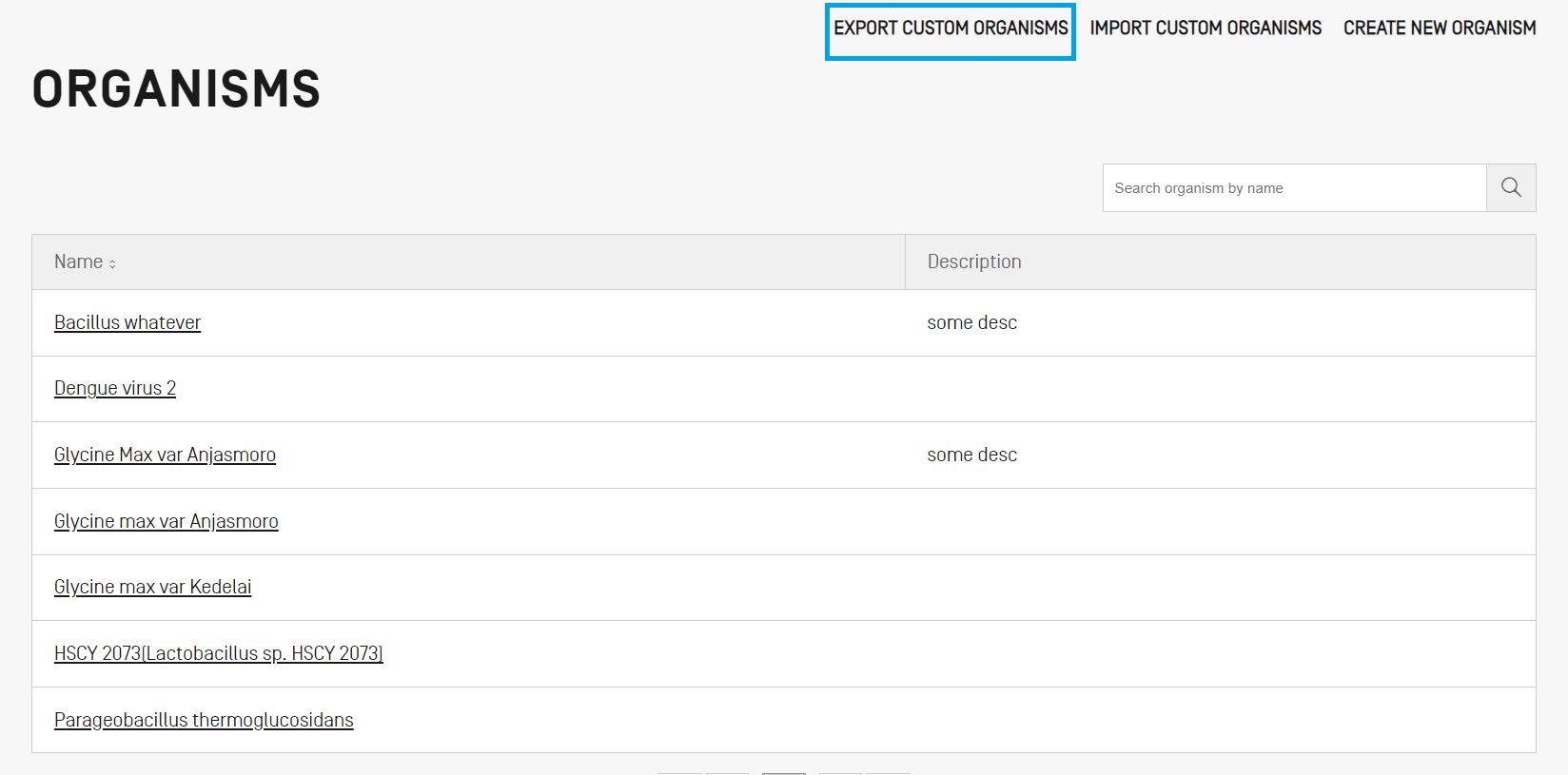
Une boîte de dialogue s’ouvre alors pour vous permettre de choisir le nom du fichier et l’emplacement du fichier souhaité.

Le fichier qui est exporté est un fichier texte qui comprend le nom et la description de l’organisme qui doit être modifié et importé dans le logiciel. Télécharger un exemple.
Importer les organismes personnalisés
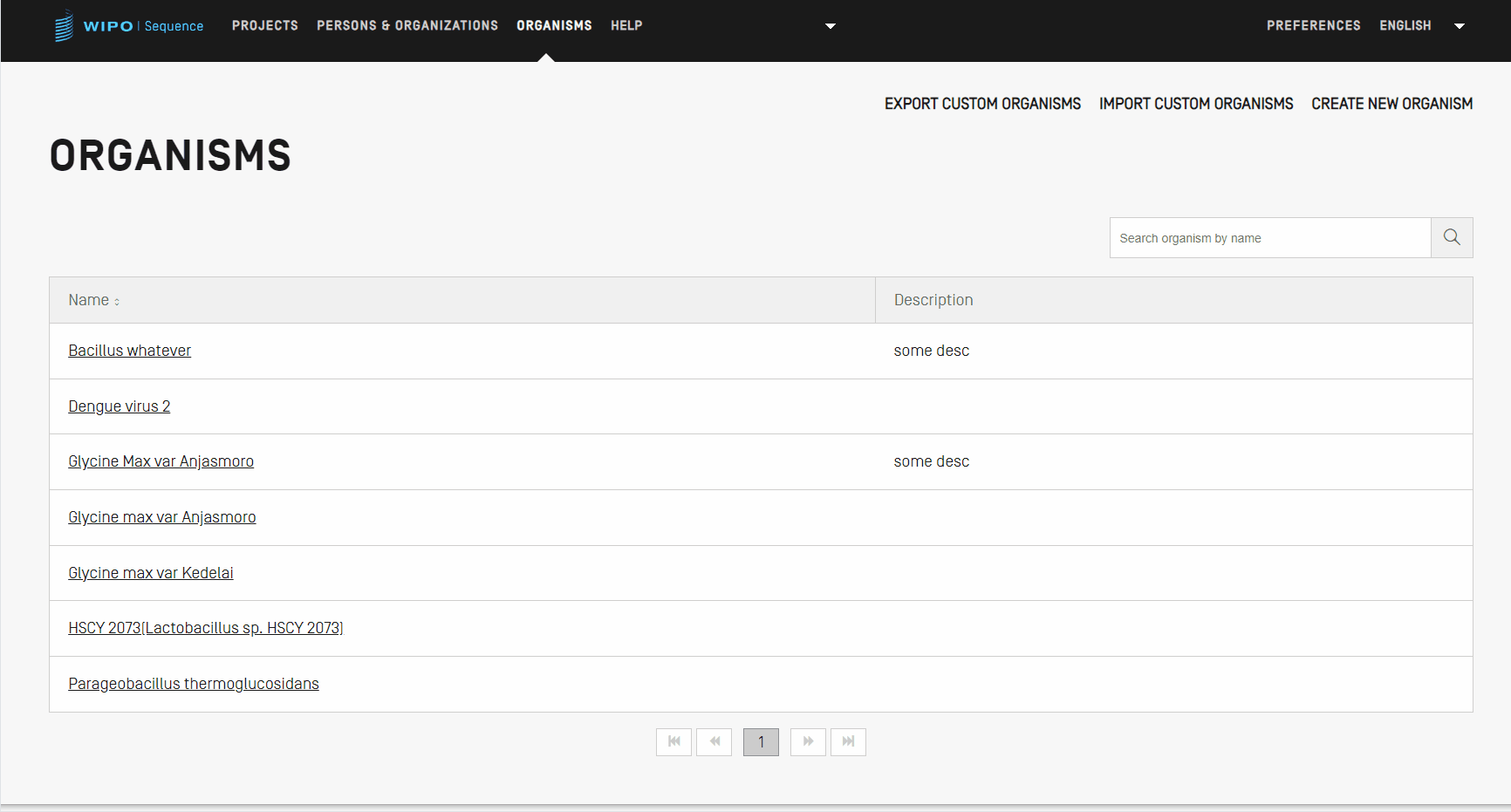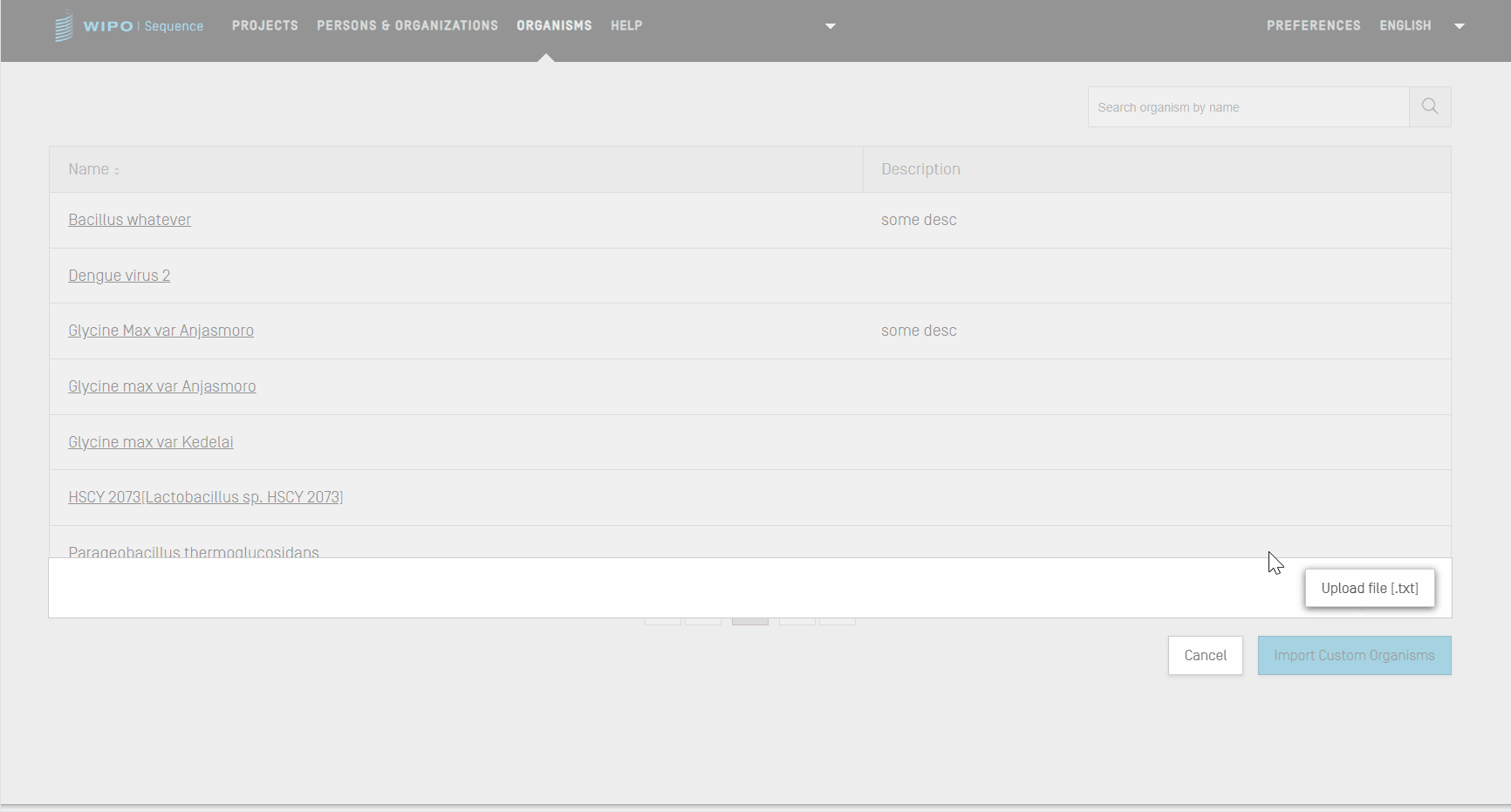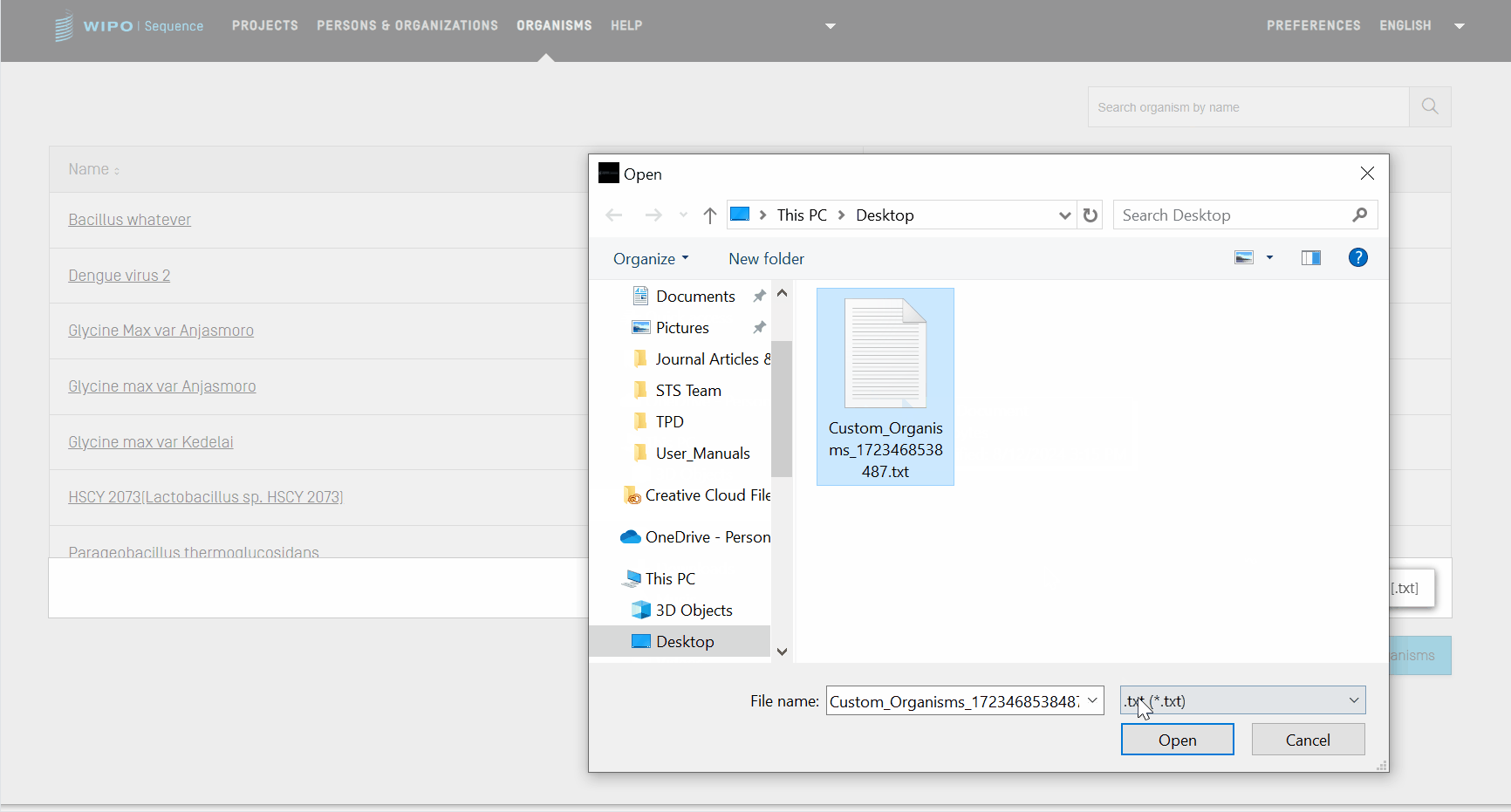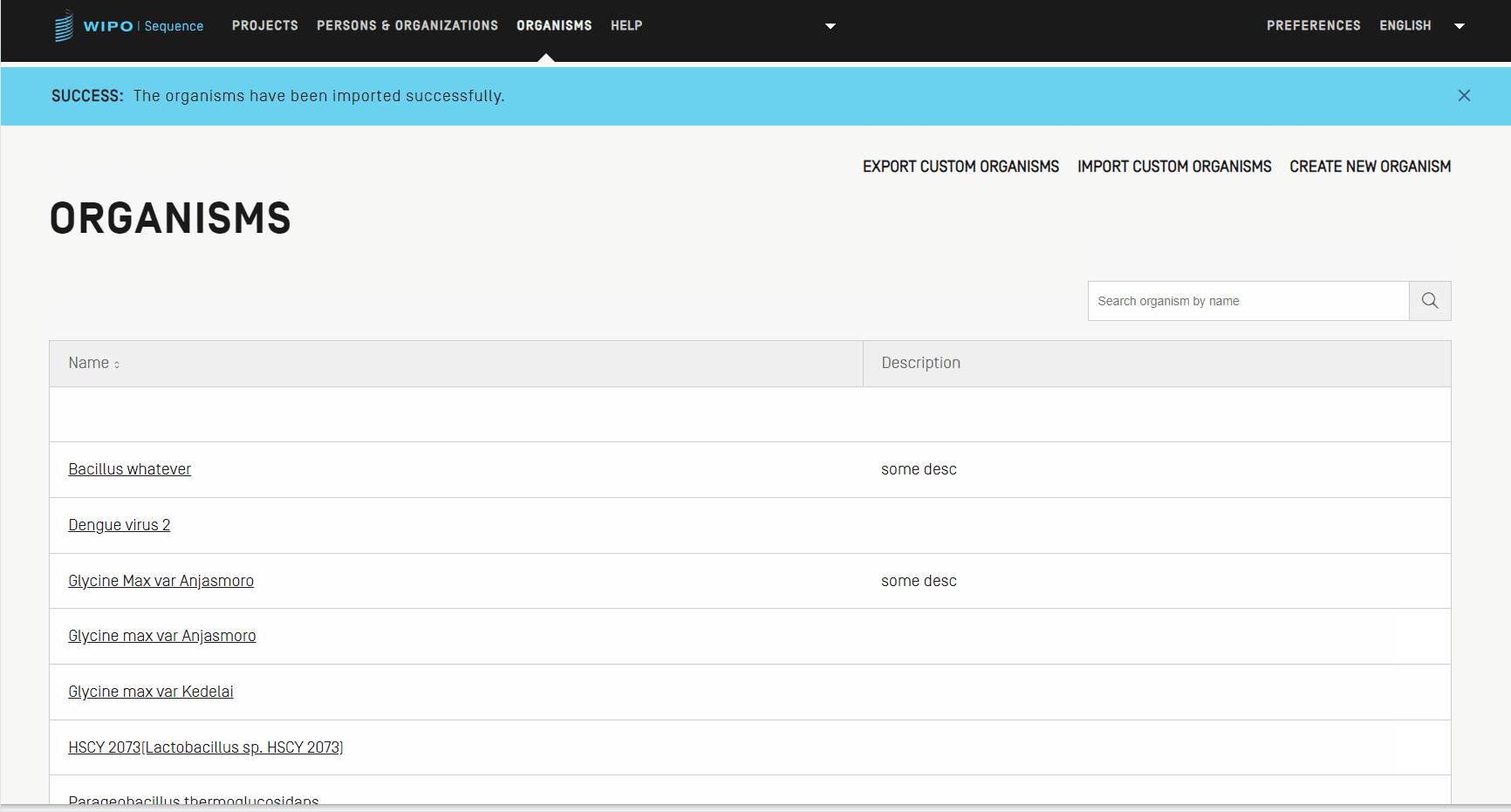
Dans un premier temps, pour importer une liste d’organismes personnalisés, vous devez cliquer sur le lien “IMPORT CUSTOM ORGANISMS” (IMPORTER LES ORGANISMES PERSONNALISÉS) en haut de la vue. Une zone grisée s'ouvre alors en-dessous du tableau des organismes personnalisés.
- Cliquez sur le bouton “Upload file [.txt]” (Télécharger un fichier [.txt]).
- Choisissez le fichier contenant les noms d’organismes personnalisés dans la boîte de dialogue.
- Enfin, cliquez sur le bouton bleu “Import Custom Organisms” (Importer les organismes personnalisés).
Note : le fichier à importer doit être un fichier texte (*.txt) contenant des noms d’organismes personnalisés en texte simple (UTF-8), chaque élément figurant sur une nouvelle ligne.
2.5 Préférences de système
La vue “System Preferences” (Préférences de système) permet de modifier plusieurs paramètres de configuration du logiciel. Ces paramètres s'appliqueront à chaque projet créé ou modifié par le logiciel.
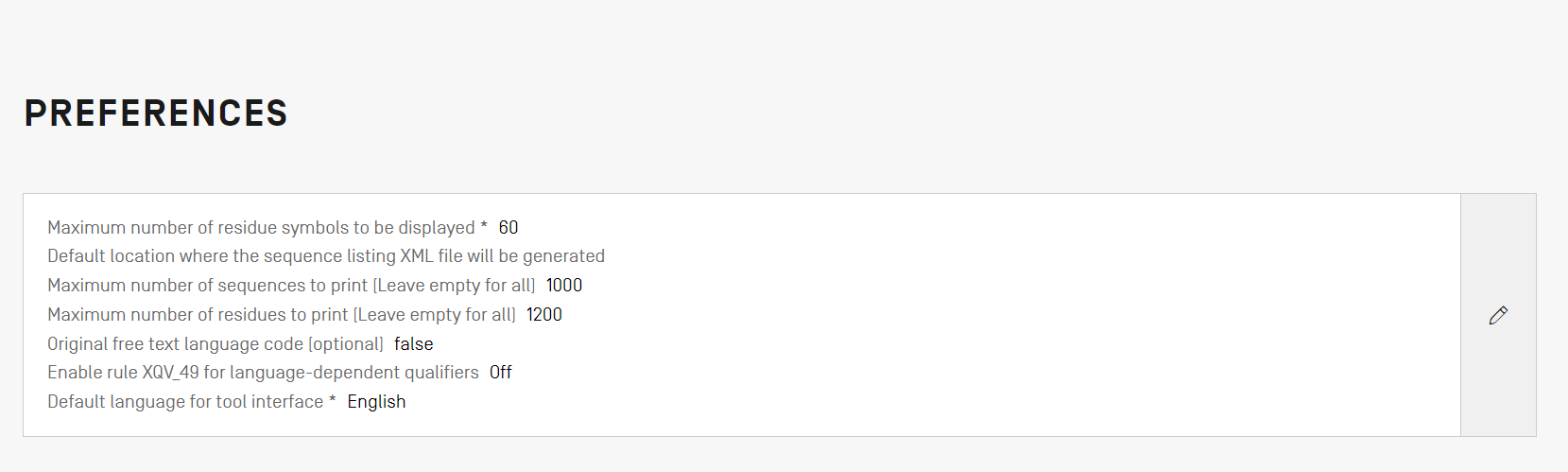
Pour modifier les préférences de système, vous devez cliquer sur l’icône représentant un crayon dans la figure ci-dessus pour ouvrir le panneau de modification.
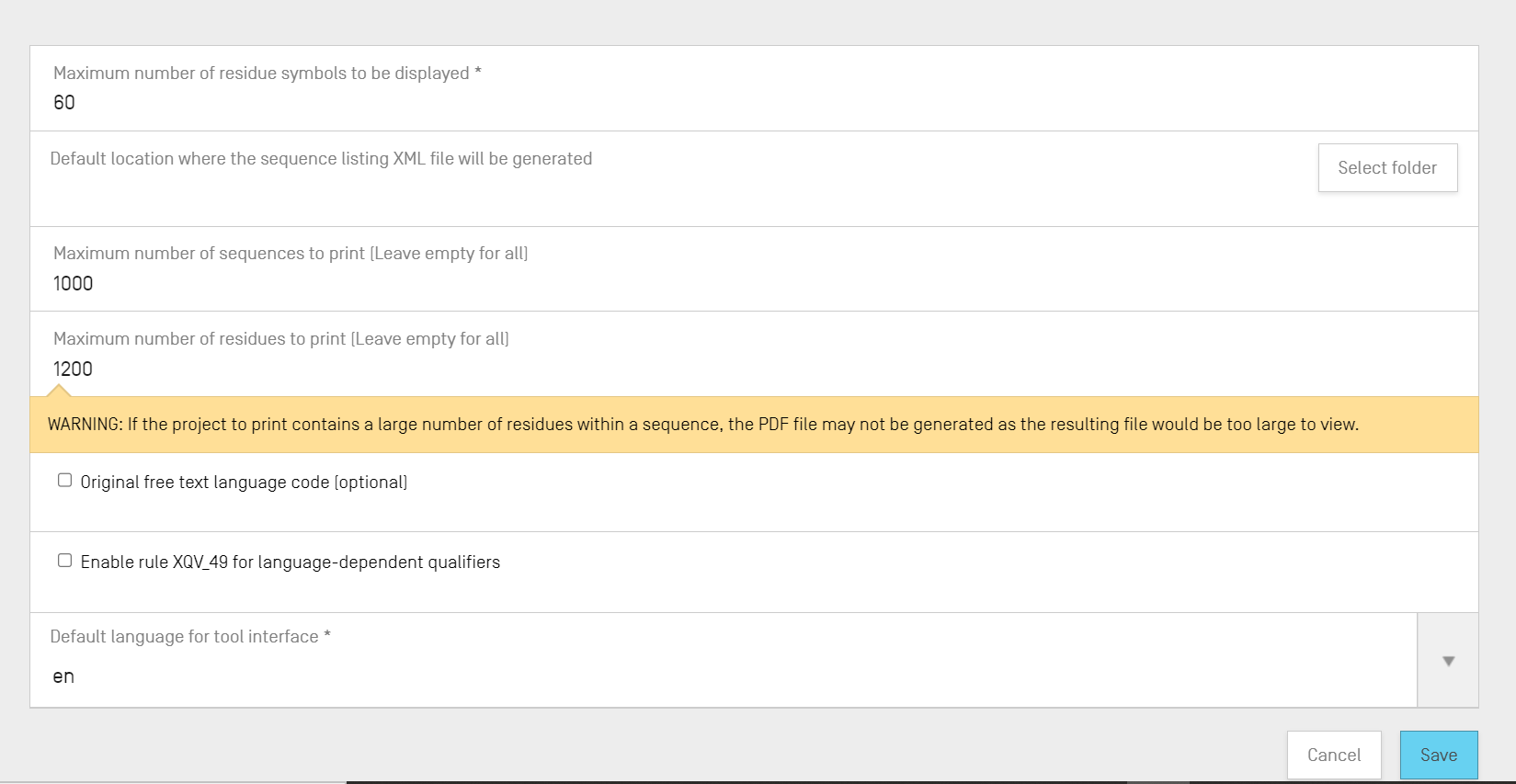
Les paramètres de configuration pouvant être modifiés dans cette vue sont les suivants (dans l’ordre) :
- "Maximum number of residue symbols to be displayed" (Nombre maximal de symboles de résidus à afficher) : ce paramètre définit le nombre de résidus qui apparaîtront dans chaque ligne lorsqu'une séquence sera affichée. Par défaut, il est de 60 résidus.
- “Default location where the ST.26 sequence listing file (.xml) will be generated” (Emplacement par défaut où sera généré le fichier XML de listage des séquences). Il n’est pas nécessaire d’indiquer cet emplacement.
- “Maximum number of sequences to print (leave empty for all): the default is 1000” (Nombre maximal de séquences à imprimer (laisser vide pour la totalité) : le nombre par défaut est de 1000 séquences).
- “Maximum number of residues to print (leave empty for all): the default is 1200” (Nombre maximal de résidus à imprimer (laisser vide pour la totalité) : le nombre par défaut est de 1200 résidus).
- Code langue du texte libre original : si cette case est cochée, alors un avertissement sera émis lors de la validation si le code langue du texte libre n’est pas indiqué. Par défaut, cette case n’est pas cochée.
- Enable XQV_49 : si cette case est cochée, alors un avertissement sera émis s’il n’y a pas de valeur English (anglais) indiquée pour un qualificateur de texte libre dépendant de la langue. Par défaut, cette case n’est pas cochée.
- Default interface language (Langue de l’interface par défaut) : il s’agit de la langue dans laquelle l’interface s’affichera lors du lancement de WIPO Sequence. Par défaut, cette langue est l’anglais.
Note : le troisième et le quatrième éléments sont pertinents lorsqu’on souhaite imprimer le projet dans un fichier PDF. Pour les listages des séquences très volumineux, le fichier PDF obtenu peut contenir plusieurs milliers de pages et être impossible à afficher.
3. Format de fichier
Norme ST.25 de l'OMPI
Le format des fichiers conformes à la norme ST.25 de l’OMPI est décrit en détail dans la norme.
Un exemple figure à l'annexe III de la norme.
Texte brut
Ce format ne permet de décrire qu'une seule séquence. Le code génétique est écrit sous sa forme la plus simple, sans ajout d'information. Lorsqu'ils sont importés, le type de molécule, les caractéristiques et le nom sont ajoutés à la séquence par le logiciel.
Exemple:
aggatatagatagtatatgatagtatgatatgatgatgtatgtatagtgtagttatga
Multiséquence
Le format multiséquence permet de décrire une ou plusieurs séquences, ainsi que leur nom, le type de molécule et le nom de l'organisme. C'est l'un des formats autorisés pour importer des séquences au moyen du logiciel “PatentIn”. La première ligne de texte non vide est l’en-tête, qui comporte les éléments suivants :
<SequenceName; SequenceType; OrganismName>
Les informations de l'en-tête seront interprétées par WIPO Sequence comme suit :
| Entrée de l'en-tête | Entrée autorisée | Interprété comme |
|---|---|---|
| Nom de la séquence | Nom de la séquence (texte libre) | Nom de la séquence dans le fichier de projet de l'OMPI (ne fera pas partie du fichier XML) |
| Type de séquence | Un des éléments suivants :
|
mol_type Notez qu'en fonction de l'entrée Organisme, il sera nécessaire de définir davantage le mol_type pour l'ADN et l'ARN (norme ST.26, paragraphes 75 à 84) |
| Nom de l'organisme | Nom de l'organisme (texte libre) | Note sur la source/l'organisme : si l'entrée est un synthetic construct, le “mol_type” doit être soit “other RNA” soit “otherDNA” (norme ST.26, paragraphe 84.a)). |
Les données relatives aux séquences commencent à la ligne suivant l'en-tête. Toute nouvelle séquence est délimitée par une nouvelle ligne dans le fichier, à la fin des informations sur la séquence de la séquence précédente. Il peut y avoir une ou plusieurs lignes vides entre la fin d'une séquence et le début de l'en-tête suivant. Les séquences d'acides aminés doivent être présentées au moyen de codes à une lettre (Annexe I, Section 3, Tableau 3). Les symboles des nucléotides autorisés sont ceux qui figurent à l'annexe I, section 1, tableau 1. À noter que le symbole “u” dans les séquences d'ARN sera automatiquement converti en symbole “t” comme l'exige la norme ST.26, aux paragraphes 14 et 19, et nécessitera une intervention manuelle après importation. Il est recommandé de convertir le symbole “u” et “t” avant toute importation multiséquence d'un fichier contenant des séquences d'ARN.
L’exemple ci-après présente un ensemble de trois séquences définies au format multiséquence.
Exemple:
<First Sequence; RNA; Albies alba> uuuucuuauuguuucuccuacugcuuaucauaaugauugucguaguggcuuccucaucgucucccccaccgccuaccacaacgacugccgcagcggauuacuaauaguaucaccaacagcauaacaaaaagaaugacgaagaggguugcugauggugucgccgacggcguagcagaaggaguggcggagggg
<Second Sequence; DNA; synthetic construct> attgacgtcagtgacgcggtactgacgtcagctgcagtactgacgtaccaaccacgtggtgagctctcgacatgcaactgactcgtcgctattgacgtcagtgacgcggtactgacgtcagctgcagtactgacgtaccaaccacgtggtgagctctcgacatgcaactgactcgtcgctcagt
<Third Sequence; AA; Mus musculus>
SPPGKPQGPPPQGGNQPQGPPPPPGKPQGPPPQGGNRPQGPPPPGKPQGPPPQGDKSRSPR
FASTA
Ce format contient des résidus et une description. Lors de l’importation, vous avez la possibilité d’enregistrer la description en tant que qualificateur de note.
Exemple:
AJ011880.1 Artificial oligonucleotide sequence SSR primer (CAC13R)
CTCAACAATCTGAAGCATCG
4. Dépannage
Séquence OMPI offre une fonctionnalité "Aide", accessible à partir du menu supérieur.
- Les options d’aide dirigent l’utilisateur vers des informations qui :
- fournissent un lien vers le Manuel de l’utilisateur
- fournissent un lien vers La Base de connaissances de la norme ST.26 de l’OMPI
- fournissent un lien vers le formulaire permettant de contacter l’équipe d’appui à WIPO Sequence
- fournissent un lien vers la version la plus récente de la norme ST.26 de l’OMPI
- fournissent des informations générales sur l’outil bureautique WIPO Sequence
Problèmes courants et solutions de contournement
Le rapport d'importation ou de validation indique que le projet contient plusieurs qualificateurs ID.
Solution de contournement : supprimez la caractéristique comprenant un identifiant ID en double. Recréez la caractéristique au même emplacement mais n'ajoutez pas le qualificateur avant de sauvegarder. Ensuite, modifiez la caractéristique pour ajouter le qualificateur qui convient et actualisez la caractéristique.
Le nom de fichier suggéré pour la distribution Linux est incorrect.
il existe un problème connu en cas d’utilisation de la distribution Linux : une barre oblique inversée “\” supplémentaire apparaît dans le nom de fichier suggéré par défaut. Pour résoudre ce problème, il convient de la supprimer manuellement avant de sauvegarder.
Veuillez vous reporter à la base de connaissances WIPO Sequence pour obtenir d'autres réponses.
Une erreur apparaît lorsque l'on tente d'afficher le listage des séquences au format HTML
Si le listage des séquences généré au format XML est d’une taille supérieure à 100 Mo, au lieu d’afficher le listage des séquences en format HTML, un message d'erreur indique que le listage des séquences est trop volumineux pour être rendu au formant HTML.
Pour toute autre question, contactez-nous.